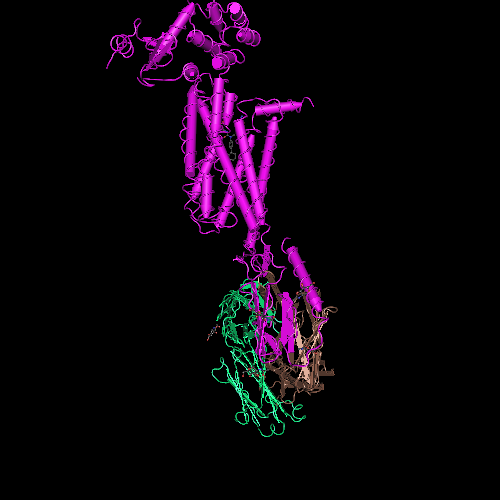*
*
2017.01.15 13:35:14
http://www.myshared.ru/slide/643151/
Цитата: 15 МЕТИОНИН - незаменимая АМК. Необходима для синтеза белков, участвует в реакциях дезаминирования, является источником серы для синтеза цистеина. Метионил-тРНК участвует в инициации трансляции. Метильная группа метионина - мобильный одноуглеродный фрагмент, используемый для синтеза ряда соединений в реакциях переноса этой группы на соответствующий акцептор (реакция транс- метилирования) Метильная группа в молекуле метионина прочно связана с атомом S, поэтому непосредственным донором этого одноуглеродного фрагмента служит активная форма метионина - S-аденозилметионин (SAM)
16 S-аденозилметионин (SAM) - сульфониевая форма метионина, образующаяся при его присоединении к молекуле аденозина (продукта гидролиза АТФ) Реакция активации метионина Присутствует во всех типах клеток Структура (S + -CH 3 ) в SAM - нестабильная группировка, определяющая высокую активность метильной группы (отсюда термин «активный метионин»). Это уникальная реакция, единственная, в результате которой освобождаются все три фосфатных остатка АТФ. Отщепление метильной группы от SAM и перенос ее на соединение -акцептор катализируют метилтрансферазы в реакциях транс- метилирования. SAM в ходе реакции превращается в S-аденозилгомоцистеин (SAГ).
Конец цитаты.
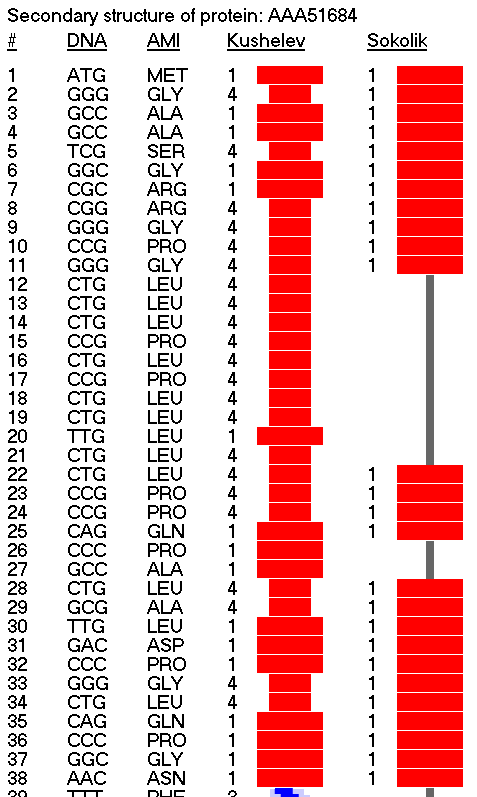
Кушелев: Понятно. Связь Met-Cys косвенно подтверждается, т.к. Cys образуется из Met, а значит, что в мышином лизоциме тоже возможно превращение Met в Cys в процессе дозревания белка.

>NM_207361.5 Homo sapiens FRAS1 related extracellular matrix protein 2 (FREM2), mRNA
GGGATTTCCCTCAAAGCCCAGGGTCCGGGGACCTGGAAAGTAGAAGTGGAGGGATTCAATTCTCCGCGCG
ATTGAGGCGCTAGCGGCGGAGCTGGACGGCCTGGGAAGGCTTCGGCTCCTCGGCTGCGGCTCCAGCCCGG
ACGGCGCCGCGCAACTTTGCCATCCTTCTGGCCCAGCCCCGGCTACAGGAGGACCCCGCGGGCAACGCGC
GGAGTTCCTGGCACTTCCCGGCGGTGTCTCTTGTTGTCTGCCCGGGGACCGACTTCGCATGCTCTCAGGC
TGACCTGTCCAAGCCCGAACACCGGGACCATGCACTCAGCCGGGACTCCCGGGTTATCCTCGCGCCGGAC
AGGCAACTCCACCAGCTTTCAACCAGGACCGCCACCGCCGCCCCGGCTGCTGCTGCTGCTGCTGCTTCTC
CTGTCACTGGTAAGCCGCGTCCCGGCACAGCCCGCTGCCTTCGGCAGGGCGTTGCTGTCCCCTGGTCTCG
CGGGGGCTGCAGGGGTCCCTGCTGAGGAGGCCATAGTGCTGGCGAACCGCGGACTCCGGGTGCCTTTCGG
CCGTGAAGTCTGGCTGGATCCCCTGCATGACCTGGTGTTGCAGGTGCAGCCCGGGGACCGCTGCGCGGTT
TCGGTACTAGACAACGACGCACTGGCCCAGCGACCGGGCCGCCTGAGTCCCAAGCGCTTCCCGTGCGACT
TTGGCCCTGGCGAGGTGCGCTACTCTCACCTGGGCGCGCGCAGCCCGTCTCGGGACCGCGTCCGGCTGCA
GCTGCGCTATGACGCGCCCGGAGGGGCAGTAGTGCTACCACTGGTACTGGAGGTGGAGGTGGTCTTCACC
CAGCTGGAGGTTGTGACTCGGAACTTGCCTCTGGTCGTGGAAGAGCTGCTGGGGACCAGCAATGCCCTGG
ACGCGCGGAGCCTGGAGTTCGCCTTCCAGCCCGAGACAGAGGAGTGCCGCGTGGGCATCCTGTCCGGCTT
GGGCGCGCTGCCTCGCTATGGAGAACTCCTCCACTACCCGCAGGTCCCTGGAGGAGCCAGAGAGGGAGGC
GCCCCGGAGACTCTCCTGATGGACTGCAAAGCTTTCCAGGAACTAGGCGTGCGCTATCGCCACACAGCCG
CCAGTCGCTCACCAAACAGGGACTGGATACCCATGGTGGTGGAGCTGCGTTCACGAGGGGCTCCTGTGGG
CAGCCCTGCTTTGAAACGCGAGCACTTCCAGGTTCTGGTGAGGATCCGAGGAGGGGCCGAGAACACTGCA
CCCAAGCCCAGTTTCGTGGCCATGATGATGATGGAGGTGGACCAGTTTGTACTGACGGCCCTGACCCCAG
ACATGCTGGCAGCCGAGGATGCTGAGTCTCCCTCTGACCTGTTGATCTTCAACCTTACTTCTCCATTCCA
GCCTGGCCAGGGCTACTTGGTGAGCACCGATGATCGCAGCCTGCCCCTTTCCTCCTTCACTCAGAGGGAT
CTGCGGCTCCTGAAGATTGCCTACCAGCCCCCTTCTGAAGACTCTGACCAGGAGCGCCTCTTTGAACTGG
AATTGGAGGTAGTGGATCTAGAAGGAGCAGCTTCAGACCCTTTTGCCTTCATGGTAGTGGTGAAGCCCAT
GAACACAATGGCTCCGGTGGTCACCCGGAATACCGGTCTTATTCTCTATGAGGGTCAGTCTCGGCCCCTC
ACAGGCCCTGCAGGCAGTGGTCCGCAAAACTTGGTCATCAGCGATGAGGATGACCTAGAAGCAGTGCGGC
TAGAGGTGGTGGCTGGGCTCCGGCATGGTCACCTTGTCATTCTGGGTGCTTCCAGTGGCAGCTCTGCTCC
CAAGAGCTTTACAGTGGCTGAGCTGGCAGCCGGCCAGGTGGTCTACCAGCATGATGACAGAGACGGCTCG
CTGAGCGACAACCTGGTGCTTCGCATGGTGGATGGAGGAGGCAGGCACCAGGTACAGTTTCTGTTCCCCA
TCACCTTAGTGCCTGTGGATGACCAGCCACCTGTTCTCAATGCCAACACGGGGCTGACACTGGCAGAGGG
TGAAACAGTGCCCATCCTGCCCCTTTCCCTGAGTGCAACTGACATGGATTCAGATGATTCTCTGCTGCTT
TTTGTGCTGGAGTCACCCTTCTTAACTACGGGGCATCTGCTTCTCCGCCAAACTCACCCTCCCCATGAGA
AGCAGGAACTTCTCAGAGGCCTTTGGAGGAAGGAGGGGGCATTTTATGAGCGAACAGTGACAGAGTGGCA
GCAGCAGGACATAACAGAGGGCAGGCTGTTCTATAGACACTCTGGGCCCCATAGTCCTGGGCCAGTCACA
GACCAGTTCACATTTAGAGTCCAGGATAACCATGACCCTCCTAATCAGTCCGGGCTACAGCGGTTTGTGA
TTCGTATCCATCCTGTGGATCGCCTCCCTCCGGAGCTGGGCAGTGGCTGTCCCCTTCGTATGGTGGTACA
GGAATCCCAGCTCACACCACTGAGGAAGAAGTGGCTGCGCTACACTGACCTGGACACAGATGACCGAGAA
CTACGTTACACAGTGACTCAGCCCCCCACAGACACAGACGAAAATCACCTGCCAGCCCCACTGGGTACCT
TGGTCTTGACTGACAACCCCTCAGTCGTGGTGACCCATTTTACCCAAGCCCAGATCAACCATCATAAAAT
TGCTTACAGACCCCCGGGTCAAGAACTGGGCGTGGCTACTCGAGTGGCCCAGTTCCAGTTCCAGGTGGAA
GACCGAGCTGGGAATGTGGCTCCAGGTACCTTTACCCTTTACTTGCATCCCGTGGACAACCAGCCACCTG
AGATCCTCAACACCGGCTTCACTATTCAGGAGAAGGGTCACCACATCCTGAGTGAGACAGAGTTGCACGT
GAATGATGTAGACACTGATGTTGCCCATATCTCTTTCACTCTCACTCAGGCACCCAAACATGGCCACATG
AGAGTGTCTGGACAGATCCTGCATGTAGGGGGTCTCTTCCACTTGGAGGACATAAAACAGGGCCGAGTTT
CCTATGCCCATAATGGGGACAAGTCCCTGACTGATAGCTGCTCCTTGGAAGTCAGTGACAGACATCATGT
GGTGCCCATCACTCTCAGAGTAAATGTCCGGCCAGTGGATGATGAAGTGCCCATACTGAGCCATCCTACT
GGCACTCTGGAGTCCTATCTAGATGTCTTAGAAAATGGGGCTACTGAAATCACTGCCAATGTTATTAAGG
GGACCAATGAGGAAACTGATGACTTGATGTTGACTTTCCTCTTGGAAGATCCACCTTTGTATGGGGAAAT
CTTGGTCAATGGCATTCCAGCAGAGCAGTTTACTCAAAGGGACATCTTGGAGGGCTCTGTTGTATATACC
CACACCAGTGGTGAGATAGGCCTATTGCCTAAAGCGGATTCTTTTAACCTGAGTCTGTCAGATATGTCTC
AAGAATGGAGAATTGGTGGCAATACTATCCAAGGAGTTACTATATGGGTGACCATCCTGCCTGTTGATAG
CCAGGCCCCAGAAATCTTTGTAGGTGAACAGTTGATAGTAATGGAAGGTGATAAAAGTGTTATAACATCA
GTGCATATAAGTGCTGAAGATGTCGACTCCCTGAATGATGACATCTTGTGCACTATAGTTATTCAGCCTA
CTTCAGGTTATGTTGAAAACATTTCTCCAGCACCAGGCTCTGAGAAATCAAGAGCAGGGATTGCCATAAG
TGCTTTCAACTTGAAAGATCTCAGGCAGGGCCACATAAACTATGTCCAGAGTGTCCATAAAGGGGTGGAA
CCTGTGGAGGACCGATTTGTATTTCGTTGTTCTGATGGCATTAACTTTTCAGAGAGACAGTTCTTCCCCA
TTGTAATCATTCCCACCAATGATGAACAGCCAGAGATGTTTATGAGAGAATTTATGGTGATGGAAGGCAT
GAGTCTGGTAATTGATACACCCATTCTCAATGCTGCTGATGCTGATGTTCCCCTGGATGATTTAACTTTC
ACTATTACCCAATTCCCCACTCATGGTCACATCATGAATCAGCTGATAAATGGCACGGTTTTGGTCGAAA
GCTTCACCTTGGATCAGATCATAGAGAGTTCCAGCATTATTTATGAGCATGATGACTCCGAGACCCAGGA
AGACAGTTTTGTGATTAAACTAACAGATGGGAAGCACTCTGTGGAAAAGACGGTCCTCATTATAGTTATC
CCTGTTGATGATGAGACGCCCAGAATGACTATCAATAATGGACTAGAAATAGAAATTGGGGATACCAAGA
TTATCAACAACAAAATATTAATGGCAACAGATTTAGATTCAGAAGACAAATCTTTGGTTTATATTATTCG
TTATGGGCCAGGACATGGCTTATTACAGAGACGAAAACCTACTGGTGCCTTTGAAAATATCACACTGGGC
ATGAATTTTACCCAGGATGAAGTAGACAGAAACTTAATTCAGTATGTCCATTTGGGGCAAGAGGGCATTC
GGGACCTAATTAAATTTGATGTGACTGATGGAATAAATCCCCTCATAGATCGTTACTTTTATGTGTCCAT
CGGGAGCATTGACATTGTCTTCCCTGATGTGATAAGTAAGGGAGTGTCCTTGAAAGAAGGTGGCAAAGTC
ACTCTTACAACAGACCTACTAAGCACTAGTGACTTGAACAGTCCTGATGAAAACTTGGTTTTTACCATCA
CCAGGGCTCCCATGCGAGGTCACCTGGAATGCACGGATCAGCCTGGTGTGTCCATCACGTCTTTCACTCA
GCTGCAACTGGCTGGAAACAAAATCTACTACATCCACACAGCTGATGATGAAGTGAAAATGGACAGTTTT
GAGTTTCAAGTCACCGATGGACGTAACCCTGTCTTTCGGACATTCCGTATCTCCATTAGCGATGTGGACA
ATAAAAAGCCAGTGGTCACCATCCACAAGCTGGTTGTCAGTGAAAGTGAAAACAAGCTGATTACTCCTTT
TGAGCTCACTGTCGAAGACAGAGATACTCCTGACAAGCTCCTGAAATTCACTATCACCCAGGTGCCTATT
CATGGCCATCTCCTATTCAACAATACCAGACCTGTCATGGTTTTTACCAAGCAAGACTTGAATGAAAACT
TAATCAGCTACAAACATGATGGCACTGAGTCAAGTGAAGATAGCTTCTCCTTCACAGTGACTGATGGCAC
CCATACAGACTTCTATGTTTTTCCTGATACGGTGTTTGAAACAAGGAGACCCCAAGTGATGAAGATCCAG
GTCTTGGCTGTTGACAACAGTGTCCCCCAAATCGCAGTGAATAAGGGGGCCTCTACACTTCGCACTCTAG
CCACTGGCCACTTGGGGTTCATGATCACAAGCAAAATATTGAAAGTGGAGGACAGAGACAGCTTACACAT
TTCTCTTAGATTTATCGTGACAGAGGCCCCTCAACATGGATATCTTCTCAACCTGGACAAAGGCAACCAC
AGCATCACTCAGTTCACACAAGCTGACATTGATGACATGAAAATATGCTATGTCTTAAGAGAAGGGGCTA
ATGCCACAAGTGATATGTTCTATTTTGCAGTTGAAGATGGTGGTGGAAACAAGTTAACGTACCAGAATTT
TCGTCTGAATTGGGCATGGATCTCCTTTGAAAAGGAATATTACCTGGTCAATGAGGACTCCAAATTTCTA
GATGTTGTTCTTAAACGTAGAGGTTACTTGGGAGAAACTTCTTTTATAAGTATTGGCACAAGAGACAGAA
CTGCAGAAAAAGACAAAGACTTCAAGGGCAAAGCACAGAAACAAGTGCAGTTCAACCCAGGCCAGACCAG
GGCCACATGGCGAGTGCGGATCCTGAGTGATGGGGAGCATGAGCAGTCTGAAACCTTTCAGGTGGTACTC
TCAGAGCCCGTGCTGGCTGCCTTGGAATTCCCCACAGTCGCCACTGTTGAGATCGTTGATCCAGGAGATG
AGCCAACTGTGTTTATTCCCCAGTCCAAATACTCCGTTGAAGAAGATGTTGGTGAGCTGTTCATTCCCAT
CAGGAGGAGCGGAGATGTGAGCCAGGAGTTGATGGTGGTCTGTTATACCCAACAAGGAACAGCAACTGGA
ACTGTGCCGACTTCCGTGTTGTCTTACTCTGATTACATATCCAGGCCTGAGGACCACACCAGTGTTGTCC
GCTTTGACAAAGATGAACGGGAGAAACTGTGTCGGATAGTCATAATTGATGACTCTTTGTACGAGGAGGA
GGAAACCTTCCATGTCCTTCTGAGCATGCCCATGGGGGGAAGAATCGGATCAGAGTTCCCAGGGGCTCAA
GTTACAATCGTTCCTGACAAAGATGATGAACCCATCTTTTACTTCGGTGATGTGGAATACTCTGTGGATG
AGAGTGCTGGCTATGTGGAAGTGCAGGTGTGGAGAACGGGCACTGACCTGTCCAAGTCTTCTAGTGTCAC
AGTGAGGTCTCGGAAAACAGATCCTCCCTCTGCAGATGCTGGAACAGACTATGTGGGCATCAGCCGTAAT
TTAGATTTTGCACCTGGAGTCAACATGCAGCCTGTTCGTGTTGTCATTCTGGATGACCTTGGACAACCAG
CGCTGGAGGGAATTGAGAAATTTGAACTGGTGCTTCGCATGCCTATGAACGCAGCCCTTGGCGAGCCCAG
CAAAGCCACAGTGTCCATAAATGACTCTGTCTCCGATTTGCCTAAGATGCAATTCAAAGAACGAATATAT
ACTGGCAGCGAAAGTGATGGGCAGATAGTTACAATGATCCATAGGACTGGGGATGTCCAGTACAGATCTT
CAGTGAGATGCTACACCCGGCAGGGGTCTGCACAGGTGATGATGGACTTTGAAGAACGCCCAAACACTGA
TACCTCCATCATCACATTCCTCCCTGGTGAGACAGAAAAGCCCTGCATTCTTGAGCTGATGGACGATGTG
CTCTATGAGGAGGTAGAGGAGCTCCGCCTGGTACTCGGCACTCCACAAAGCAACTCTCCCTTTGGGGCTG
CAGTTGGTGAACAAAATGAAACTCTCATAAGGATCCGAGATGATGCTGATAAGACTGTTATTAAATTTGG
AGAAACCAAATTTAGTGTCACTGAACCCAAAGAACCTGGAGAGTCGGTGGTTATAAGAATTCCAGTGATT
CGCCAAGGAGACACTTCAAAGGTTTCCATTGTGAGAGTCCACACCAAGGATGGCTCGGCCACCTCTGGAG
AAGACTACCACCCTGTGTCAGAAGAAATTGAGTTTAAGGAAGGGGAAACCCAGCACGTGGTTGAAATCGA
AGTTACCTTTGACGGGGTGAGAGAGATGAGAGAGGCCTTCACTGTTCACCTAAAACCTGATGAAAATATG
ATAGCAGAGATGCAGTTGACGAAAGCCATTGTGTACATAGAAGAAATGAGCAGCATGGCAGATGTCACTT
TTCCTTCTGTCCCTCAAATTGTATCCCTGTTGATGTATGACGACACTTCCAAAGCTAAGGAGAGTGCTGA
ACCCATGTCTGGCTATCCTGTCATCTGTATCACAGCTTGCAACCCCAAATATTCAGACTACGATAAAACA
GGCTCTATCTGTGCAAGTGAGAACATCAATGACACTTTGACGCGGTACCGGTGGCTGATTAGTGCACCTG
CGGGCCCTGACGGTGTGACCAGCCCTATGAGAGAAGTGGACTTCGACACCTTTTTTACGTCATCCAAGAT
GGTCACACTGGACTCCATATACTTTCAGCCTGGCTCCCGGGTACAGTGCGCAGCTCGTGCTGTGAACACC
AATGGGGATGAAGGCCTGGAGCTCATGAGCCCTATTGTAACCATCAGCAGAGAAGAAGGTCTTTGTCAGC
CCCGTGTACCTGGGGTTGTTGGAGCAGAGCCGTTCTCAGCTAAATTGCGCTACACAGGCCCTGAGGATGC
AGACTACACAAACCTTATCAAGCTCACTGTCACAATGCCACACATAGATGGCATGCTCCCCGTGATCTCC
ACTAGAGAGCTTTCCAACTTTGAGCTCACCCTCAGCCCTGATGGCACAAGAGTTGGAAACCACAAGTGCT
CCAACCTCCTGGATTATACTGAAGTGAAGACTCATTATGGTTTCTTGACTGATGCTACCAAAAATCCAGA
AATAATTGGAGAGACATATCCTTACCAGTACAGCTTGTCCATCAGAGGTTCCACTACCTTGCGCTTCTAC
CGGAACCTGAACCTAGAGGCCTGTTTATGGGAGTTCGTTAGCTACTATGACATGTCAGAACTCCTTGCTG
ACTGTGGTGGCACCATTGGAACAGATGGACAGGTCCTAAACCTAGTGCAGTCCTATGTGACCCTTCGAGT
CCCTCTGTATGTTTCCTACGTGTTCCATTCCCCCGTGGGGGTAGGAGGCTGGCAGCATTTTGACTTGAAG
TCAGAGCTTCGTCTAACTTTTGTGTACGACACCGCCATCTTGTGGAATGATGGAATTGGCAGCCCCCCAG
AGGCTGAACTTCAAGGTTCTCTCTATCCAACCAGCATGCGCATCGGTGATGAGGGGCGCTTGGCCGTGCA
CTTCAAGACAGAGGCTCAGTTCCATGGCTTATTTGTGCTGTCACATCCCGCATCCTTTACAAGCTCAGTG
ATCATGTCAGCTGATCATCCAGGCCTGACATTTTCCCTCCGCCTCATAAGGAGTGAACCAACCTATAACC
AGCCAGTACAGCAGTGGAGCTTTGTCTCTGACTTTGCCGTACGTGACTACTCAGGGACCTATACTGTGAA
GCTGGTGCCATGCACTGCCCCATCACATCAGGAATACCGCCTGCCAGTCACCTGCAACCCCAGAGAACCT
GTCACCTTTGACCTTGACATCCGATTCCAACAGGTCAGTGATCCAGTGGCTGCTGAGTTTAGCTTGAACA
CCCAAATGTACCTGCTCTCTAAGAAGAGTCTCTGGTTGTCTGATGGATCCATGGGATTCGGGCAAGAGAG
TGATGTTGCTTTTGCAGAAGGTGATATAATTTATGGTCGTGTCATGGTGGATCCTGTCCAGAATCTGGGT
GACTCCTTTTACTGCAGCATTGAGAAGGTGTTTCTATGCACTGGAGCTGATGGCTATGTTCCCAAGTATA
GTCCAATGAATGCAGAATATGGCTGCTTAGCCGACTCTCCTTCACTCTTATATAGATTTAAAATTGTGGA
CAAAGCTCAGCCAGAGACACAAGCGACCAGTTTTGGAAATGTCCTATTTAATGCCAAACTAGCAGTGGAT
GACCCTGAAGCCATTCTCTTAGTGAATCAGCCTGGATCTGATGGATTTAAAGTCGACTCAACACCACTCT
TTCAGGTCGCTCTAGGCCGAGAATGGTATATACATACGATCTATACAGTGAGATCGAAAGACAATGCCAA
TCGAGGTATTGGCAAAAGAAGTGTGGAGTACCATTCTCTGGTGAGTCAAGGAAAGCCCCAATCCACCACC
AAGAGCCGGAAGAAGAGAGAGATCAGGAGCACACCCTCACTGGCATGGGAGATTGGTGCTGAAAACAGTC
GAGGAACAAACATCCAGCACATTGCCCTGGACCGCACCAAGAGGCAGATCCCCCATGGGAGAGCACCTCC
AGATGGCATCCTCCCCTGGGAGCTCAACAGCCCCAGCTCTGCAGTCAGCCTGGTCACTGTGGTGGGAGGC
ACCACGGTAGGGTTACTCACCATCTGCCTCACTGTCATTGCAGTGCTGATGTGCAGGGGCAAGGAAAGTT
TCAGGGGGAAGGATGCCCCGAAAGGCTCCAGCAGCAGTGAGCCCATGGTGCCCCCACAGAGCCATCACAA
TGACAGCTCAGAAGTTTGATGACTGCAGGTAGAATTCAACCTTTTCCGTAAGTGCCTCGGAAAAGATCAC
AATGGAACCTTAAATACTTCTGGTAAACCATAGAGAATGGAGGATGGCTGTGATGAAGCTGCTTAGAGAA
TATGACTGTACTAATGGAGTTTGATTTGAATTCTCCATACTCTTTTTTGCATCAAAGGACAAATTAAGGC
ATCTTTCCTCTTGCCCCAAAGGCAGCAACAACGTACTCATATGTACACAGAGCCATGATGTGAGGAATGT
ACTCCTCATTTTAGACATATTCTCTATGCAGTGGAGATAAATCTATTAAAAGGTGCTAACAGACTCTCTT
ACAAGTGTAAGAGGAATCTACTGTTGCTATGTTAGTGTGAATGTTGAAGGTGCAATTATCACATTGTTTA
TTAATTGTATAACAGATTATTACTAGAAAGGTTTTTGTTGCTAGTCTGGAAAACTGGTGAACACACTGTT
TATTATGACAAGTTTTCAAATAGGTGAAGACAGCATAGAATTATGACCAGGGTGGCTCAACCCACAAATC
AACTGATCTACTACTTGGGAGACAGTGGAAGGAAAGATAGAGGGAAGGAAGTTCACTCACTTGAATTTGA
ATTGCTTCTCATTCTCATCAGACTCTCCTTGTTTTGTTTGCATATTTAATTTTAAATTAAACCAAAGAAT
ATGTTAATTCTGAAAGAGATTTTTAAGAGATGCTGCTCTGTTTTGGCATATAGTGAGTAATGGTAGAGCT
CTTGCATGGTAATATACCTTATTGGTGCTCAACATTTGTGGGAAATTAGAGGGTTGGTGAGATTTTGGTG
ATGAATAAATGCCATAGAGTTAGAGTCACTACAAGACAATGTTCTCTAAGAACTGAGCCTAGATGTTTGC
AGTATTGAACCCATAAATGATAATAAGAAACATTGTTACAGATGGTGAAGGGAGAAAGTGGTATTTATTA
ATATAATGTGTATAATCAGAGTGCCTCTTATACATACTTTATAGAATAAAGGAACATTTTGACAGATGAG
GTTATCCCTCGGAGTAGCGTAATCACACAATAACATTTAGAACTTAAATTGGCTACAGGACATTTTATTA
GCTTTCTATCCAGCATCTGGTCCAAACGATGGGACTCTCTGCAACTATCATTCCAGAAATGGTCTTGGAG
GTGGCCAGAATCATTGACCATATTTTTATTTGGTTCAAGAAGACATCGCTCTGGCATCCCCAGTTCAGTG
AACTGGTACAATGTGGTCCCTCTCAATATTTAAAAATATAATTTGTAACAGTGCTTACCATTCACCTCCC
AAAGCCAAGGAACTTGAGTGAGCTTCCTTGGCAAGCCTCCCATTGCCTTTCTGCATCAATGTGTTTGTTA
CCAAATAGCAGAAATTTCGCTTAGAAGGGGAAAACTTCAGCTTTCCAAAAGCTAGTAAACATGTTTCAAA
GAAAAATAAGGTCAGGAGAAAAAGAATAATGCCTACAGATTATCCTTCAGATGCTGGCTAGAATCCCCTT
TGACTTCTAGCCATAAGCTGGCCACATTGGACATCTTGGTCACTGCAGAATTTTAAAAGGCAAACTTCTC
AGAACAGATGTGCACATCTTAATTTGGTGTCTGTAAGTATCAACCATTTCTTGATCAAGATAAGGAAGCA
ACACTGATACTGAGAGAGAGGTCATATGTCCAATAGTTGCCTTTCTTCTCTTAGGCAGCTCTGCACTTCA
TGTTCAGTTTTTTAAAAATAGACTGTAGTATCCTTTCATTGGAGAATTTACAAAAATATCATCTACAGCT
CAAAAGTCCCACTAACGGCTTATATGAACAGAAAGTACTGTGTAGCCCAGGAGTTTATTTAGCAAGATAA
ATAGTGGTAATTTCTTAATCATTCCCCTTTCCTTTGCTTTACCTTCTGGCAGATGCTTAGCTACTTCTTT
GGCTACCTCATAATTTGTATGGGAATGAACATGAATTAGGGAACTTCAAATAGTGCGTAGACTTTTCCCT
ACTTACCATTGGGTTCAATTGATGTAGACTGTTAGAGCAAGTATACTCAATCAACTTGCTGATATTTTAG
TAGTTAACAGCTTCCGAGCCTATCATGCAATTTGGAGTCTGTCAATGAATATAGCAATCGGTGAAGCATT
ACTGTACGAAGTCTGTTTTCAGGCTTTGGGTCAAGCTGGTTTTATACCAAATTTCACTCTACAATGCATA
TTCTAATGAAGCTACTTTATATAAATTGGAGAGCTTATAAAATGCAGAACATTTCTATCCTATCCTACCA
AATATTTATCCTTCTCTGTCCTCTATTCTCTTACTCTCCTCTCTCCCTTTTTCCTTCCCTTTATTCCCTT
GAAAAAAGAGAATCTGTGGGAAATAGGCAGATATAGCTTTTTAAAATATAAAGTACTTGGGATTTTGCAT
TATTTTTCTTAATATCTATTTACTAAGTATGTAATTTAATATACATTAATTATTTTTTGAAACTCTTGTT
AGTGGGAAGAATATGGTAAATTTTTTGTTAAATAAAATAGACCCTTATGTTTAGCATTTTGTTTTTAGAG
AACTATTCTGGTACTATCAGAACAAATACATAAAATAACTTCCCATAGAGAACAGGATATAGCAATAATA
GCTCCTTAGATACTCAGTGGCTTCTGACTCCAATCAAGGTCTTGTTGATATTATATAGTAAAAATAAAAC
CAAAAATAAATATTATTCAAGTGGCTCTTCTAAGCATGTGAATCATGAAGCACTGAAATATGTATTTTAA
TGATGATCTTATTTATTCCCATTTTTGCCCTTAGTTAACATTTACTGGTGCTCACCTAGGATTGGCTATT
CTGAGGGATTGCATAGAAACCAAGCTCCACTTGCTGTCCTTGGGAAGGTTATAACTGAATGCAGCTCTTT
ATTTGGACTAAAGTGTCAGGATATGCATTAGATTCTCTCCTGAACCAAAAACACAACAGTCATTATCTGT
GAACCATAATTTAAAAATCTTTCTAGAATAACAACAGCAGACTCCACTCTTGTTTGTCTAAAAGAGCCCT
ACTGGGTATGGATCATTCTGATGACAGATTTATACAAAATGATTCAAACCAGTAACTTAGTAAAATTGAC
CTTCGCAAAACCTCACTGGGGGAGTGCCTTGTAGAGCTGTGGGTGGGACTGCACATTCTTTTCCTCTTAG
TAAAAGATAGGCCCACTTTATTCCAAGAATAACACTTAGCACATAAACTCTTCTTCCAGCTCGTTAGCAG
CATTAGCACCTTCTGAATTCCACCCTCTCAGAAGAATCCACAGTGTTTGAACAATTTGCATAAAGGTCAG
CTAGCATCCTGCTGCCAAGCCACTGCATAGCATTTGTGATAAGAAGGACCAACTCTAGGCTCAATATGAA
GGGATTTAGTTCTGTAAGCAGCAAAAAAGCTTCTTTATCAAGTCATCTTACCTCTAATTCTTTTCCAGTG
TGCCAACTCCAAAGTCAACATTAAAAATGTAAATGGACCTGTGTAAATATCACAGAGAGCTTTTCCTTAT
ACATCTCAATGCTGAGAGTTAAAATATTCCCAGGTTAAAATTTTTTTAAAGTACCAATAATAGAGCTAAA
TACAATGACATTTGCTTTTAAAAGGTGGATATTTTATTTCTGCTTTTTGAAAATACTTATTTAGTATTGA
CTTGGAAGCCAATTTGGTCCTTTAATAAGTAAAGAAAATAATATGTTTAAAAATGTAAATGTTTTACAAA
TTTGAAACTTTCATAATTGTATTAATCAGAAAACAAGCACATTGCCATTCTTTGAAACTCATGTTTCTAG
ACATGACAGCAGTAATAAAAGGATGAAAACAAGTGTCTTCACTAAGCGTATGGCCAATAAATGGGACCCA
AACGTTCAATCTGTTCAGTTTACCAAGGTTCAGAAATACGTAATTTAGCAGGAAACTATAAATACCAGTG
CTATCACAGCCACACATACACACACACAGACATAAAATAACCAAACATCTCATTTCTAGGAAAGAGATAA
CACTAAAGGCATCATAGGTTTAACTGAAATACGTTATATGAAGTTTTACAAAAAGGTCAACAGAAAGCTC
ATTTGTGAAAACATACTCTCATGGGAGCTTCTTTAACATTAGTTCAGAGGTTAATATATTTCCTGGAGGT
GTTTTCCTAGAATTGATTGCACTATTGCATGGTAATAACATTTAATTGTTAAGGAAACATTATATATAGG
TTCAAATTATCCCTTAATGTTGATTTCTCCCCTTTTCCATGGATTTTGATACTAAGAAACAAAATGCTTT
GAGATTTTGGTAACTATTTTGATTTTGATAAAACATGTTAAAATAGAAGGACATGATATTTTTCTATAGT
TTCCATCAGGAAGAGTACATCAGAAACTTCTCCATAAGGAAAGAAAACTGACTCTCTCTTGAACTAGTGT
TGACAAAATACACTAATGTCTTTCTTAATTTTATTTTATTAGGAGAAAAATCAGAATATTAAATTTGCAA
ACTTTTGAACAGCAGTAATAGCTCCTTAGACACTCAGTATCTTTCTTCCCTATCTTTCAAGCACATTTAA
TTTCTTTCCCCTGGTATTTGTTTGCTTTGTGTTTTTTCAATATTTTGTTTGCTAAAGAACAATAACAACA
ACAAAGCTGGTCCAGCCCTGACACCTACTTTTGCTTTTTTTCTTTCTTTCTTTTAATTTGTTGAACTATA
GGTTTCTTTCTGAGATGTATTTTTCATTCTTCCAATAATGCTCTCTTTTTCATTTCAAAACCATTACCAC
TTTCAATATGTAATTAACATCCTCATTCATCAGAAAGCTGGTAAGTCCACACAGCATTTGTGCAGTATCC
CTTCTGTGTAAAATGTTAAGGCTTTCAGATGAAAGAAAAAAGAACTTCCTGCAAAAGATCAAAAGCATCA
TACTAAATAAAATGATGACAATATGTGCCTTGATTTTTCTCTCAATGGAGGTTCAACATTGACATTTAAG
ATGTAAAAAAAATTACTTTGATGGTTTAATTAAGAATAATAAGTCAAATATCACATGTATGTAGTTCTTT
TGGATGACCGAATACCTTAAAATGAACTAAATAACTGAAATAAAGTGTTAAAATGTTAACAGCGTTGAAG
TTTTTAAAATTATAATTATCAAATGCACAATGTTCTTTTACTATGCCAAATGTTGCCTACACTGATTACA
TGATTGTCACAGTTTAAGTGAATTTCTACTATGGATCTTTATTTTCCCCAATGTTTGGATCATCCTCTCA
GATAAGAATTGTCAGTATTGCACCTCAAGAAAGAAGAAAAAATAAGAATGTAAATTTTTAAAAGCTAGCA
TTAAAAGTAAAGAGCAGTACAATAAAAATCACTTAAAATAGAGAAAGGCAACTACTGAGTAAGAAGAAAA
GATAATTTCTGTAAAAGCTAAATTTATGCACATTACAAAAAGTAGTCAAGTCAGTCTTCCATTGTGACTT
CAGGAATTCAAATGCAAGTTGTTGGCAAAATATATATATACACACACACACACATACACACACATATATG
TACATACATATATATATAGAGAGAGAGAGATAGATTTTTTTTTTTTGAGACAGAGTTTCGCTCTTGTTGC
CCAGGCTGGAGTGCAATGGAATGATCTTGGCTCACCGCAACCTCCGCCTCCAGGGTTCAAGTGATTCTCC
TGCCTCAACCTCCTGAGTAGCTGGAATTACAGGTGCCCACTACCATGCCTGGCTAGTTTTTGTATTTTTA
GTAGAGACAGGGTTTCTCCATGTTGGTCAGGCTGATCTCGAACTCCCAACCTCAGATGATCCTCCCGCCT
CCACCTCCCAAAGTACTGGAATTACAATGGCGTGAGCCACCACAAGATATTTTTAAAGCCCATTTGTCTA
TAAAAGAAAACTCTTGGACAAGGAGCTTTGTGTTTTATAATTGTATTACGTGCCTGCTTCCTTACCAAGT
GCCCTCCATAACACTAAGTAAATTTATATTTATCTAGCCTCTGAGTGACATATCTGTGTGTAATGGAATT
ATTTGCAGAATTTAATAGCCTTTTTTATTTTCGCATGAGAATTTTTCAAAAACTATCAGGGTCTAGTTTT
ATCATACATTTACTAAGTTACTACATATTGGTGGATATTGACGTTTATTCCTGGGAATCTAATTTTGCAA
ACAATTTGGAGCCATTCAGTCATAAATTCATGTCAACAATAAGTCTTGCTGCCGCTTGTGTTTCATAACT
ACGCTTGCTTTCCTTCAAAATATACCAAGTGTGTAATATAAATAAAGCCCATATCAATATAAAAAAAAAA
AAAAAAAAA
Композиционный код 6:
5,3,5,5,3,5,5,3,4,4,1,1,3,2,3,5,5,2,5,3,3,5,5,3,5,5,3,5,5,2,3,2,5,3,5,5,3,5,3,3,4,4,4,1,1,1,2,5,5,5,
2,3,5,2,1,1,1,1,1,1,1,1,1,1,1,4,4,2,5,5,2,3,3,5,3,4,4,4,3,5,5,5,3,5,5,2,5,3,5,3,3,1,1,4,1,3,2,1,4,1,
1,4,1,6,1,2,5,5,3,5,5,3,1,4,1,4,2,1,1,1,1,1,3,3,2,2,5,2,4,4,1,4,4,4,4,4,4,4,3,5,5,2,5,2,1,1,1,4,2,5,
5,3,1,1,1,1,4,1,4,1,3,3,4,4,4,3,2,5,5,3,3,5,5,5,2,4,4,4,1,1,2,4,4,4,3,5,5,3,3,4,4,4,3,5,5,3,1,4,4,1,
1,4,1,1,4,1,1,1,4,3,5,2,2,5,5,5,2,5,5,5,2,4,1,1,4,3,1,1,1,1,4,1,1,3,5,3,1,1,4,1,1,3,1,4,1,4,1,1,4,3,
4,1,1,1,4,4,1,4,1,3,5,5,5,2,5,2,2,5,2,2,5,2,4,1,4,1,4,1,1,1,1,4,1,3,5,3,5,5,5,3,4,4,4,3,1,4,4,4,1,1,
3,1,4,1,4,4,1,4,1,1,1,1,1,1,1,2,1,1,1,1,4,1,1,4,1,1,1,1,4,4,3,5,3,2,3,1,1,1,1,4,1,1,3,2,2,5,3,5,2,1,
1,4,1,3,1,4,6,1,1,3,3,5,5,3,2,5,5,5,3,5,5,2,5,5,3,5,2,2,1,1,1,1,2,1,6,4,4,1,4,3,2,2,5,3,3,5,5,5,3,3,
5,3,1,1,1,1,1,3,4,4,1,1,2,2,4,1,1,1,3,2,5,5,5,3,1,4,1,6,6,6,6,1,4,1,1,3,2,4,4,1,4,1,2,4,4,4,2,5,5,3,
3,5,3,5,3,1,4,1,1,1,1,3,3,3,2,5,5,3,1,1,1,1,1,4,1,1,3,3,1,1,4,1,3,5,5,5,3,5,5,3,4,4,1,4,1,3,1,1,1,1,
3,3,3,5,3,1,1,1,1,1,3,3,5,3,5,5,2,5,3,2,3,2,2,3,2,5,3,3,5,5,5,2,4,4,1,1,6,1,2,5,3,4,4,1,1,4,3,5,3,3,
3,5,3,5,3,5,3,5,5,5,2,5,3,2,5,3,3,5,3,1,1,1,1,1,3,5,3,5,2,3,2,5,5,2,4,4,4,3,4,4,4,3,3,5,3,5,3,5,3,3,
5,3,5,5,3,3,5,5,5,3,2,5,3,5,5,2,1,1,1,4,1,1,1,3,3,5,3,1,1,4,4,1,1,1,4,4,3,4,4,4,3,2,2,1,1,1,1,2,5,3,
4,1,1,1,1,3,5,3,5,3,5,5,2,3,3,5,3,1,1,4,4,4,2,5,2,5,3,3,2,4,1,1,4,1,3,5,5,3,2,3,5,5,3,2,3,3,3,5,5,3,
3,5,5,5,2,5,5,3,3,5,5,3,5,3,5,5,3,3,5,3,5,3,5,5,5,3,3,5,3,5,3,1,1,1,1,4,2,3,3,5,2,2,5,2,1,1,1,1,1,1,
2,2,1,1,1,4,1,3,3,5,3,5,5,3,3,3,5,2,5,2,5,5,5,2,3,3,5,5,3,5,3,5,3,3,3,4,4,4,2,5,5,3,5,3,3,5,3,3,5,3,
5,5,3,4,1,4,1,3,5,3,5,3,3,5,5,2,5,3,5,5,5,2,2,4,1,1,1,1,4,1,1,3,5,5,5,2,3,5,2,3,2,3,5,2,5,3,5,5,5,2,
5,2,5,3,3,5,5,2,5,2,5,3,1,1,1,1,3,5,5,5,2,1,4,4,1,3,3,5,3,1,1,1,1,3,3,3,3,3,5,3,5,5,3,3,3,4,4,4,3,3,
2,4,1,1,1,1,1,1,4,3,5,2,3,5,3,5,3,2,3,5,3,5,3,5,5,3,1,4,1,1,1,2,3,1,1,1,1,1,1,1,3,3,5,5,5,3,1,1,1,4,
3,5,2,1,1,1,4,3,3,2,5,3,3,3,5,3,5,3,5,3,5,3,5,2,5,3,3,5,5,5,3,5,3,2,4,4,4,3,2,5,3,1,1,1,1,1,1,2,3,5,
5,2,3,5,3,5,3,3,4,1,1,1,4,3,3,1,1,1,1,3,5,3,5,3,3,3,4,4,1,1,3,5,3,2,3,5,5,2,5,3,3,3,5,5,2,5,5,3,3,3,
5,3,5,5,5,3,2,3,5,3,3,3,5,3,3,3,5,3,5,3,3,3,5,5,5,3,5,3,3,3,1,1,6,1,3,5,5,5,3,3,2,3,5,3,5,3,5,5,5,3,
5,3,2,2,5,5,3,3,3,1,1,1,1,1,1,3,3,2,3,5,5,5,3,3,5,2,5,2,5,3,3,5,3,3,3,5,5,3,5,2,3,5,3,3,3,5,3,3,3,5,
3,3,5,3,2,3,3,2,1,4,1,1,4,3,3,3,5,5,5,2,3,5,3,2,3,3,5,5,2,2,5,3,3,3,3,3,3,2,2,2,5,3,2,3,3,3,3,1,1,1,
4,3,3,1,1,1,1,3,2,3,3,5,3,3,2,3,3,3,3,5,3,3,2,2,2,5,3,5,3,2,3,2,5,3,5,2,3,3,5,5,5,3,3,1,1,1,1,1,2,5,
3,5,5,3,5,3,3,5,5,3,3,5,5,5,2,3,2,3,3,3,3,3,5,3,5,3,2,5,3,1,1,1,1,3,2,5,3,5,5,3,3,3,5,2,5,5,3,5,3,3,
3,5,5,5,3,5,5,3,5,2,3,3,2,5,3,5,3,3,3,3,3,3,3,5,5,3,3,3,3,5,3,3,5,3,5,5,3,3,3,5,5,5,3,5,5,2,3,5,5,3,
5,5,3,1,1,1,1,3,5,5,2,5,3,5,5,3,3,3,5,3,3,1,1,1,1,1,3,5,3,3,5,3,3,2,2,3,5,5,5,3,5,3,1,4,1,1,3,2,3,5,
3,3,3,3,5,5,5,3,5,3,5,3,3,2,2,3,2,3,3,5,3,5,5,3,5,5,5,3,2,3,5,2,2,3,3,3,2,3,5,3,3,5,3,3,3,3,3,3,5,2,
2,3,5,3,3,5,3,2,3,3,3,3,5,3,3,3,5,2,5,5,5,3,3,5,5,3,3,2,5,3,5,3,3,5,3,5,3,5,5,3,5,5,3,5,5,2,3,3,3,3,
5,3,3,2,2,3,5,5,2,3,3,5,3,3,4,1,1,4,1,3,5,3,5,5,3,3,5,2,3,5,2,5,5,5,3,3,3,5,3,5,3,3,2,2,5,2,2,5,3,3,
5,5,5,3,3,3,3,5,5,3,3,1,1,1,1,3,5,5,2,3,5,5,3,5,5,3,5,3,3,4,1,1,4,3,5,3,5,5,3,5,3,2,5,3,1,1,1,1,1,2,
3,3,3,3,5,3,5,5,3,3,5,3,3,5,5,3,2,3,5,3,5,3,5,2,5,3,5,5,3,5,3,5,5,3,3,5,2,4,1,1,1,1,1,4,3,5,3,3,3,3,
4,4,4,3,3,3,3,5,5,3,5,3,5,3,3,3,3,1,1,1,4,3,5,3,1,1,1,4,3,3,3,5,3,5,2,5,5,3,5,3,3,5,5,3,3,5,5,3,5,5,
3,3,5,3,5,5,5,3,3,3,5,3,5,2,3,3,3,1,1,1,1,2,5,3,3,5,5,3,2,5,5,3,3,3,3,3,5,5,3,3,2,5,3,5,3,4,6,1,1,1,
1,1,3,3,5,5,3,5,5,3,5,2,5,3,5,5,5,3,2,3,5,3,2,5,3,1,1,1,4,1,6,1,2,5,3,2,5,3,5,5,5,3,5,5,3,5,3,3,3,3,
3,5,5,2,5,5,3,3,3,2,3,3,1,1,4,1,3,1,1,1,1,1,3,5,5,2,3,3,5,3,3,5,5,3,2,5,3,5,3,3,3,5,3,3,5,2,3,3,5,5,
3,3,2,3,3,3,3,3,2,5,5,3,5,5,5,3,3,3,5,3,5,2,5,5,5,3,3,5,3,3,5,5,5,3,5,5,5,3,3,2,3,3,3,3,3,3,3,3,5,5,
2,3,3,3,3,2,3,3,5,2,3,5,3,3,2,3,3,5,3,1,1,1,1,3,2,5,3,3,4,1,1,1,2,1,1,1,1,1,2,5,2,4,4,1,4,3,3,5,5,3,
5,5,3,3,5,3,5,5,2,5,2,1,1,4,4,3,5,5,3,5,5,2,5,5,3,3,5,5,3,3,2,2,3,5,2,3,5,3,3,5,5,5,3,5,5,3,3,3,3,3,
3,5,5,5,3,1,1,1,1,1,2,3,4,1,1,1,1,6,4,1,3,3,5,3,3,2,2,2,3,2,3,5,5,3,5,5,5,3,5,3,3,5,2,5,5,3,1,1,1,1,
3,3,5,5,3,5,3,3,3,5,5,3,5,3,5,2,5,2,3,3,5,3,1,1,1,1,1,3,5,5,3,5,3,4,1,6,1,6,4,2,3,5,2,2,5,5,2,5,3,3,
3,2,5,3,3,5,3,3,3,3,5,5,3,5,5,3,3,5,3,5,3,5,3,5,3,3,5,3,5,3,4,1,4,1,3,5,5,3,1,4,1,1,3,3,3,5,2,5,5,3,
5,3,2,3,3,5,3,2,3,3,2,2,5,3,4,1,1,1,3,3,3,3,3,2,3,2,1,1,6,1,3,3,3,3,5,3,5,3,5,3,2,3,2,5,5,5,2,3,5,3,
3,3,5,5,3,5,5,3,5,5,2,5,3,1,1,1,1,3,5,2,5,5,2,3,5,3,5,5,3,5,3,5,5,3,5,3,3,2,2,3,3,5,5,3,3,3,5,5,2,3,
2,5,5,3,5,3,5,3,5,5,5,3,3,2,5,3,1,1,1,4,1,4,3,2,1,4,6,6,1,3,3,3,5,2,5,3,3,1,1,1,1,2,5,5,3,3,5,2,3,5,
5,5,3,3,1,4,6,1,3,5,5,3,5,5,2,1,1,1,1,4,2,5,5,3,2,3,5,5,3,5,3,5,3,2,3,3,3,3,3,3,3,5,2,5,5,2,3,3,3,3,
5,3,3,3,3,3,2,3,5,3,3,3,5,3,3,5,3,3,3,2,4,4,4,3,2,3,3,2,5,3,5,3,2,5,3,2,5,3,5,3,5,3,1,1,1,1,3,1,4,1,
1,3,2,3,5,5,5,3,5,2,3,3,3,5,3,5,3,5,3,1,1,1,4,3,3,5,3,3,5,3,4,4,4,3,5,5,3,5,5,5,3,3,5,2,3,3,3,3,3,5,
2,2,1,6,1,1,4,3,5,3,5,5,2,3,3,6,1,1,6,2,3,5,3,3,3,3,5,3,3,3,2,1,4,1,6,3,5,5,3,5,3,3,5,5,3,3,3,5,5,3,
5,3,3,5,5,3,5,2,3,5,5,5,3,3,2,5,5,3,3,2,5,3,5,3,2,3,5,5,5,3,5,3,1,4,4,1,4,1,4,3,3,2,3,5,5,3,5,3,5,5,
5,3,5,3,3,4,1,1,1,1,3,3,5,2,1,1,6,1,2,5,5,5,2,5,3,5,3,4,4,4,2,5,5,2,3,3,3,5,5,5,3,5,3,3,1,4,1,1,6,1,
3,3,2,5,5,5,3,3,3,3,3,3,5,5,3,2,3,5,3,3,2,2,5,5,5,2,3,3,5,5,5,2,5,3,5,3,2,5,5,2,5,3,5,5,5,3,5,2,5,2,
5,2,3,1,6,1,1,4,1,1,3,3,5,3,5,5,3,1,1,1,1,1,3,3,5,2,3,3,2,1,1,1,1,1,1,1,4,3,3,3,3,5,5,3,3,3,3,5,5,3,
3,3,5,3,3,2,3,2,3,2,5,2,3,3,1,1,1,1,1,1,1,3,3,5,3,1,1,1,1,1,4,1,4,1,2,5,5,3,3,5,5,5,3,5,5,3,5,5,2,3,
5,3,3,5,3,3,5,5,3,2,2,3,2,5,5,2,5,2,5,5,5,3,5,5,3,2,5,3,5,3,3,1,1,4,1,3,1,1,4,4,2,2,5,5,5,3,3,5,5,5,
2,5,3,3,2,3,3,4,1,1,1,1,1,1,1,3,3,2,3,5,5,5,2,5,3,3,3,3,3,3,5,3,2,1,1,6,1,1,3,3,1,4,1,1,1,4,1,1,1,2,
5,3,5,5,3,5,3,3,5,5,2,3,5,2,5,3,2,5,2,5,5,5,2,3,3,3,2,5,5,2,3,1,1,1,1,2,5,3,3,2,5,3,5,5,2,2,1,1,1,1,
3,5,3,5,3,5,2,3,5,5,2,5,5,3,3,4,1,4,4,2,5,3,5,2,2,3,5,3,4,4,4,2,1,1,1,1,1,3,3,3,5,5,3,5,3,5,5,2,5,3,
5,5,3,3,2,5,3,3,5,3,1,1,1,1,3,6,1,4,1,3,5,5,3,5,5,5,3,3,2,5,5,2,5,5,3,5,3,3,3,3,3,2,3,3,3,2,3,3,3,3,
4,4,4,3,3,5,5,3,5,3,5,5,3,5,5,5,3,4,4,4,3,2,5,3,2,3,3,5,3,3,5,5,3,3,2,5,3,2,3,3,5,5,3,5,5,3,3,2,5,3,
3,3,3,3,3,5,5,3,3,5,2,5,2,3,5,5,3,3,2,3,5,2,3,3,5,3,2,2,5,3,5,3,3,5,3,5,3,5,3,5,3,2,3,3,2,3,3,5,5,2,
2,2,5,3,5,5,3,2,5,2,3,5,3,2,3,5,5,3,2,5,3,5,3,5,3,5,3,2,3,3,5,3,3,3,5,5,5,3,3,5,5,3,3,2,5,5,3,1,1,1,
1,1,4,1,1,3,1,1,1,1,2,5,2,5,2,5,5,3,3,3,3,5,3,2,2,2,1,1,1,1,3,1,4,1,1,1,1,1,1,1,1,3,5,3,2,3,2,3,1,1,
1,1,1,1,1,1,1,1,1,3,2,1,1,4,1,3,5,5,2,4,4,4,2,5,3,1,1,1,1,1,3,5,3,2,4,4,6,1,1,1,1,3,3,1,1,4,1,3,5,5,
3,1,1,1,1,3,1,1,6,4,1,2,5,5,3,5,3,5,5,2,3,3,1,1,4,1,2,3,5,5,3,5,3,5,5,5,2,3,3,5,3,2,5,3,3,3,3,3,3,2,
5,3,5,5,3,5,3,3,3,3,3,5,3,2,1,4,1,1,1,3,5,3,5,3,5,3,3,3,3,5,5,3,2,3,3,5,2,3,1,1,1,1,3,5,5,3,3,3,3,3,
5,5,2,5,2,5,3,5,3,3,3,3,3,3,3,3,3,5,3,2,5,5,2,3,2,3,3,3,3,5,3,5,3,5,5,3,5,5,3,2,5,5,2,3,5,3,3,3,5,5,
5,3,3,5,3,3,3,5,5,3,5,3,3,3,2,3,5,3,5,5,2,5,5,3,3,3,3,5,3,3,3,3,5,5,3,2,3,5,5,3,5,3,3,3,6,1,1,4,3,3,
5,2,3,3,4,1,1,1,1,2,5,3,2,5,3,3,3,5,5,3,3,5,2,3,3,3,3,5,3,5,3,5,5,3,5,5,5,3,3,5,3,3,5,3,5,3,3,2,5,3,
5,3,3,5,3,5,3,3,5,3,5,5,3,3,5,2,3,3,1,1,1,1,1,3,5,5,2,2,5,3,3,3,3,3,3,5,5,3,3,3,4,4,4,3,3,5,5,5,2,3,
5,2,5,3,5,2,2,3,5,3,3,5,5,3,5,5,2,3,3,5,3,3,3,5,2,3,3,3,5,3,3,3,5,3,1,1,1,1,3,5,2,3,3,3,2,5,3,2,5,3,
5,3,3,2,3,3,3,5,3,3,2,2,3,1,1,6,1,3,5,5,2,2,5,5,2,5,3,5,3,5,5,3,3,5,2,5,5,3,3,3,3,5,3,2,2,3,3,2,5,3,
5,3,5,5,5,3,3,5,3,3,3,3,2,3,5,5,3,3,5,2,3,3,3,5,3,3,3,5,5,3,5,2,5,2,3,5,5,3,3,2,6,1,1,1,2,2,3,3,3,2,
3,3,5,3,3,5,3,2,3,5,3,5,5,3,3,5,5,3,5,5,2,5,5,2,5,3,5,5,5,3,5,3,3,5,3,5,5,3,3,5,3,3,5,3,5,5,5,3,2,3,
3,3,3,3,3,2,5,3,3,5,5,5,3,3,3,2,5,3,2,3,3,3,2,3,3,5,5,3,5,3,5,3,2,5,2,5,5,2,3,5,2,3,5,2,5,5,3,3,3,5,
3,3,3,5,5,3,3,2,3,3,3,5,5,3,2,3,5,5,3,1,1,1,1,2,5,5,3,1,1,1,1,1,3,3,3,5,5,3,3,5,3,3,3,3,1,1,1,1,3,2,
3,2,3,3,5,3,5,3,2,2,5,3,3,5,3,3,5,3,3,1,1,1,1,2,3,5,2,2,5,3,3,5,5,3,5,5,3,2,5,5,5,3,3,5,3,3,3,5,5,5,
3,3,3,3,5,3,5,3,5,3,1,1,1,1,6,3,5,5,5,3,5,5,5,2,3,3,5,2,5,5,5,3,5,2,3,2,3,3,5,5,3,3,5,5,3,2,5,3,2,3,
3,2,5,3,5,5,5,3,3,3,2,3,5,5,3,3,3,3,5,5,3,5,5,2,2,5,3,5,5,3,5,3,3,3,5,3,5,5,5,3,3,2,2,3,5,3,2,5,3,5,
2,5,5,3,3,5,3,3,5,3,2,5,3,3,2,5,5,2,3,3,2,3,5,3,3,3,3,5,5,5,3,3,5,2,5,5,3,2,3,5,3,5,3,3,2,5,3,5,3,3,
5,5,3,3,3,5,3,3,5,3,3,3,3,3,3,5,2,3,5,5,2,5,3,3,3,2,5,3,3,5,3,3,2,3,3,5,3,2,3,3,5,5,2,3,3,3,2,5,3,3,
3,3,5,3,5,5,3,5,2,3,2,5,5,3,3,3,5,2,3,5,5,3,5,3,5,3,5,3,3,5,5,3,3,3,3,5,3,2,3,2,5,3,5,5,2,5,3,2,2,3,
5,3,3,5,5,3,3,3,5,5,3,5,3,2,1,1,1,1,1,2,3,3,3,3,3,3,5,3,3,3,3,3,2,3,5,3,3,5,3,5,3,3,3,2,5,3,3,2,3,5,
5,5,3,3,3,5,2,3,5,5,3,3,5,3,5,5,5,2,3,5,3,5,3,3,5,2,5,3,3,5,3,3,2,3,3,5,3,5,5,3,2,3,3,5,5,3,3,3,2,2,
5,3,5,3,5,3,5,3,1,1,1,1,3,5,2,2,5,3,5,3,3,2,5,3,3,3,3,3,3,2,2,2,2,5,5,3,3,3,3,2,3,1,1,1,1,2,5,5,3,5,
5,2,3,3,5,3,5,3,3,5,3,5,3,2,3,5,5,5,2,3,3,4,4,4,3,1,1,1,1,3,4,1,1,1,2,5,3,5,3,5,5,5,2,5,2,3,3,5,5,3,
5,3,5,5,2,5,3,3,4,4,4,3,2,2,3,2,3,3,3,5,5,3,5,3,3,5,5,3,3,5,2,3,5,2,1,1,1,1,3,3,3,5,5,2,5,3,5,3,3,3,
5,5,5,3,5,5,3,3,3,3,5,3,5,5,3,5,5,3,3,5,5,3,5,5,2,3,1,1,1,1,3,5,3,5,2,3,2,5,2,2,3,3,6,1,6,1,4,3,3,3,
5,3,5,3,5,3,2,3,5,3,3,5,3,5,3,3,5,5,3,3,3,3,5,3,5,3,3,3,3,3,5,3,5,3,5,3,4,4,4,3,3,3,3,5,3,3,3,3,3,3,
3,2,5,3,5,5,2,3,5,5,3,3,5,5,3,3,5,3,3,5,3,3,3,3,3,2,3,5,3,3,2,5,5,3,5,2,3,5,5,5,3,5,3,3,5,5,2,3,5,5,
2,1,1,1,1,1,1,3,3,5,5,3,3,4,4,4,3,5,3,5,5,2,5,3,3,2,5,3,2,3,5,3,3,2,3,2,3,3,3,2,2,5,3,3,5,5,5,3,5,5,
5,2,5,2,5,2,5,2,3,5,2,5,5,2,3,2,2,5,3,1,1,1,1,3,3,5,3,5,5,3,5,3,3,3,3,3,3,3,3,3,5,3,3,3,5,5,3,3,3,5,
5,2,3,3,3,2,3,3,5,5,3,2,3,1,4,1,4,3,5,5,3,5,5,5,3,5,5,2,2,2,3,3,3,5,2,5,3,2,3,5,5,3,3,5,3,3,3,3,5,5,
3,3,2,5,3,5,2,2,3,5,3,5,3,5,3,3,3,5,3,3,3,3,3,3,2,3,5,3,3,5,2,5,3,3,3,2,3,1,1,1,1,3,2,2,3,3,3,2,5,2,
5,3,3,5,3,1,1,1,1,3,5,3,3,2,2,5,3,5,3,3,3,3,3,5,5,3,3,5,3,3,3,3,2,5,3,5,2,2,2,2,3,3,5,2,5,3,5,3,3,5,
3,3,2,5,2,3,3,5,3,5,5,2,3,3,5,3,5,3,3,3,3,3,3,5,5,3,2,2,2,2,2,1,4,1,1,1,1,1,2,3,5,3,3,5,3,3,3,3,3,5,
5,5,3,5,3,3,3,5,5,3,3,2,5,5,3,5,5,3,3,3,3,5,3,5,3,5,3,5,3,5,5,3,2,5,2,5,5,3,5,5,2,3,2,5,5,3,3,2,2,5,
3,5,3,3,5,3,5,3,2,3,3,3,3,1,1,1,4,3,5,5,3,5,5,2,2,3,3,5,5,2,2,3,5,5,3,3,5,2,5,5,3,3,3,5,3,3,3,3,3,3,
3,5,3,3,3,3,3,5,3,3,5,2,3,5,2,3,5,5,5,3,5,5,5,3,5,3,3,5,5,5,2,3,3,2,5,3,5,3,3,5,5,3,3,3,3,3,3,2,3,5,
3,5,2,5,5,3,5,3,5,3,3,5,2,5,5,3,3,3,3,5,3,3,3,3,3,2,2,5,5,3,3,5,5,3,3,1,1,1,1,2,3,5,3,3,5,3,5,5,2,3,
5,5,3,3,5,3,3,3,3,3,3,3,5,3,3,3,3,5,5,5,3,3,3,5,3,3,5,3,5,2,3,3,5,5,3,3,5,2,3,3,2,3,3,3,3,5,5,3,2,3,
3,3,3,5,3,3,2,5,5,3,5,3,5,3,1,1,1,1,2,3,3,2,3,2,5,2,5,2,5,2,5,2,3,2,3,2,3,2,3,3,5,3,5,2,3,3,3,3,5,3,
5,3,5,3,3,3,5,5,3,2,5,3,5,3,3,3,5,2,5,3,3,5,3,2,4,1,1,1,1,1,1,1,1,1,4,3,5,5,3,5,5,5,2,2,2,5,5,3,3,3,
3,3,3,3,3,5,5,3,3,2,3,5,2,5,5,3,2,3,5,5,5,3,1,1,1,1,1,1,1,2,5,5,5,3,5,3,1,4,1,1,1,1,2,3,3,5,5,3,3,2,
5,5,3,3,3,2,3,2,3,5,3,3,2,3,2,3,3,5,5,5,3,5,5,5,3,2,5,5,5,2,3,3,3,3,3,1,1,1,4,2,3,5,3,2,5,3,3,3,2,3,
3,3,5,3,3,3,3,5,5,3,3,3,3,3,2,2,5,3,3,3,5,2,3,3,2,3,5,5,2,5,5,3,3,5,3,3,5,5,3,3,3,3,2,5,3,5,5,2,5,3,
3,3,5,5,2,2,2,3,3,3,5,3,3,3,2,5,2,5,5,3,5,5,3,2,5,3,5,3,3,2,3,3,5,3,5,3,2,3,3,3,3,3,
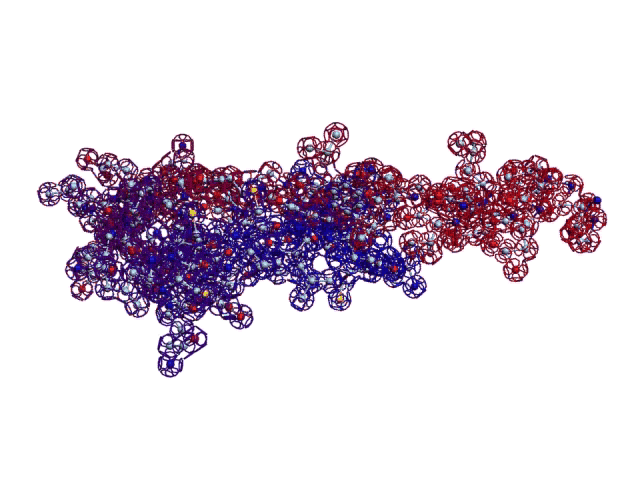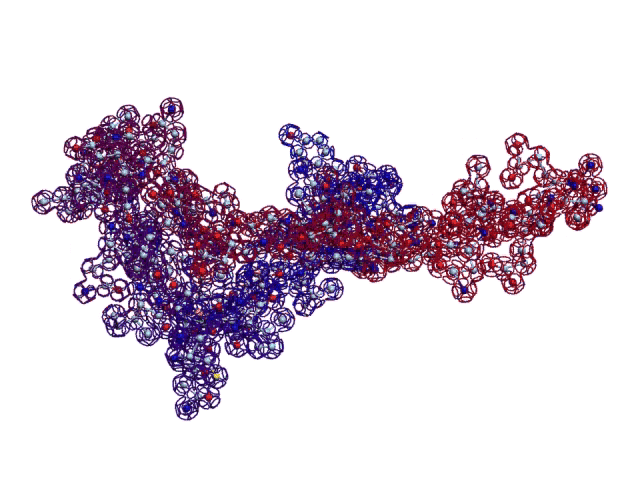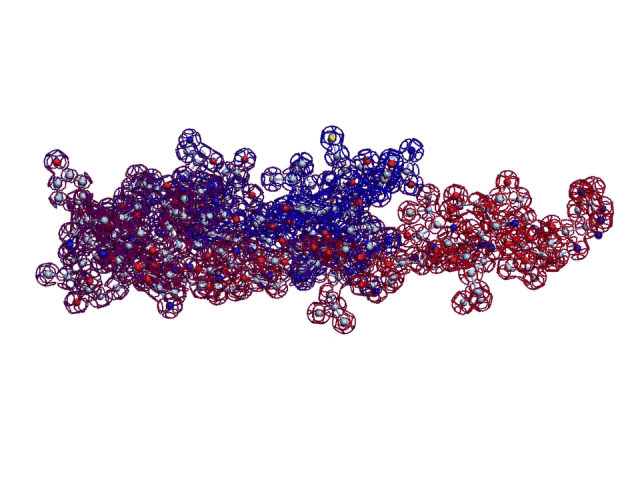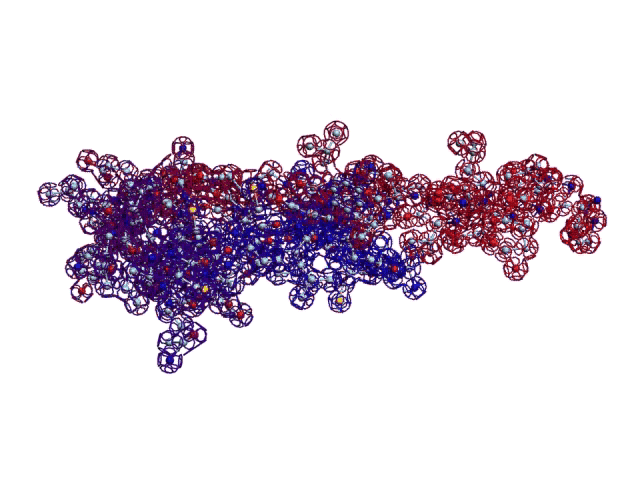
Вторичная структура (Пикотех-2017) весит около 6 мегабайт в стандарте PNG: https://cloud.mail.ru/public/KKjH/goMgmMGAn
На яндекс-фотки не грузится. Но самый интересный фрагмент удалось вырезать в программе Paint:
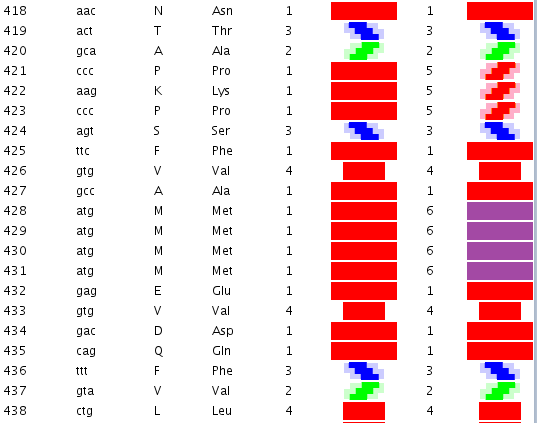
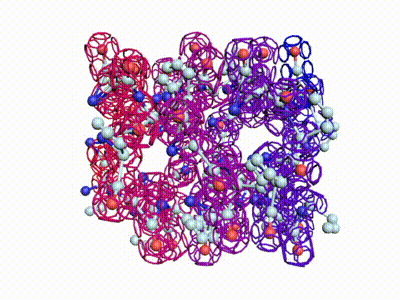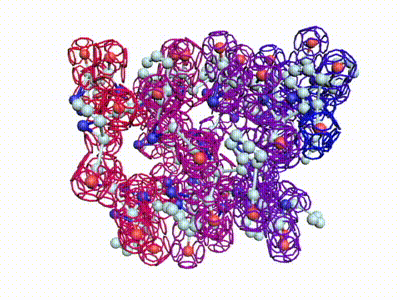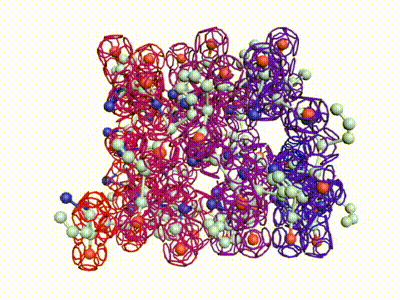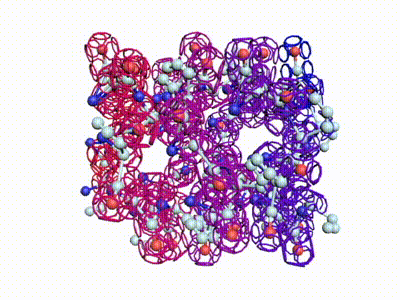
Сиреневым цветом отмечен виток необычной метиониновой спирали. В отличие от альфа-спирали у неё более длинный шаг. Метиониновая спираль отличается от обычной альфа-спирали примерно как резьба с более крупным шагом от резьбы с менее крупным шагом:
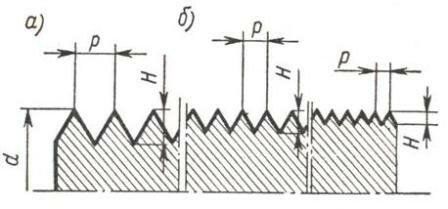
Шаг метиониновой спирали примерно на 25% крупнее шага обычной альфа-спирали. При этом на один виток приходится тоже 3.6...3.7 аминокислотных остатка Met.

Ещё один интересный участок этого белка 3916...4164. Он содержит все типы композиционных кодов: 1,2,3,4, но ... не содержит ни одного витка спирали! Интересно оценить вероятность случайной комбинации композиционных кодов такого типа. Напомню, что спираль возникает при трёхкратном (или более) повторении кодов 1, 4 или их комбинации. При этом третий код должен быть "4", либо спиральных кодов (1,4), идущих подряд, должно быть больше 3.

Белковая молекула кончается витком полилизиновой пи-спирали, похожей на винт Архимеда.