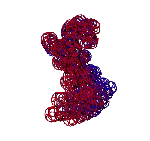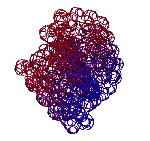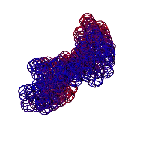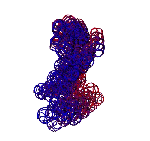Тезисы на Конференцию "ДНК-2020"
Рентгено-структурный анализ: что мы видим?
Высоко периодичные структуры белков
ПИКОТЕХНОЛОГИЯ БЕЛКОВ
СТРУКТУРЫ И ФУНКЦИИ
Благодаря метду Пикотехнологии мы видим интересующую нас белковую молекулу в трёхмерном изображении, с любой нужной степенью детализации. Получаем координатные файлы всех атомов. А также - аналитику для исследования физико-химических взаимодействий и решения других задач.
В нашем распоряжении -100%-е 2D и 3D структуры белковых молекул с точностью до пикометра и все без исключения участки.
В отличие от РСА, который "видит" только 3% белков и не замечает десятки крайних аминокислотных остатков - "хвостов, не различает типы плотных спиралей и не видит неплотные, новая технология показывает практически 100% структуры 100% белков.
При этом только 3% белков подлежат кристаллизации, т.е. исследованию методом рентгено-структурного анализа
Гипотеза о функционале реактора
Гипотеза о функционале броуновской частицы
Молекулы, соответствующие по форме ДНК
Многослойные спирали и мезомеханизмы
(белки для работы с ДНК. рекуператоры, теплотрансформаторы, спиральные конвееры, белковая вязь и др.)
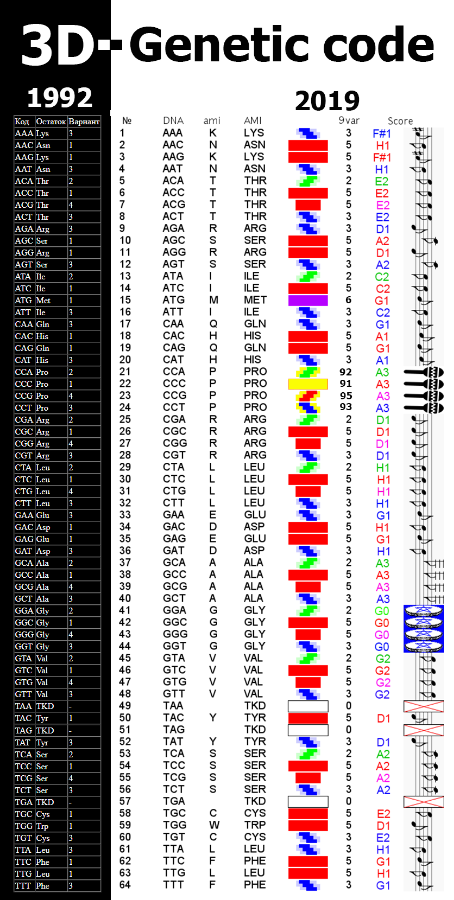
Версия 7var
Содержание 2D диаграмм Пикотех - табличная функция 3D структуры
2D структуры Пикотехнологии отображают, по какому пространственному алгоритму цифровой (цветовой) композиционный код белка организует форму 3D структуры молекулы
Одна клеточка - один аминокислотный остаток
Согласно Таблице композиционного генетического кода,
- виток альфа-спирали - 4 аминокислотных остатка с кодом "1",
- виток бета-спирали - 3 аминокислотных остатка с кодом "2",
- виток 310-спирали - 3 аминокислотных остатка с кодом "4",
- виток пи-спирали - 5 аминокислотных остатков с кодом "3"
Альфа спираль или одиночный альфа-код - красный
Бета спираль или одиночный бета-код - зелёный
Пи спираль или одиночный пи-код - синий
310-код - оранжевый
Код метионина в составе альфа-310 спирали - сиреневый
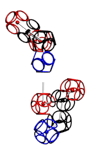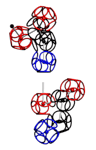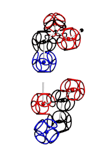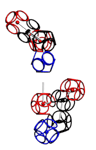
БЕЛОК AAS15574
Шарики обозначают аминокисльтные остатки. Цвета соответствуют Композиционноу (3D) генетическому коду:
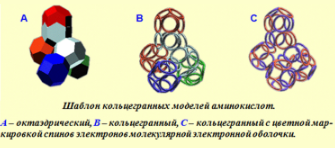
ШАБЛОН КОЛЬЦЕГРАННЫХ МОДЕЛЕЙ АМИНОКИСЛОТ
Кольцегранный подход к моделированию молекул приводит к детерминированным решениям структурных задач, т.к. фактор неопределённоти в начальных условиях таких задач отсутствует.
Кольцегранные модели наглядно показывают, что все аминокислотные остатки (АО) имеют единый общий структурный шаблон (отличаются только радикалами), который служит базовым конструктивным элементом белковой цепи.
Поскольку форма структурного шаблона всех АО строго задана, как и алгоритм присоединения последующего к предыдущему, мы получаем точно детерминированную стреометрическую форму белковой цепи.
Алгоритм задан Композиционным генетическим кодом, задающим угол поворота последующего АО относительно предыдущего.
2D диаграмма содержит всю информацию об объёмной форме белковой молекулы
Новейшая версия 3D Пикотех будет выдавать стереометрическое изображение белковой цепи одновременно с цеветной диаграммой вторичной структуры. Эта версия будет интерактивной, позволяющей моделировать девиацию кодируемых и некодируемых углов.
Структура 2D диаграммы Пикотех содержит полную и однозначную информацию о пространственной форме белковой цепи
СОКРАЩЁННАЯ 2D ДИАГРАММА ПИКОТЕХ
Красный - код альфа-спирали.
Оранжевый - код 310-спирали.
Розовый - одиночный код альфа/310 спиралей.
Голубой - код пи-спирали.
Зеленый - код бета-спирали.
Сиреневый - код метиониновой спирал. У неё более крупный шаг "резьбы", чем у обычной альфа-спирали.
Черный в сокращённом представлении и белый в развернутом означают либо неизвестный код, либо конец трансляции.
Циклическое повторение цветов - программная спираль.
Программные спирали - повторение последовательности композиций. Например, один код альфа-спирали, затем один код пи-спирали. n(35) задаёт программную спираль, а n3 или n5 - простые спирали (пи-спираль и альфа-310-спираль).
232323 - программная 23-спираль
141414 - программная 14-спираль
 |
 |
 |
 |
 |
| Альфа-спираль | Бета-спираль | Пи-спираль | 310-спираль | Метиониновая спираль |
Кушелев: В структуре белка используется ряд подструктур, похожих на базовые, но построенных по микропрограммам. Это позволяет уточнить интерпретацию экспериментальных данных из Protein Data Base, а в ряде случаев заменить неправильную интерпретацию на правильную.
3212-спираль, в которой регулярно повторяется фрагмент композиционного кода 3212, напоминает бета-спираль, только имеет пятиричную квазисимметрию, как и семеричная спираль. Сбоку видны характерные ~прямые углы, как и в бета-спирали.
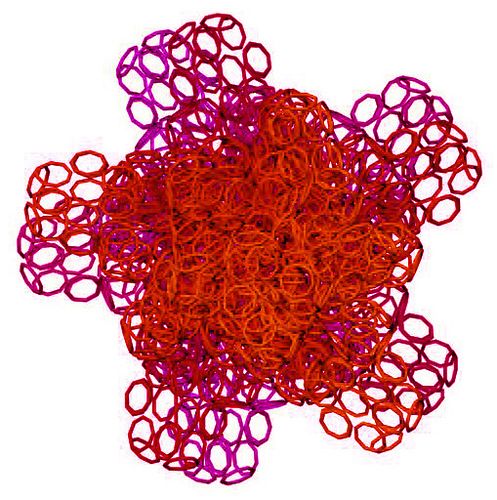
Любопытный повтор композиционного кода: 23323323. Поворот до совмещения с соседним аминокислотным остатком происходит на 45 градусов, значит спираль имеет 8-ричную квазисимметрию.

Эта конструкция напоминает складчатый бета-слой. Композиционный код: 13323133231
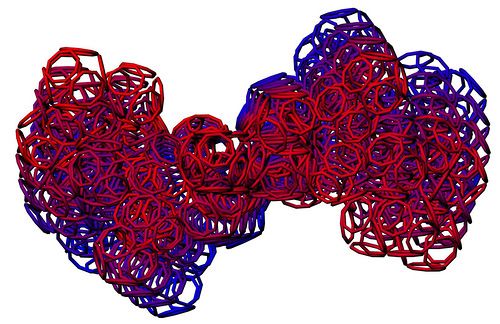
Большинство исследователей полагает, что бета-тяжи плоские, т.е. "спираль с поворотом каждой следующей аминокислоты на 180 градусов. Пикотехнологические модели показывают, что это не тяжи, а именно правые спирали (2.5 остатка на виток). 310-спираль - правая. Имеет примерно 3 остатка на виток. Альфа-спираль тоже правая. Имеет ~3.6 остатков на виток. Пи-спираль - левая имеет примерно 4.1 остатка на виток.
Модели всех типов простых спиралей показаны здесь: http://nanoworld88.narod.ru/data/302.htm
Моделирование аминокислотных остатков http://nanoworld88.narod.ru/data/279.htm
РАЗВЁРНУТАЯ 2D ДИАГРАММА ПИКОТЕХ
Композиционный генетический код, версии 4var - 9 var
Код "2" (спираль - не спираль).
Код 4var более подробный (альфа-, бета-, пи-, 310-).
Код 6var ещё более подробный. В нём есть различие "одиночный альфа-код", "код альфа-спирали", "код 310-спирали", "Код метионина в составе спирали".
Код 7var различает одиночный код пи-спирали от кода в составе пи-спирали .
Код 8var от отличает одиночные коды от спиралей
Код 9 var показывает одиночные пролины и пролиновые участки
Триплеты ДНК кодируют аминокислоты. В упрощённом представлении третья буква триплета управляет углом поворота. Это показывают пикотехнологические 3D модели аминокислотных остатков. Один аминокислотный остаток можно повернуть относительно другого на углы 0, 120 и 240 градусов. Для описания пространственных структур белковых молекул представлен Композиционный генетический код.
Кушелев: В упрощённом представлении третья (как бы лишняя) буква управляет углом поворота. Это я понял, когда сделал 3D модель аминокислотного остатка. Один аминокислотный остаток можно повернуть относительно другого на углы 0, 120 и 240 градусов. Когда я в этом убедился, построив пластмассовые модели нескольких белковых молекул, начались уточнения. Ведь водородные связи между соседними витками спирали могут слегка менять углы поворота. Анализ белковых структур помог составить таблицу композиционного генетического кода
Кушелев: Официальная наука "знает" альфа-спираль, 310-спираль, пи-спираль и бета-тяж (не знает, что спираль). В тралиционной науке спирали не кодируются. Для того, чтобы узнать их код, нужно применить композиционный генетический код, что, собственно, я и сделал в 1992-ом году.
В упрощённой модели пикотехнологии учитывается поворот вокруг одной оси на 3 разных угла. Вариант "альфа" в дальнешем расщепляется на "Альфа-спираль", "310-спираль", "одиночный альфа-код".
Согласно методу Пикотехнологии, электрон имеет "скелет" в виде кольца диаметром типа 1.7 ангстрема (для внешних оболочек водорода, углерода, азота, кислорода).
Кольцевые "скелеты" электронов формируют кольцегранные электронные оболочки и кольцевые механизмы молекул.
Модели этих механизмов показывают, как складываются альфа-спираль, 310-спираль, смешанные альфа-310-спирали, пи-спирали и бета-спирали, которые оказались не по 2 остатка на виток, и белковые цепочки в целом
В настоящее время считается, что белок в неопознанной части структур, полученных методом РСА, "не имеет определенной структуры", но пикотехнология показала, что это не так.
В программе "Пикотех" вся структура определена.
Пикотехнология не может определить структуры, полученные не по программе рибосомой. Если после сборки белка он обработан ферментами, например, разрезан и переделан, то программа "Пикотех" об этом не даёт информации.
Построение 2D Диаграмм и структур 3D Пикотех
Алгоритм построения Пикотехнологических моделей белков основан на открытии Композичионного генетического кода, см. монографию А.Кушелева и В.Соколик "Геометрия живого наномира. Пикотехнология белков".
Структуры 2D Пикотех строятся с достоверностью 100% в автоматическом режиме.
Структуры 3D Пикотех в зависимости от состава молекул строятся в автоматическом режиме (геометрический алгоритм) либо в ручном режиме (с учётом физико-химических взаимодействий ,свойств сустава Pro и других факторов). Метод построения структур 3D Пикотех на базе 2D структур Пикотех является ноу-хау Лабораторатории Наномир.
В настоящее время делается 3D версия, позволяющая автоматиировать поправки, производимые в ручном режиме, моделировать девиацию кодируемых и некодируемых углов, а также учитывать физико-химические взаимодействия и структруные особенности Met и сустава Pro
ЧТО ОЗНАЧАЕТ НОТНАЯ ЗАПИСЬ В ПРАВОЙ ЧАСТИ ДИАГРАММЫ ПИКОТЕХНОЛОГИИ?
У каждой аминокислоты есть радикал, который "отзванивает" ноту на частоте, которую я рассчитал по формуле пружинного и математического маятников
Формула эта имеет вид: f = sqrt(1/(Sum(ml))), где
f - частота
m - масса атома в радикале
l - Расстояние атома до точки закрепления радикала
Суммируются произведения масс на расстояние до точки закрпления для одного радикала. Из обратной величины извлекается квадратный корень.
Понятно, что это - грубая оценка резонансных частот, поэтому они многократно уточнялись, в частности, по гармоничности мелодий, звучащих в зонах активных центров белковых молекул. Эта работа ещё далека до завершения. Очевидно, что в частотах есть ошибки. Поэтому встречаются "фальшивые нотки"
ИНФОРМАТИВНОСТЬ ПИКОТЕХНОЛОГИИ
ТАБЛИЧНАЯ ФУНКЦИЯ
По сути методом Пикотехнологии мы видим стреометрически каждый аминокилотный остаток, его положение относительно предыдущего и последующего в пространстве, объёмные пространсвенные изображения любого звена белковой цепи и всей цепи в целом, а также располагаем координатным файлами всех атомов и электронов. Также мы получаем данныедля анализа физико-химических взаимодействий и интерактивными возможностями менять углы поворотов/сгибов при сборке молекудлы.
Пикотехноологический столбец предстарвляет собой описание строго детерминированной стереметрической модели
ПОЧЕМУ ПИКОТЕХНОЛОГИЯ ТОЧНА НА 100%
Специалисты по РСА не в курсе, что электрон имеет "скелет" в виде кольца диаметром типа 1.7 ангстрема (для внешних оболочек водорода, углерода, азота, кислорода... Кольцевые "скелеты" электронов формируют кольцегранные электронные оболочки и кольцевые механизмы молекул.
Модели этих механизмов показывают, как складываются альфа-спираль, 310-спираль, смешанные альфа-310-спирали, пи-спирали и бета-спирали, которые оказались не по 2 остатка на виток.
Кольцегранные модели помогут специалистам по РСА более точно определять структуры белков и не только.
Пикотех не может определить структуры, полученные не по программе рибосомой. Если после сборки белка он обработан ферментами, например, разрезан и переделан, то программа "Пикотех" об этом не знает. Так что полностью пренебрегать РСА не стоит. Но в плане дополнения РСА "весит" в несколько раз меньше, чем "Пикотех".
В современных базах данных структура белка изображается в виде опознанных участков альфа и 310-спиралей.
Здесь спиральные участки (альфа- и 310-) показаны красным цветом. Остальное - неопознанная часть. В настоящее время считается, что белок в неопознанной части не имеет определенной структуры, но пикотехнология показала, что это не так.
В программе "Пикотех" вся "неопознанные" структуры определены.
Данный белок состоит из программных спиралей, но в стандартном представлении его покажут либо вообще без структуры, либо изобразят кусками альфа- и 310-спиралей. Оба варианта в корне ошибочны.
Ещё один типовой пример ошибки - изображение структуры коллагена тройной альфа-спиралью. В действительности структура данного коллагена представляет собой одинарную, но программную 335-спираль:
Хотя специалисты по РСА (рентгеноструктурному анализу) заявляют, что точность метода достигает долей ангстрема (меньше размера одного атома), но в действительности не могут на протяжении десятилетий отличить тройную альфа-спираль от программной 335-спирали коллагена. Это значит, что они не могут достичь атомной точности даже сегодня. Реальная погрешность РСА находится в диапазоне от нескольких атомных радиусов до сотен атомных радиусов. Например, "хвосты" белковых молекул (до 50 аминокислотных остатков) РСА может вообще не заметить. А это - сотни атомных радиусов в длину.
РСА часто ошибается при распознании спиральных участков. Иногда вообще не замечает или показывает там, где спирали нет. А перепутать альфа-спираль с 310- Пи- или программной спиралью – повсеместно.
Вместо тройной спирали коллагена" программа Пикотех показывает одиночную, но "трёхзаходную" программную 335-спираль. Другими словами форма похожа, но микроструктура другая. И получена она в автоматическом режиме, т.е. по табличной функции.
РАБОТОСПОСОБНОСТЬ ПИКОТЕХНОЛОГИИ
Кушелев: Работоспособность пикотехнологии имеет смысл демонстрировать во-первых на уровне вторичной структуры длинных спиралей, в т.ч. программных. Для них легко получать точные 3D-модели.
Акцент имеет смысл сделать на замыкании циклов лизоцима. Случайно получить замыкание циклов невозможно. Вероятность 1/30 000 000 000.
А для программных спиралей случайность кодв имеет вероятность типа 4^-3982=4^-2398
Далее имеет смысл демонстрировать возможности Пикотех на примере белков заказчиков. Заказчики сами увидят свои белки более точно, чем о них рассказывают специалисты по РСА. Именно это и привлечёт заказчиков к более тщательному ознакомлению и использованию новой технологии. Они должны убедиться в её работоспособности и эффективности не по наслышке.
У меня уже был негативный опыт работы с заказчиками через посредников. Искаженная и неполная информация о новой технологии только отпугивает заказчиков. Они думают, что результаты работы Пикотех должны полностью совпасть с результатами РСА. Это не так. На уровне вторичной структуры Пикотех имеет 100%-ную достоверность, а РСА на уровне 70%. 97% белков для РСА вообще недоступны, т.к. они не кристаллизуются.
Преодолеть барьер слепой веры в РСА - дело тонкое. С помощью "испорченного телефона" задача не решается. Так что ждём потенциальных заказчиков. В этом случае есть шанс донести до них информацию о новой технологии без искажений.
Пять надежных методов проверки Пикотехнологии
Первый - достоверная корреляция с данными РСА. Конечно, РСА не даёт высокой достоверности, но она часто выше 50%.
На таких белках, как инсулин, гемоглобин, окситоцин корреляция приближается к 100%, т.к. в окситоцине, например, есть замкнутые циклы через дисульфидные мостики. Это позволяет проверить и РСА, и Пикотех.
Второй - замкнутые через дисульфидные мостики циклы типа окситоциновых. Вероятность случайного замыкания шести циклов, насчитывающих десятки аминокислотных остатков из разных окситоцинов, порядка 1/30 000 000 000. Речь идёт о вероятности для каждого цикла - все шесть могут случайно замкнуться с вероятностью (1/30 000 000 000)^6.
Третий - самоповерка на примере сверхдлинных базовых и программных спиралей, где наблюдается 100%-ная корреляция между вторичной и первичной структурами.
Четвёртый - микроскопия. 3D модель "монстра" первой хромосомы хороша тем, что её можно проверить, рассматривая молекулу белка в флуоресцентный микроскоп. "Монстр" первой хромосомы такой крупный, что его видно в микроскоп в отличие от мелких белков. В частности, диаметр начальной фрактальной петли (правая часть) примерно 100 нанометров, т.е. 0.1 микрона.
Пятый - сопоставлени модельных экспериментов Пикотех и натурных экспериментов.
Фрактальная программная спираль первой хромосомы (Нейробластома)


Модельные эксперименты с помощью 3D принтера показыват, что набором из 9 вариантов по три угла (композиционный, транспоцизионный и пролиновый) можно описать только часть белковых структур, например, прямой участок альфа-, 310-, пи-, бета-спиралей, произвольную последовательность композиционных (3D) кодов в том случае, если нет ограничений по водородным, вандерваальсовым и гуковским связям. В реальных белках, которые часто содержат программные спирали, такие ограничения есть, поэтому набором из 9 вариантов по 3 угла большинство белковых структур описать не удастся. Без учета физики, т.е. на уровне геометрического алгоритма. Учесть физику трудоёмко, но можно добавить базу данных для программных спиралей, что сильно расширит возможности геометрического алгоритма. И хотя программных спиралей великое множество, но ходовых программных спиралей не так уж много, а редкие программные спирали придётся исследовать с учетом физики. Пока в ручном режиме. Наиболее актуальной является интерактивная 3D-версия, где можно визуально наблюдать за появлением различных ограничений в процессе построения модели белка.
Модель девиации композиционного угла
Пикотехнология как дополнение к РСА:
Программа Пикотех не может полностью заменить РСА. Однако она очень усиливает самые слабые стороны РСА, а именно достоверно показывает вторичную структуру ,и ближний порядок расположения атомов третичной структуры в момент сборки белковой молекулы рибосомой.
Чувствительность РСА такова, что он не замечает не только отдельных атомов, но и отдельных аминокислотных остатков. Более того, "хвосты" белковых молекул, которые не кристаллизуются, РСА вообще "не видит", а эти "хвосты" могут насчитывать до 50 аминокислотных остатков. Также РСА в соновном не различает альфа-, 310- и пи-спирали и не видит бета-спирали (которые из-за низкой атомной плотности сливаются с фоном рентгенограммы).
Особый класс образуют 97% белковых молекул, которые не кристаллизуются. Про них РСА просто ничего не знает, а программа Пикотех так же достоверно показывает их вторичную структуру и ближний порядок расположения атомов в третичной структуре.
Литература
1.Монография А.Кушелев, В.Соколик "Геометрия живого наномира. Пикотехнология белков" https://www.morebooks.de/ru/search?utf8 … 0%BE%D0%B2
Предисловие рецензентов и авторов. Начало монографии
2. Кушелев А.Ю., Соколик В.В. Пикотехнология – новый подход в моделировании пространственной структуры белка / Заочная Международная научно-практическая конференция «Современная наука: тенденции развития» (24 января 2012), Краснодар: НИЦ Априори. – 2012. – С.203-207.
3. Соколик В.В. Предсказание пространственной структуры белка insilico на основе информации генома и геометрического алгоритма – альтернатива квантово-механическому подходу // Материалы Международной научной конференции «Математическое и компьютерное моделирование в биологии и химии. Перспективы развития» (28-30 мая 2012), Казань. – 2012. – С.155-158.
4. Sokolik V.V. Protein is coded in genome and synthesized in ribosomes as a structural template of a rotameric version sequence of peptide bound configuration // The International Moscow Conference on Computational Molecular Biology, МССМВ-11, Moscow. – 2011. – P. 347–348.
5. Sokolik V.V. Algorithm of protein structural template decoding according to its determined nucleotide sequence // Fist International Conference “Fundamental medicine: From scalpel toward Genome, Proteome and Lipidome”, Pax Grid Virtual Conferences, Kazan. – 2011. – P. 117–119.
6. Sokolik V.V. Modeling of the individual structural template of protein on determining it nucleotide sequences // VII Международная конференция по биоинформатике, регуляции структуры геномов и системной биологии. BGRS\SB-2010, Новосибирск. – 2010. – С. 275.
7. Соколик В.В. Способ моделирования пространственной структуры белка по детерминирующей его нуклеотидной последовательности // Биофизический вестник. – 2010. – Вып. 24 (1). – С. 31-45.