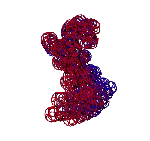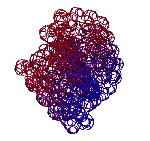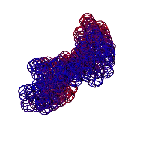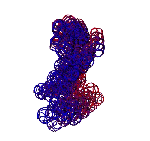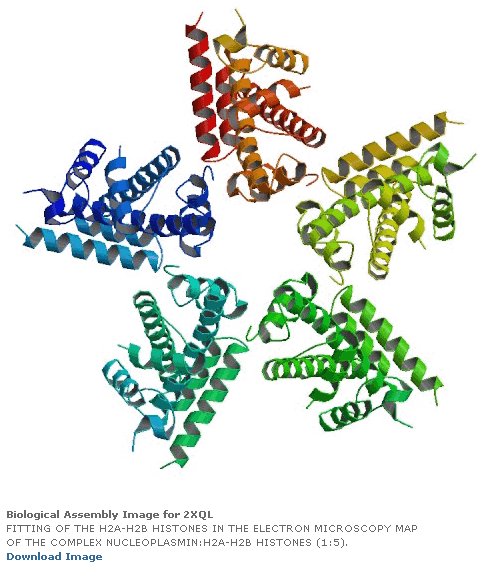ТЕЗИСЫ "ДНК - 2020"
Пикотехнологические модели аминокислот [41,39,27] помогли открыть 3D генетический код [27] в 1992-ом году. В таблице 3D генетического кода каждому триплету кодирующей нуклеотидной последовательности соответствует не только аминокислота, но и вариант её установки в растущую структуру белка. Например, триплет GGC кодирует глицин в составе альфа-спирали, GGA – глицин в составе бета-спирали, GGG – глицин в составе 310-спирали, GGT – глицин в составе пи-спирали. Для проверки таблицы 3D генетического кода оказалось достаточным знать расположение дисульфидных мостиков в структурах белков. Вероятность случайного замыкания модели фрагмента белка через дисульфидный мостик ничтожна, а для большого набора моделей фактически исключена. Дополнительным подтверждением существования 3D генетического кода является сильная корреляция результатов, полученных по таблице 3D генетического кода с данными РСА [27,1]. и КД. С помощью таблицы 3D генетического кода и системы BLAST Protein удалось найти ряд антигомологов типа «биназа-барназа». Нашлись десятки белков, содержащих протяженные участки поливалина, полиглицина, полисерина, полипролина, полиглутамина и т.д, и т.п., которые имеют разные вторичные структуры. Например, первичная структура у всех белков VVVVVVVVV…, а вторичные структуры разные, т.е. один белок-поливалин представляет собой прямую альфа-спираль, другой – прямую пи-спираль, третий – 310-спираль, четвертый – бета-спираль. РСА практически не позволяет отличить альфа-спираль от пи-спирали, но с помощью метода кругового дихроизма (КД) это возможно и является подтверждением существования 3D генетического кода «мокрым» экспериментом.
В процессе исследования обнаружились необычные регулярные структуры, в частности, поливалина. Например, вместо альфа-, 310-, пи-, бета-кодов встречается повторяющаяся последовательность. Например, альфа-пи-альфа-пи-альфа-пи… В таком случае рибосома формирует не фундаментальную, а программную спираль белка, у которой может быть любое число аминокислотных остатков на виток и любой шаг спирали. Более того, это может быть вообще не спираль, а так называемая вязь, которая похожа, например, на вязаный шарф, где нить образует петли и на обратном пути проходит через эти петли, образуя «вязанную» белковую ткань. Последовательность 3D кодов может быть фрактальной, т.е. альфа-альфа-альфа-альфа-пи-альфа-альфа-альфа-альфа-пи-. В таком случае рибосома строит фрактальную программную спираль, например, из коротких участков альфа-спиралей. Встречаются фрактальные спирали, состоящие из чередующихся участков альфа- и пи-спиралей, альфа- и бета-спиралей и т.д. Это приводит к образованию гигантских по масштабам микромира спиральных конструкций. Изучая гигантские фрактальные программные спирали удалось обнаружить подвижные мембраны, например, из пролинов, образующих заход фрактальной спирали. Пролин обладает дополнительной степенью свободы на изгиб. Если в программной спирали повторяются участки, например, по 4 пролина, то формируется заход фрактальной спирали шириной в 4 Pro и длиной в десятки фундаментальных спиралей. Это напоминает спиральную лестницу с шириной ступени в 4 Pro и числом ступеней в несколько десятков. Но в отличие от жестких ступеней, пролиновые ступени, а значит и вся пролиновая винтовая часть лестницы, оказывается мембраной, которая может вибрировать на гиперзвуковой частоте. Мощность такого фрактального активного центра белка может в сотни раз превосходить гиперзвуковую акустическую мощность простого активного центра. Если же эта фрактальная спираль сопряжена по форме со спиралью ДНК/РНК, то она может дистанционно управлять состоянием ступеней ДНК.
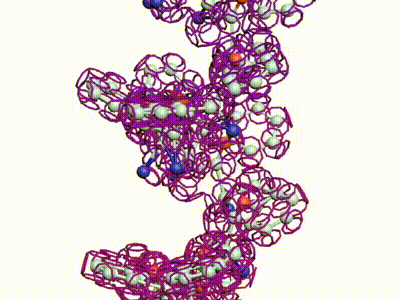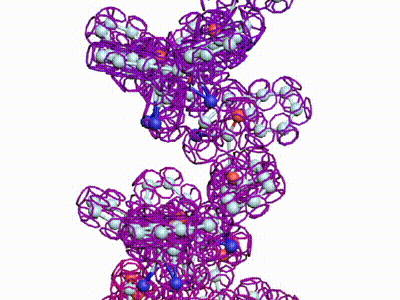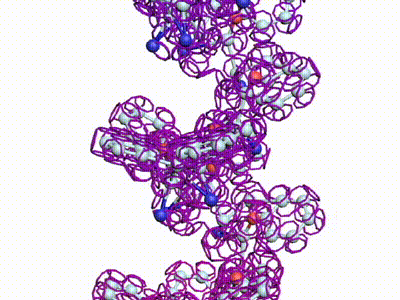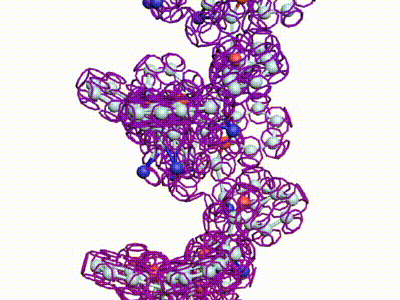
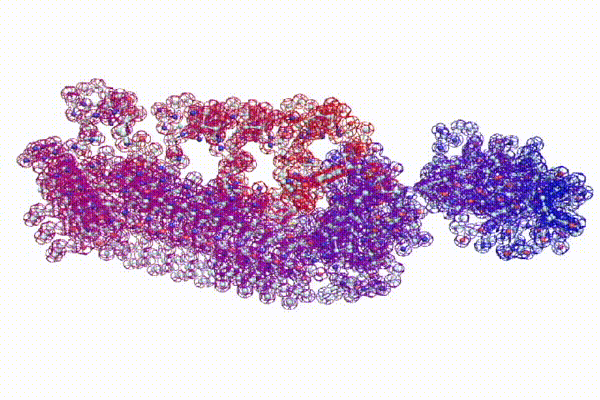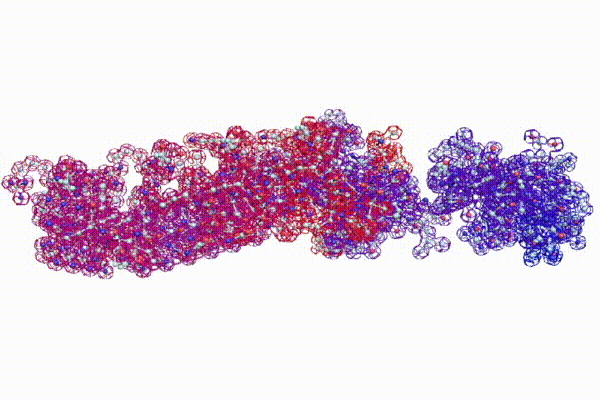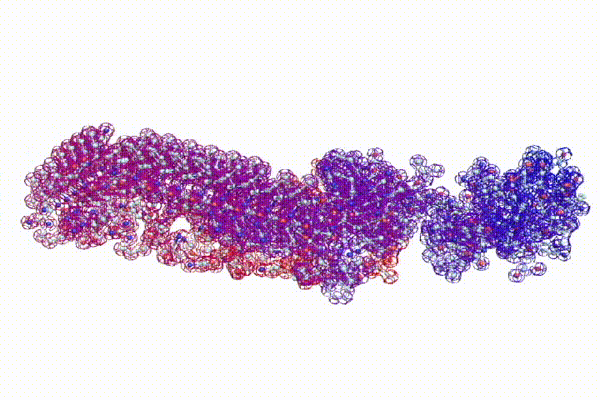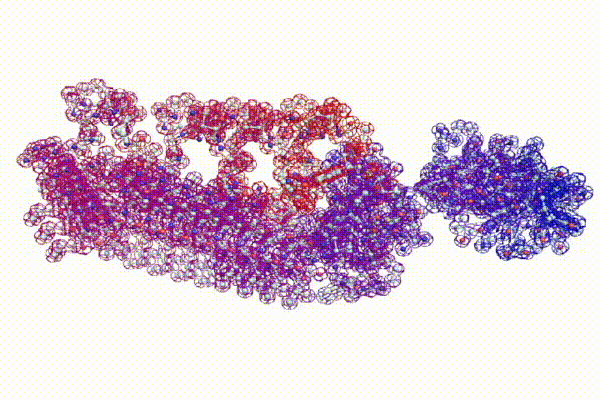
С помощью программы Picotech 3D, в основе которой лежит таблица 3D генетического кода и пикотехнологические модели аминокислот, удалось обнаружить несколько белков, форма которых сопряжена с формой ДНК на длине до 25 ступеней ДНК.
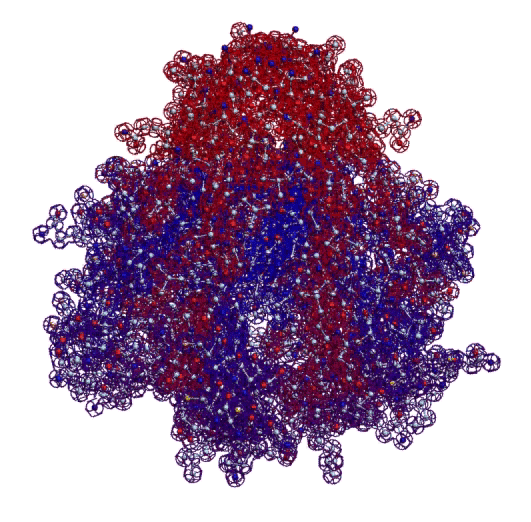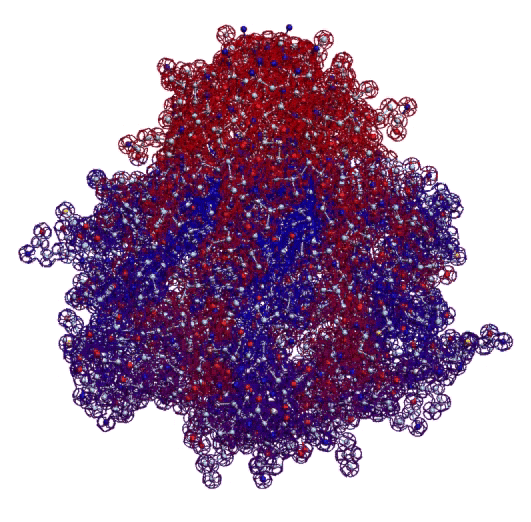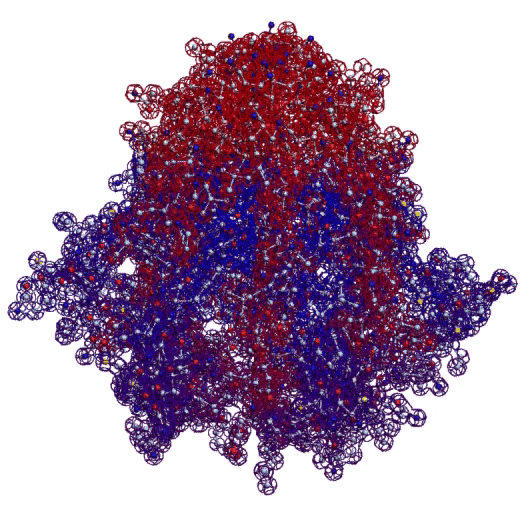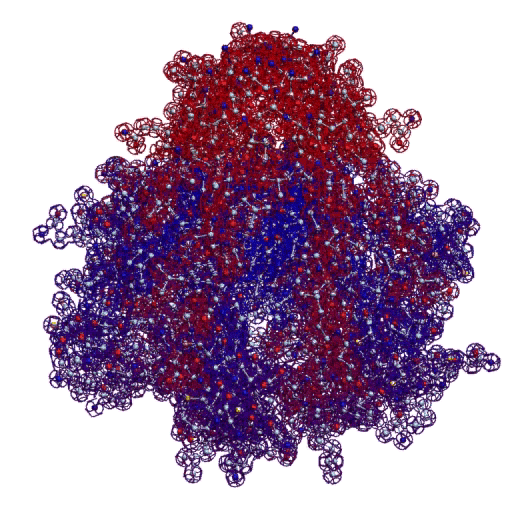
В 2017-ом году был запущен онлайн-сервис по определению белковых структур. Геномная версия программы Picotech 2D позволяет определять вторичные структуры целыми геномами, в частности, все 110 000 структур белков человека были определены с помощью геномной версии программы Picotech 2D за неделю на двух персональных компьютерах. Во время этого исследования были обнаружены несколько невероятных белковых структур. В частности, гигантская фрактальная спираль нейробластомы. Источник
Биография А.Ю.Кушелева

ССЫЛКА НА ВИДЕО

ССЫЛКА НА ВИДЕО

Гексанитробензол
ЭЛЕКТРОН КАК БУБЛИК СО СТОЯЧЕЙ ВОЛНОЙ
ЭЛЕКТРОН - ЗАКОЛЬЦОВАННЫЙ ФОТОН
ВОЛНОВЫЙ СВОЙСТВА И КОЛЬЦЕВЫЕ СТРУКТУРЫ ЧАСТИЦ МИКРОМИРА


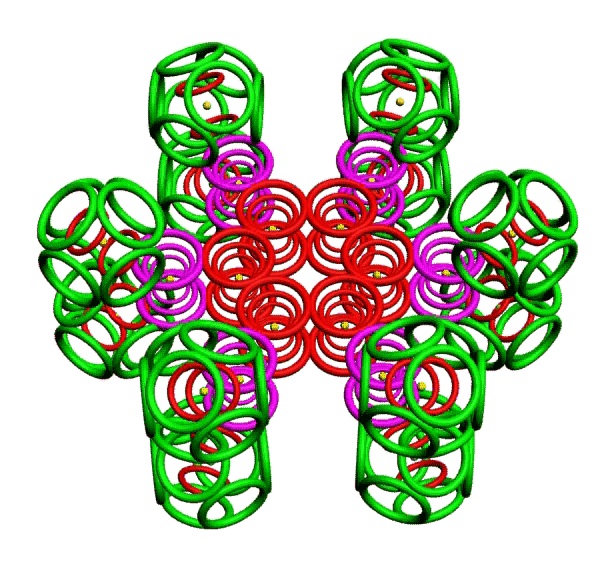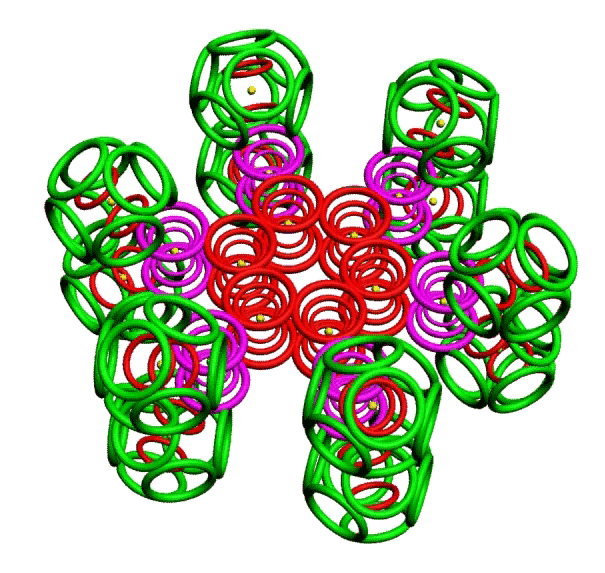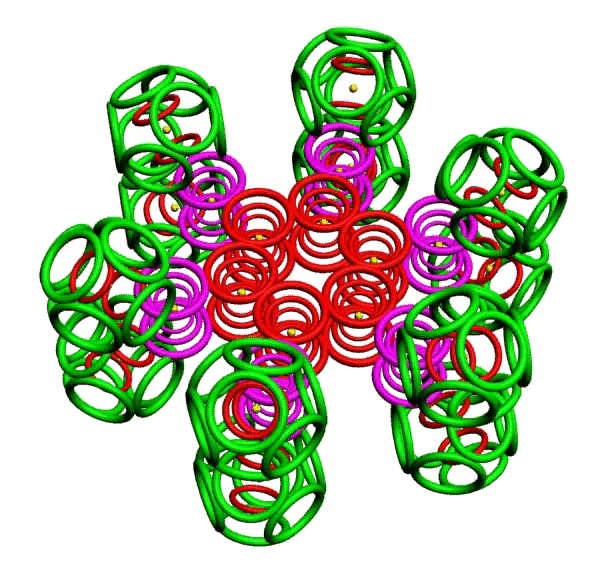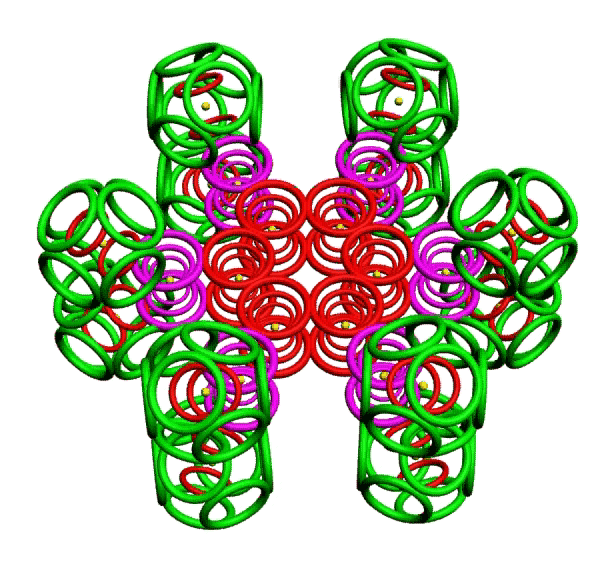
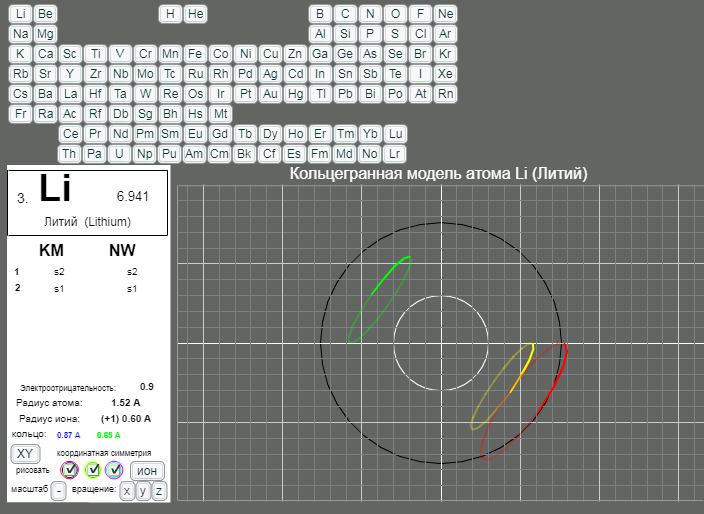
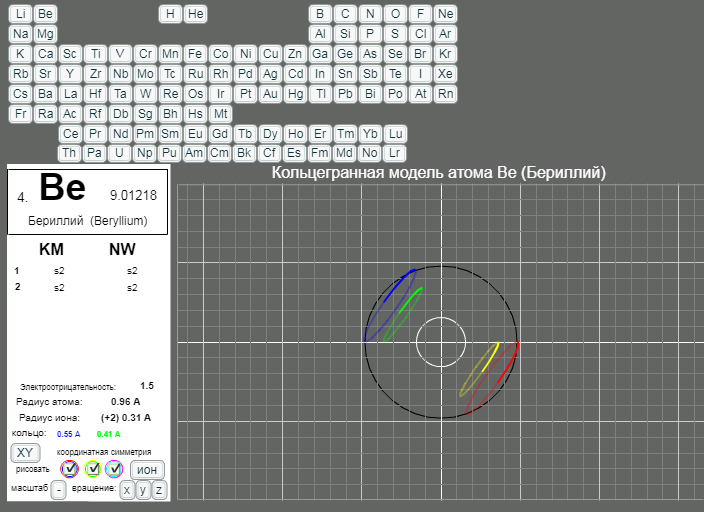
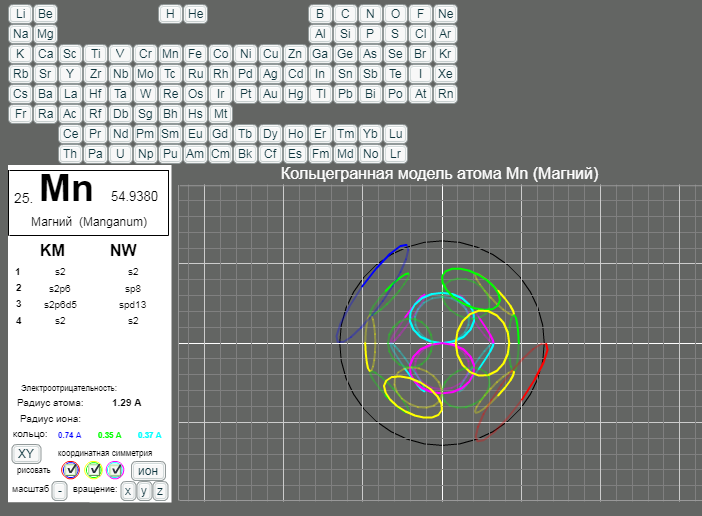
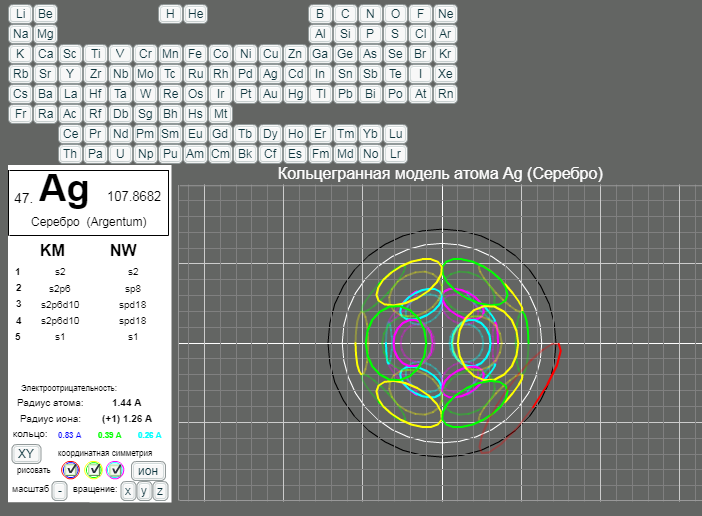
КОЛЬЦЕГРАННОЕ ПРЕДСТАВЛЕНИЕ АТОМОВ

Демонстрации кольцегранных моделей атомов Таблицы Менделеева
Кольцегранную модель атома впервые предложил К. Снельсон в 1960 году (http://www.kennethsnelson.net/main.htm). Независимо от него, в 1988 г. такую модель атомов предложил А. Кушелев.
В этой модели атома электроны представляются кольцами, имеющими магнитные и электрические свойства. Благодаря магнитным свойствам, электроны "смагничиваются" в сферические оболочки, которые вкладываются друг в друга. Особенно устойчивыми являются оболочки, составленные из 2(2*(1*1)), 8(2*(2*)), 18(2*(3*3)), 32(2*(4*4)) колец-электронов.
Утверждается, что на основе этой модели атома, можно использовать геометрический подход для построения объединений атомов. При этом возможны варианты взаимодействия атомов: встраивание оболочек, смагничивание электроных оболочек, объединение внешних электронных оболочек, ионная связь, ...
Примеры кольцегранных моделей:
- Кольцегранная модель серного газа (SO3).
- Кольцегранная модель кристаллического углерода.
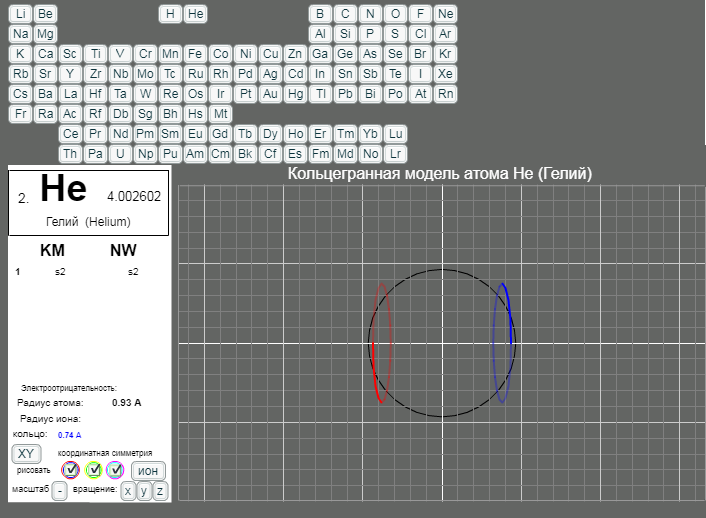
- Данная страница предназначена для демонстрации кольцегранных моделей атомов. Выбор химического элемента осуществляется кнопками с кодовыми именами элементов.
Модели масштабные. Крупный шаг сетки соответсвует расстоянию 1А. Мелкий шаг сетки равен 0.2А. Кнопка выбора масштаба "+"/"-" увеличивает/уменьшает масштаб рисунка в два раза. Размеры атома и иона рисуются окружностями черного и белого цветов. (Размеры радиусов атомов и ионов взяты из брошюры - учебного пособия для школьников ВЗМШ при МГУ - Батаева Е.В., Батаев В.А. "Общая химия. Части 1 и 2". На стр. 35 имеется "Периодическая таблица элементов по Кэмбеллу с атомными и ионными радиусами".)
Рисуются только три внешних уровня кольцегранной модели атома/иона. Синим и красным цветом рисуются кольца самого внешнего уроня. Желтым и зеленым цветом рисуются кольца первого внутреннего уровня. Голубым и розовым рисуются кольца второго внутреннего уровня. Моделируется смагниченность колец. Кольца синего, зеленого, голубого цветов представляют одну полярность, а кольца красного, желтого и розового цветов представляют противоположную полярность. Флажки в цветных кружках разрешают/блокируют рисование соответствущих кольцегранных уровней модели.
Кнопкой "XY"/"XYZ" осуществляется выбор 3/2 координатной симметрии модели. Расположение колец и размер колец для этих моделей разные.
Кнопка "атом"/"ион" определяет моделирование иона/атома текущего выбранного химического элемента.
Кнопки "x"/"X", "y"/"Y", "z"/"Z" задают/отменяют вращение модели вокруг осей координат. Ось X имеет направление слева направо. Ось Y направлена сверху вниз. Ось Z направлена на наблюдателя.
Расстояния между электронными уровнями и порядок заполнения электронов на уровнях в данной версии задаются достаточно произвольно. Варианты оболочек, имеющие двухкоординатную симметрию, не реализованы.


ЧИТАТЬ ОНЛАЙН ПОЛНОСТЬЮ
МОНОГРАФИЯ 2016 И НЕКОТОРЫЕ ПУБЛИКАЦИИ
В.Соколик, А.Кушелев. Монография
"Геометрия живого наномира. Пикотехнология белков"
LAP LAMBERT Academic Publishing (2016-08-04 )
Купить книгу
Читать фрагменты книги, предисловие рецензентов, послесловие авторов
В.В. Соколик "Никакой дополнительной информации, большей, чем та, что содержится в ДНК, для сворачивания белка не требуется" Матеріали ХІ Укріїнського біохімічного конгресу (06-10 жовтня 2014), The Ukrainian Biochemical Journal, 2014, V. 86, №5 (Supplement 1), P. 37-38.
В. Соколик. СТРУКТУРНЫЕ ПРЕДПОСЫЛКИКИ АГРЕГАЦИИ БЕТА-АМИЛОИДНОГО ПЕПТИДА
V.Sokolik. ALGORITHM OF PROTEIN STRUCTURAL TEMPLATE DECODING ACCODING TO ITS DETERMINED NUCLEOTIDE SEQUENCE
V. Sokolik. Modeling of the 3D structure of apoliprotezin E3 under its determining nucleotide sequence.
V.V.Sokolik. “Implementation of the 3D genetic code of proteins is isoacceptor tRNAs”, Journal Medical and Clinical Chemistry. 2019.V. 21, No. 3 (Appendix). P. 46-47. ISSN 2410-681X)
Виктория Соколик, научные труды

Монография посвящена 3D-структуре молекул и полимеров живых систем. Дан анализ современного понимания таких фундаментальных понятий, как физический объём атома, химическая связь, генетический код. На основе статистического анализа экспериментальных данных о структуре белка обосновано кодирование его вторичной структуры и структурного полипептидного шаблона в геноме. Предложена дополненная таблица генетического кода белков и пептидов, которая легла в основу геометрического алгоритма программ декодирования структурного шаблона белка Molecular Constructor и Picotech. Сформулирована гипотеза о перекодировании информации третьего нуклеотида кодона в соответствующий ротамер пептидной связи непосредственно 3D-структурой изоакцепторной тРНК. Математический анализ сопряженности значений углов φ и ψ (карта Рамачандрана) выявил периодичность их изменения, что позволило обосновать механизм посттрансляционного фолдинга белка. Книга рассчитана на специалистов, занимающихся исследованиями в области молекулярной биологии, биоинформатики, биохимии, биофизики
ПРЕАМБУЛА

В настоящее время одним из популярных алгоритмов моделирования пространственной структуры белка, который исходит из его аминокислотной последовательности, является способ построение модели по гомологии на основе общего структурного шаблона.
Разработан новый способ моделирования структурного шаблона белка по детерминирующей его нуклеотидной последовательности, поскольку информация о топологии вторичной структуры белка и его индивидуальном структурном шаблоне содержится непосредственно в гене каждого белка. Эта информация может быть декодирована в соответствии с таблицей генетического кода структурного шаблона белка авторскими программами Пикотех (PT) и Молекулярный конструктор (МС) в виде координатного файла в pdb-формате.
Важным достоинством метода является то, что можно построить структурный шаблон индивидуально для любого неизвестного белка лишь “прочитав” детерминирующую его нуклеотидную последовательность. Такой шаблон служит единой матрицей для пространственной структуры всех молекул данного белка в ходе посттрансляционного фолдинга in vivo и виртуального фолдинга или докинга in silico.
Модели белковых молекул в Пикотехнологии предполагают наличие "жестких" кольцеобразных структур в электронных оболочках атомов, соединяющихся в многогранники. Это позволяет нам типологизировать формы участков белковых молекул и точно описать их в трехмерном изображении, с точностью до пикометра.
ФОРМИРОВАНИЕ УГЛОВ БЕЛКОВЫХ СПИРАЛЕЙ
Согласно методу Пикотехнологии, электрон имеет "скелет" в виде кольца диаметром типа 1.7 ангстрема (для внешних оболочек водорода, углерода, азота, кислорода).
Кольцевые "скелеты" электронов формируют кольцегранные электронные оболочки и кольцевые механизмы молекул.
Кольцегранные модели наглядно показывают, что все аминокислотные остатки (АО) имеют единый общий структурный шаблон (отличаются только радикалами), который служит базовым конструктивным элементом белковой цепи.
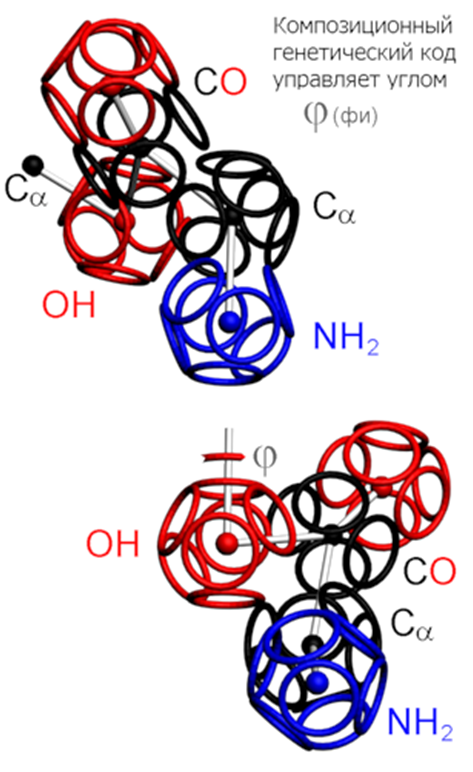
Поворот последуюего аминокислотных остатков относительно придшествующего ему по оси симметрии tRNA формирует углы альфа-310-, бета-, пи-спиралей соответственно - при этом алгоритм формирования следующего звена белковой цепи задаётся композиционным генетическим кодом (композиционными углами) .
Точность следующего порядка достигается посредством учета дополнительно транспозиционных и пролиновых углов.
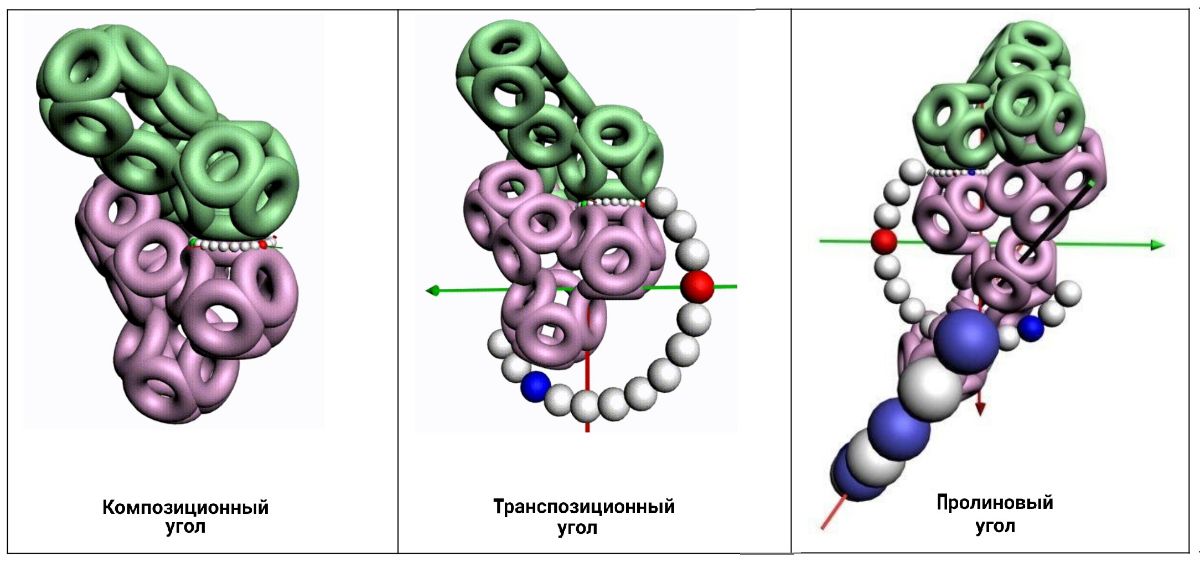
По шкале из самых мелких шариков откладывается композиционный угол. По шкале из средних шариков - транспозиционный угол. По шкале из крупных шариков - пролиновый угол. Эти три угла лежат в попарно ортогональных плоскостях. Интерес представляет тот факт, что любой аминокислотный остаток может деформироваться не только на транспозиционный угол, но и на пролиновый. Другое дело, что пролиновая деформация обычных аминокислотных остатков не превышает 10 градусов. Если же требуется больше, то очередной аминокислотный остаток заменяется на пролин (Pro), и диапазон пролиновой деформации увеличивается от +-10 до +-60 градусов. В результате зона роста белковой молекулы охватывает более 2/3 полного телесного угла.
Транспозиционный угол генами не кодируется. Он определяется из модельного эксперимента, если есть водородные связи, например, в альфа-, 310-, пи-спирали. Между спиралями этот угол может быть существенно другим, т.е. отличаться на десятки градусов.
Пролиновый угол (самые крупные шарики), тоже не кодируется генами. В спиралях он фиксирован в крайнем (нулевом) положении т.е. если пролин находится в составе спирали, то его пролиновый угол равен нулю (кольцо замкнуто). Если же он на конце спирали или между спиральными участками, то пролиновый угол можно меняться от нуля где-то до 160 градусов. Кольцо разворачивается в полукольцо.
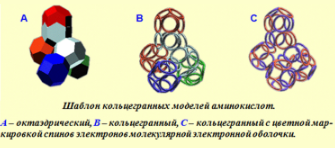
ШАБЛОН КОЛЬЦЕГРАННЫХ МОДЕЛЕЙ АМИНОКИСЛОТ
Все аминокислотные остатки имеют единый общий структурный шаблон, который служит основным конструктивным элементом белковой цепи.
Поскольку шаблон имеет определенную стереометрическую форму, а алгоритм состыковки звеньев цепи задан строгим образом композиционным кодом, то форма белковой цепи становится точно предсказуемой как науровне фрагментов, так и на уровне целостной молекулы.
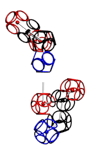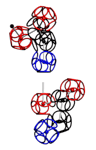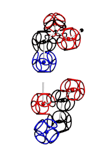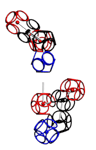
УГЛЫ ПОВОРОТА АМИНОКИСЛОТНЫХ ОСТАТКОВ
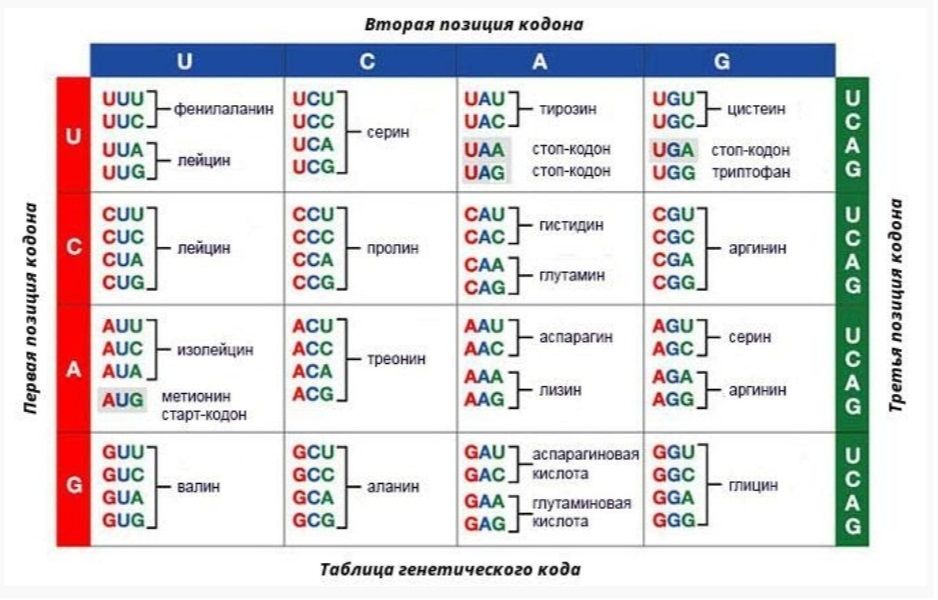
Триплеты ДНК кодируют аминокислоты. В упрощённом представлении третья буква триплета управляет углом поворота. Это показывают пикотехнологические 3D модели аминокислотных остатков. Один аминокислотный остаток можно повернуть относительно другого на углы 0, 120 и 240 градусов. Для описания пространственных структур белковых молекул представлен Композиционный генетический код.
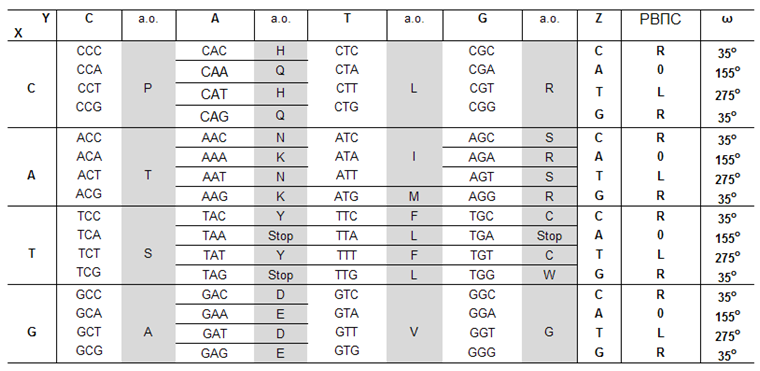
КОМПОЗИЦИОННЫЙ ГЕНЕТИЧЕСКИЙ КОД
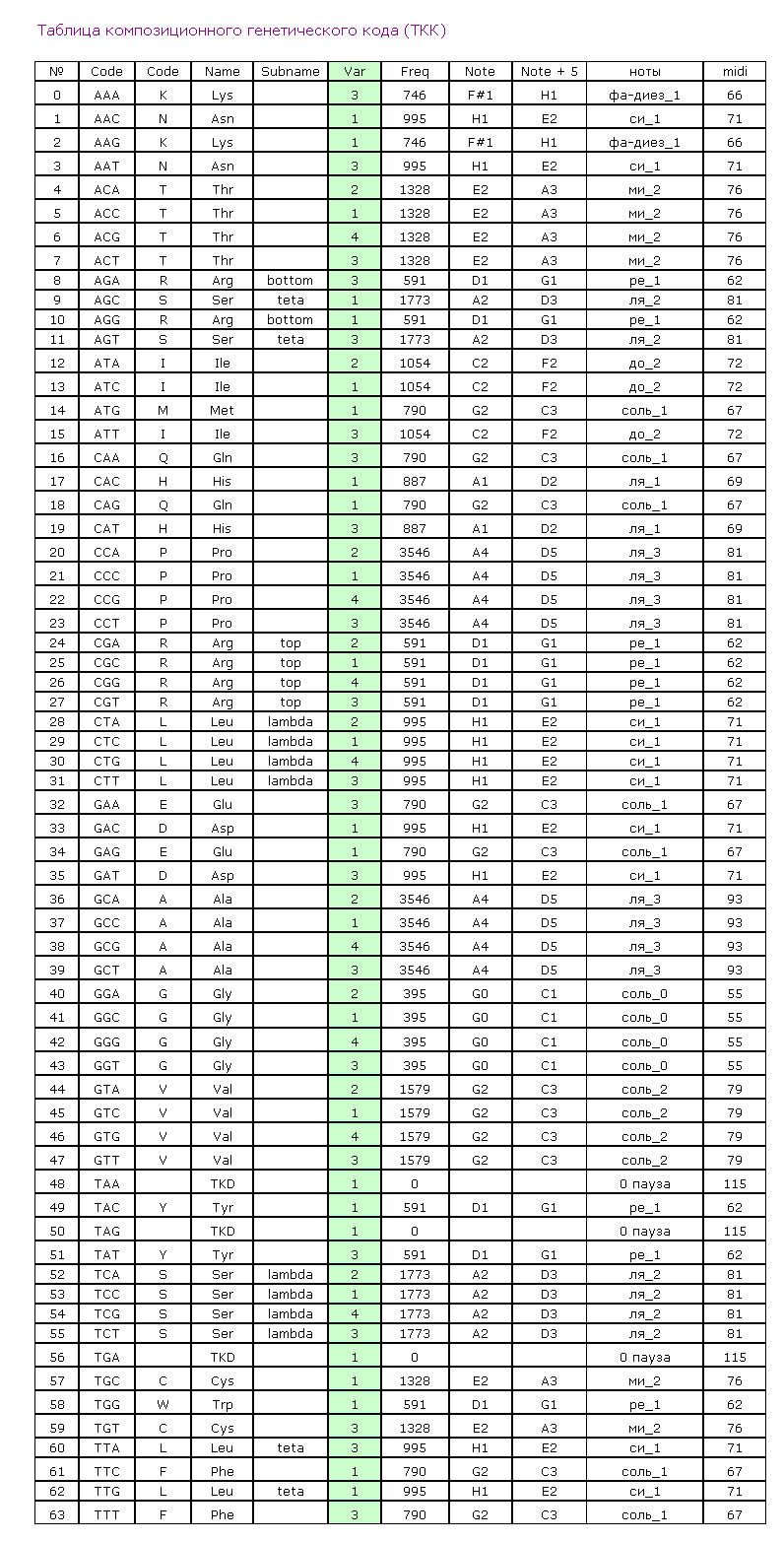
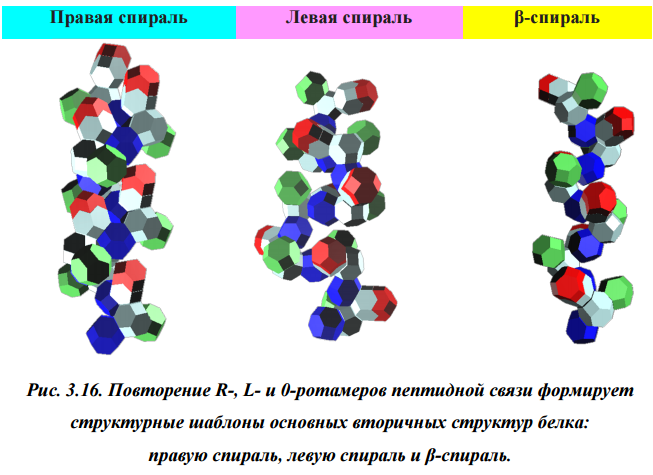
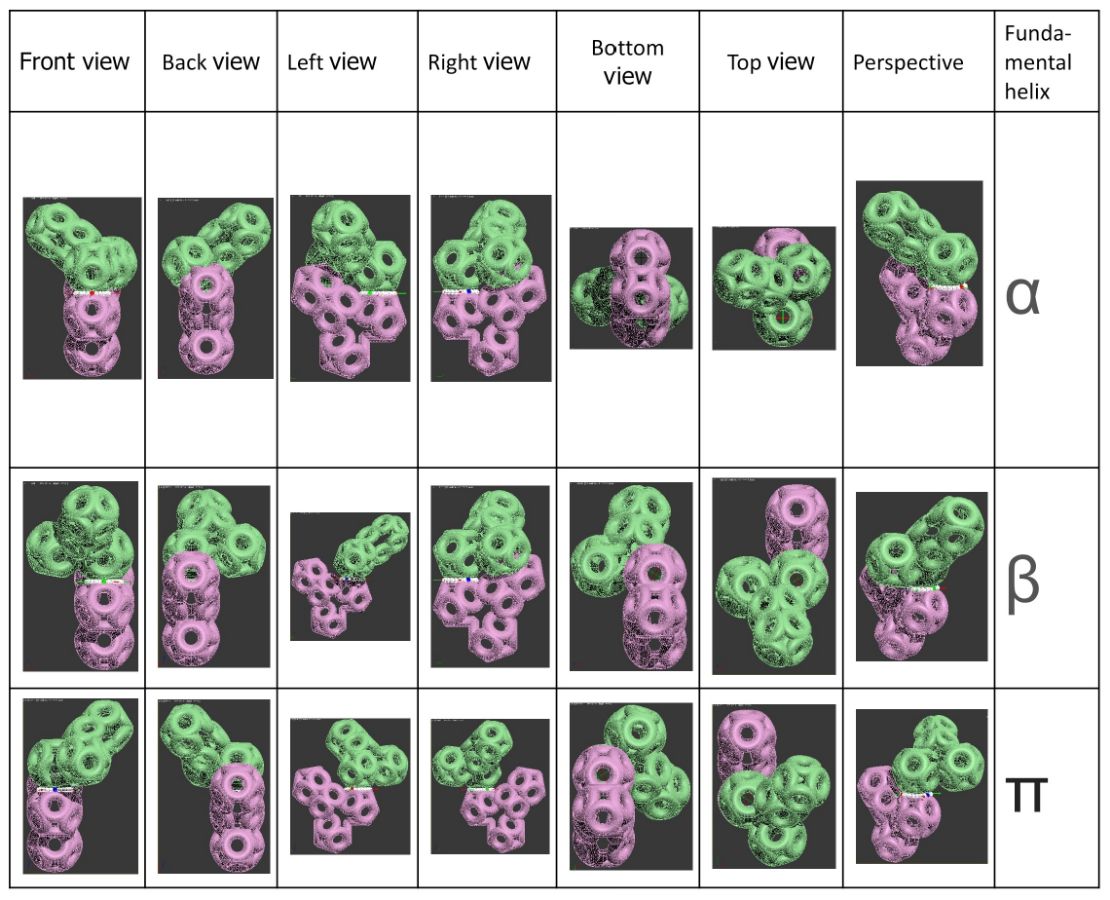
БЕЛКОВЫЕ СПИРАЛИ
 |
 |
 |
 |
 |
| Альфа-спираль |
Бета-спираль |
Пи-спираль |
310-спираль |
Метиониновая спираль |
3D СТРУКТУРЫ АМИНОКИСЛОТНЫХ ОСТАТКОВ
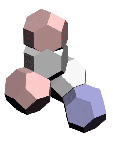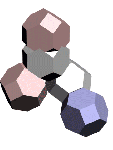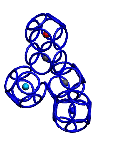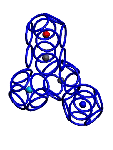
Суставы Pro и Met
Pro - это иминокислотный остаток, структура которого гнется не только в плоскости пептидной группы (транспозиционный угол), но и в другой плоскости (пролиновый угол).
Для точного пространственного моделирования программа, написанная по упрощённому алгоритму подходит только для узкого класса белков типа лизоцима, где нет ни аминокислотных остатков Pro, ни Met.
Три угловых фрактальных шкалы показывают композиционный, транспозиционный и пролиновый углы соответственно по возрастанию размеров шариков.
Транспозиционный угол откладывается по шкале, которая лежит в перпендикулярной плоскости к шкале композиционных углов.
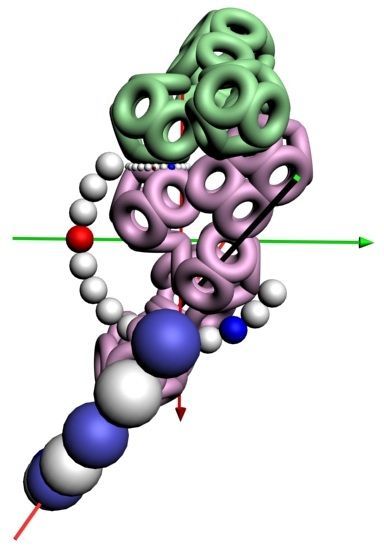
Изгиб на сустве Pro, модель
Содержание 2D диаграмм Пикотех
2D структуры Пикотехнологии отображают, по какому пространственному алгоритму цифровой / цветовой композиционный код белка детерминирует 3D структуру молекулы


СОКРАЩЁННАЯ 2D ДИАГРАММА
Одна клеточка - один аминокислотный остаток
Согласно Таблице композиционного генетического кода,
- виток альфа-спирали - 4 аминокислотных остатка с кодом "1",
- виток бета-спирали - 3 аминокислотных остатка с кодом "2",
- виток 310-спирали - 3 аминокислотных остатка с кодом "4",
- виток пи-спирали - 5 аминокислотных остатков с кодом "3"

БЕЛОК AAS15574
Шарики обозначают аминокисльтные остатки. Цвета соответствуют Композиционноу (3D) генетическому коду:
Альфа спираль или одиночный альфа-код - красный
Бета спираль или одиночный бета-код - зелёный
Пи спираль или одиночный пи-код - синий
310-код - оранжевый
Код метионина в составе альфа-310 спирали - сиреневый
КРАТКАЯ 2D диаграмма ПИКОТЕХ
Красный - альфа-спираль.
Оранжевый - 310-спираль.
Синий - пи-спираль.
Розовый - одиночный код альфа/310 спиралей.
Бирюзовый - одиночный код пи-спирали.
Тёмно-зеленый - бета-спираль.
Зелёный - одиночный код бета-спирали
Сиреневый - метиониновая спираль. У неё более крупный шаг "резьбы", чем у обычной альфа-спирали.
Жёлтый - сустав пролина
Черный в сокращённом представлении и белый в развернутом означают либо неизвестный код, либо конец трансляции.
Циклическое повторение цветов (композиционных кодов) - программная спираль.
Программные спирали - повторение последовательности композиций. Например, один код альфа-спирали, затем один код пи-спирали. n(35) задаёт программную спираль, а n3 или n5 - простые спирали (пи-спираль и альфа-310-спираль).
232323 - программная n(23)-спираль
141414 - программная n(14)-спираль

ПРОГРАММНЫЕ СПИРАЛИ ИЛИ СВЕРХВТОРИЧНЫЕ СТРУКТУРЫ БЕЛКОВ
Кушелев: В структуре белка используется ряд подструктур, похожих на базовые, но построенных по микропрограммам. Это позволяет уточнить интерпретацию экспериментальных данных из Protein Data Base, а в ряде случаев заменить неправильную интерпретацию на правильную.
3212-спираль, в которой регулярно повторяется фрагмент композиционного кода 3212, напоминает бета-спираль, только имеет пятиричную квазисимметрию, как и семеричная спираль. Сбоку видны характерные ~прямые углы, как и в бета-спирали.

Любопытный повтор композиционного кода: 23323323. Поворот до совмещения с соседним аминокислотным остатком происходит на 45 градусов, значит спираль имеет 8-ричную квазисимметрию.

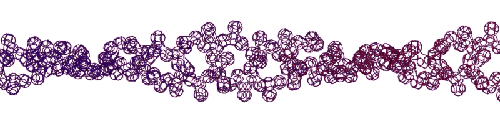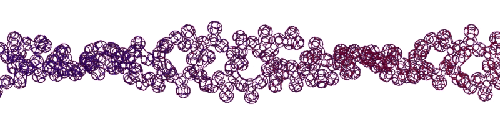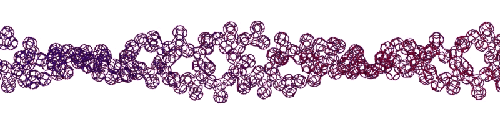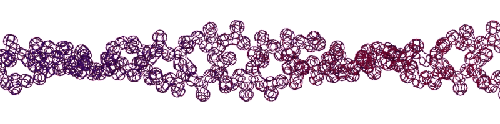
Эта конструкция напоминает складчатый бета-слой. Композиционный код: 13323133231
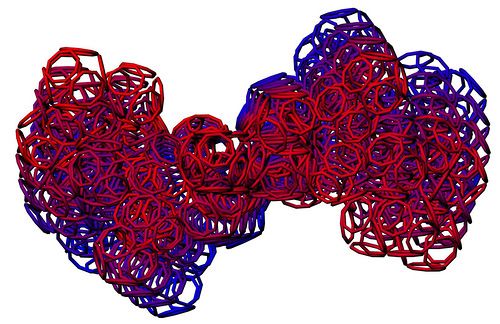
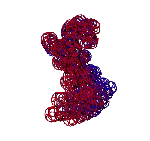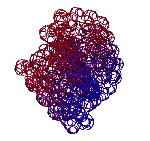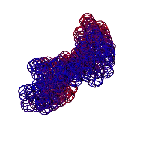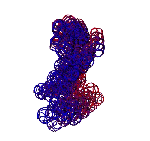

ПОЛНАЯ 2D диаграмма ПИКОТЕХ
Содержание столбцов
1 - порядковый номер аминокислотного остатка в белковой молекуле
2 - триплетный код
3 - однобуквенный код аминокислотного остатка
4 - трёхбуквенное обозначение аминокислотного остатка
5 - упрощённый композиционный код
6 - графическая интерпретация упрощенного композиционного кода
7 - композиционный код
8 - графическая интерпретация композиционного кода
9 - нота, которая звучит при установке данной аминокислоты в растущую белковую цепь
10 - графическое изображение ноты (или ударного инструмента)
Для версии Пикотех 2017 разработан Композиционный код 6var
Для версии Пикотех 2018 разработан Композиционный код 7var и 8var
Для версии 2019 разработан Композиционный код 9var
УСЛОВНЫЕ ОБОЗНАЧЕНИЯ
34343(Q-спираль) и 14141(QVQVQ-спираль)
в Thioalkalivibrio nitratireducens DSM 14787 protein
http://www.ebi.ac.uk/ena/data/view/AGA34115&display=text
>ENA|AGA34115|AGA34115.2 Thioalkalivibrio nitratireducens DSM 14787 hypothetical protein
TTGACTCGGGCTTGGACTCGGGCTCGGGTTCCGGCTCAGGCTCGGGTTCCGGCTCAGGCT
CGGGTTCCGGCTCAGGCTTGGGTTCCGGCTCAGGCTCGGGTTCCGGCTCGGGTTCCGGCT
CGGGTTCCGGCTCGGGTTCCGGCTCGGGTTCCGCGGCTCGGGCTCCGGCTCCGGCTCCGG
CTCCGGCTCCGGCTTCGGCTCGGGTTCCGGCTCGGGTTCCGGCTCGGGTTCCGGCTCGGG
TTCCGGCTCGGGCTCCGGCTCCGGCTCGGGTTCCGGCTCGGGTTCCGGCTCGGAACCGAG
TTGTGCGTTATCGATTTCGTCAGTGGTCGGCAGATCCTCGGGAGGAGCCGGGGGAAGCGT
CAGGTCCCCCAGCTCGATCCAGCTTGCCTGCATGCCCCCGCGTCCGAGATCGAGGGTCGC
CTGGCTCGCCTGCCAAACGACCAGACCGCCCACCGCGGCCCCGTGCAACGCGAGGCTCGC
AAGAATCCCGAGCGCTAG

Locus AGA34115
FT CDS 1..498
ttgactcggg cttggactcg ggctcgggtt ccggctcagg ctcgggttcc ggctcaggct
cgggttccgg ctcaggcttg ggttccggct caggctcggg ttccggctcg ggttccggct
cgggttccgg ctcgggttcc ggctcgggtt ccgcggctcg ggctccggct ccggctccgg
ctccggctcc ggcttcggct cgggttccgg ctcgggttcc ggctcgggtt ccggctcggg
ttccggctcg ggctccggct ccggctcggg ttccggctcg ggttccggct cggaaccgag
ttgtgcgtta tcgatttcgt cagtggtcgg cagatcctcg ggaggagccg ggggaagcgt
caggtccccc agctcgatcc agcttgcctg catgcccccg cgtccgagat cgagggtcgc
ctggctcgcc tgccaaacga ccagaccgcc caccgcggcc ccgtgcaacg cgaggctcgc
aagaatcccg agcgctag
//


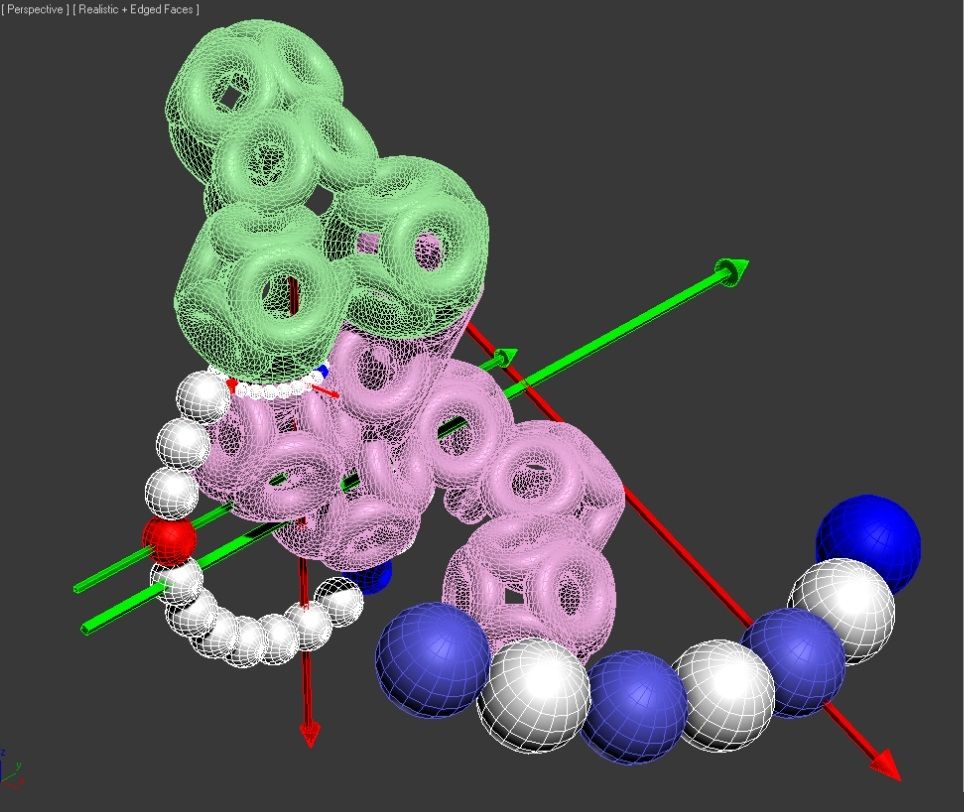
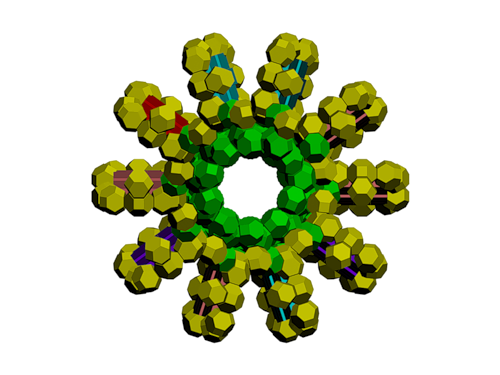
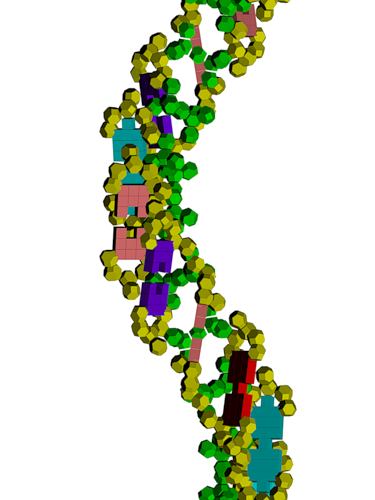
Модель ДНК
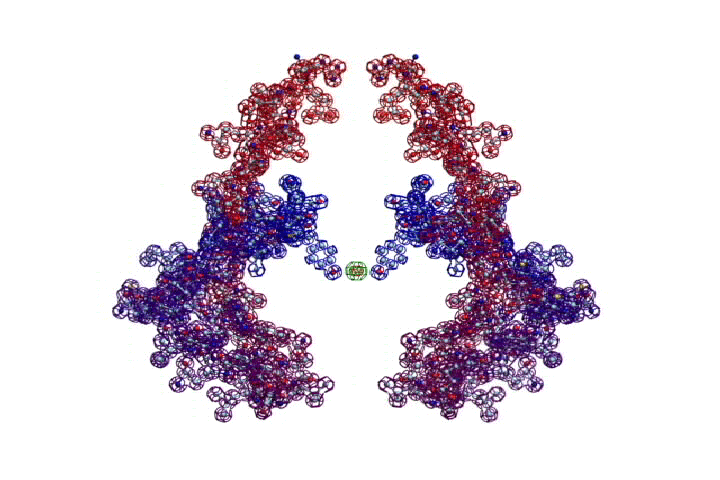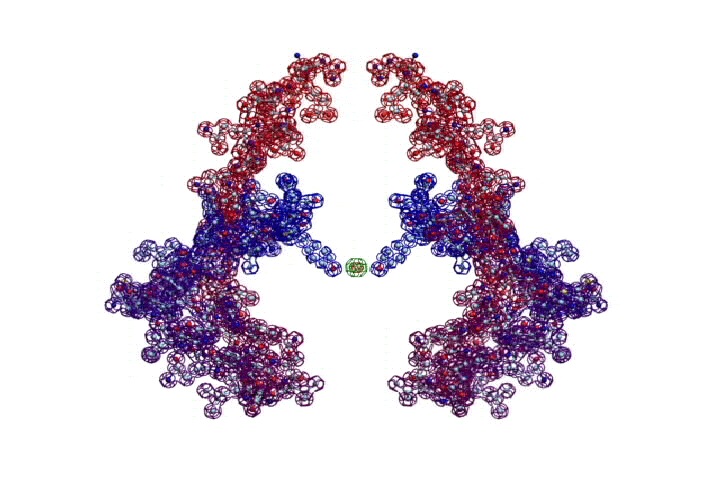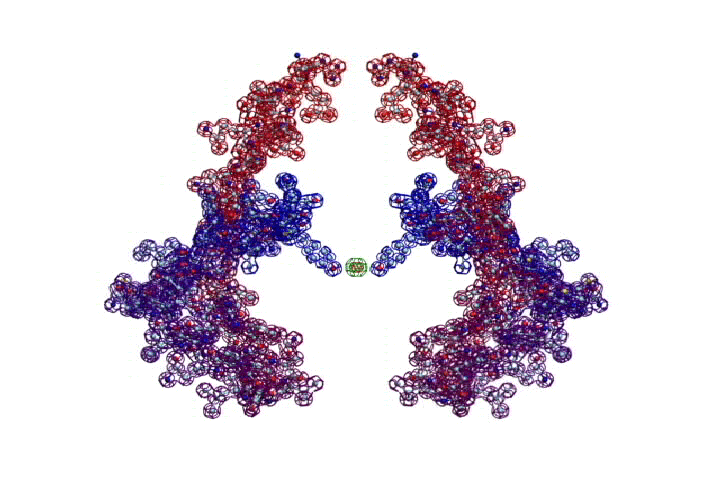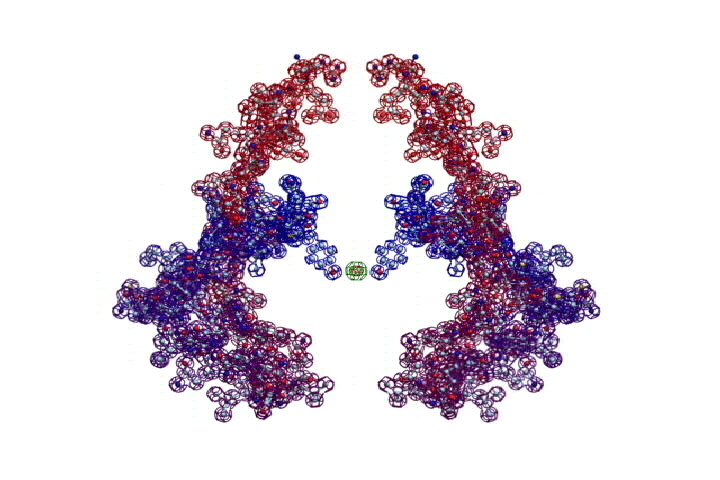
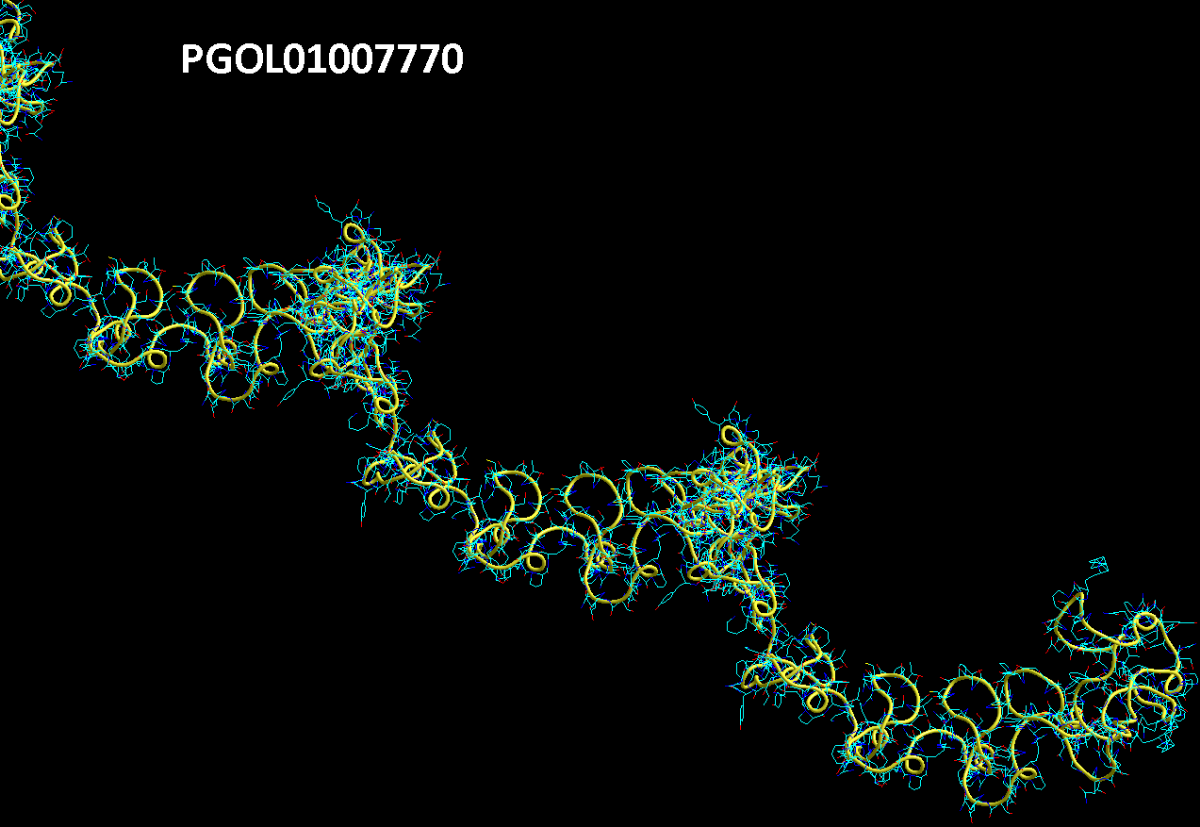
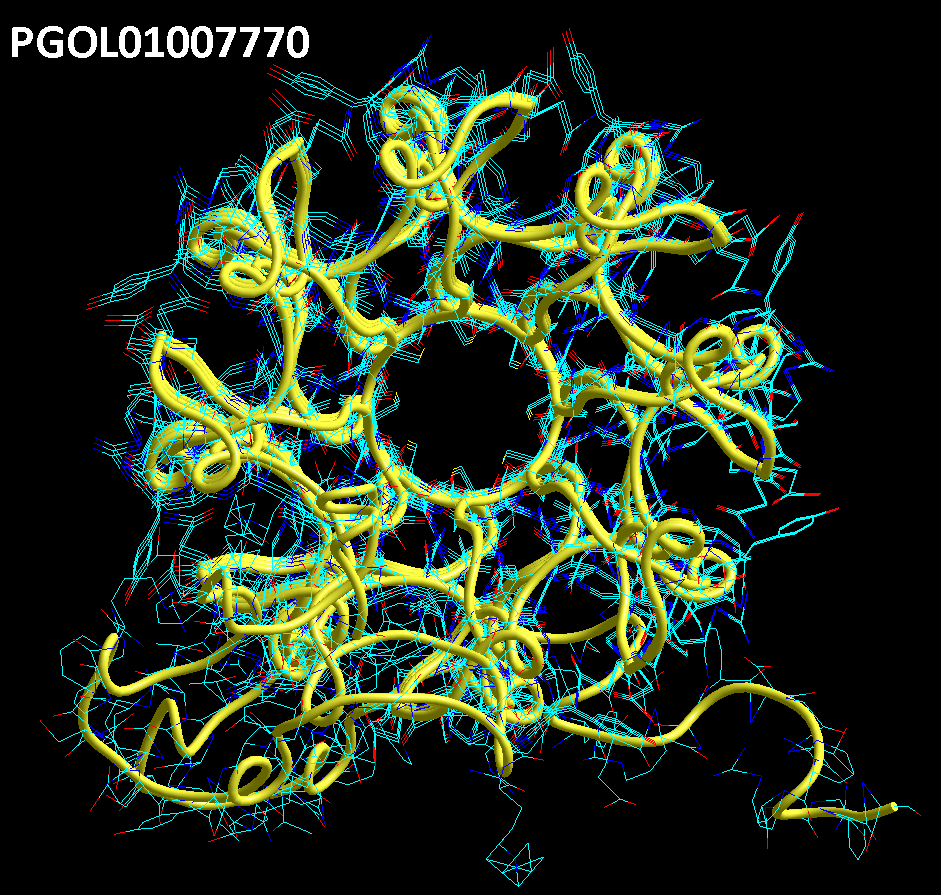
БЕЛОК, ИМЕЮЩИЙ ФОРМУ И РАЗМЕРЫ 25 СТУПЕНЕЙ ДНК
Случайное совпадение формы моедли белка и ДНК исключено. Совпадают внутренний и внешний радиус, шаг спирали,количество ступеней на виток.
>PGOL01007770.1:c3269-1 Punica granatum strain AG2017 PG007770, whole genome shotgun sequence
atccaaggatgggactcggcctggggaagatctaacgatgggattcggcccaacgaaaga
tcaaaggttgggactcggcccagggggagatctaacgatgggactcagccctacggaaga
tcaaaggttgggactcgtccctgggggagatctaacgatgcgactcggccctacgaaagt
tcaaaggatgggactcggccctgggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactccgccctgtgggagatctaacgatgggactcgaccctacggaaga
tcaaaggttgggactcagcccagggggagatctaacgatgggactcggccctacggaaga
tcaaaggttgggactcggccctgtgggagatctaacgatgcgactcggccctacggaaga
tcaaaggttgggactccgcccagggggagatctaacggtgggactcggccctacggaaga
tcaaaggttgggactcggctctgggggagatctaacgatgagactcggccctacgaaaga
tcaaaggatgggactcggcccagggggagatctaa

Развернуть: https://yadi.sk/i/K9jwT9lakQ0zAA
Слушать(MIDI): https://yadi.sk/d/UIhJRsRH669gyw
Программная спираль строится в 40-дольном музыкальном размере.
Структуры белков такого типа могут наглядно продемонстрировать триумф пикотехнологии белков.



Полновесная анимация (avi, 63Mb): https://yadi.sk/i/cdixlJKwoI9Eyw
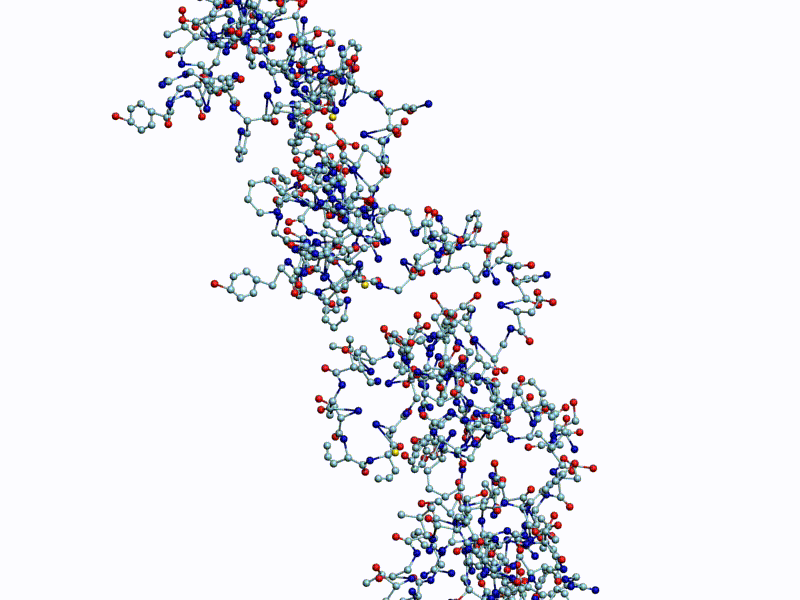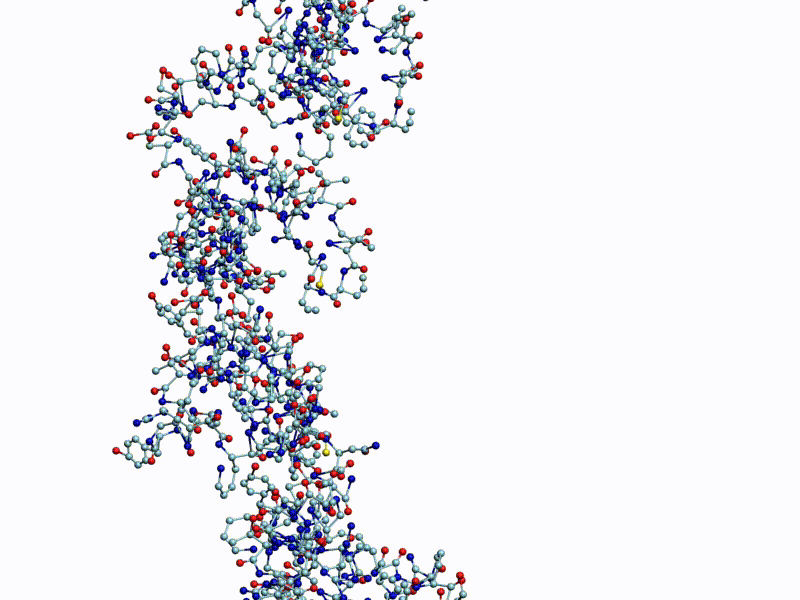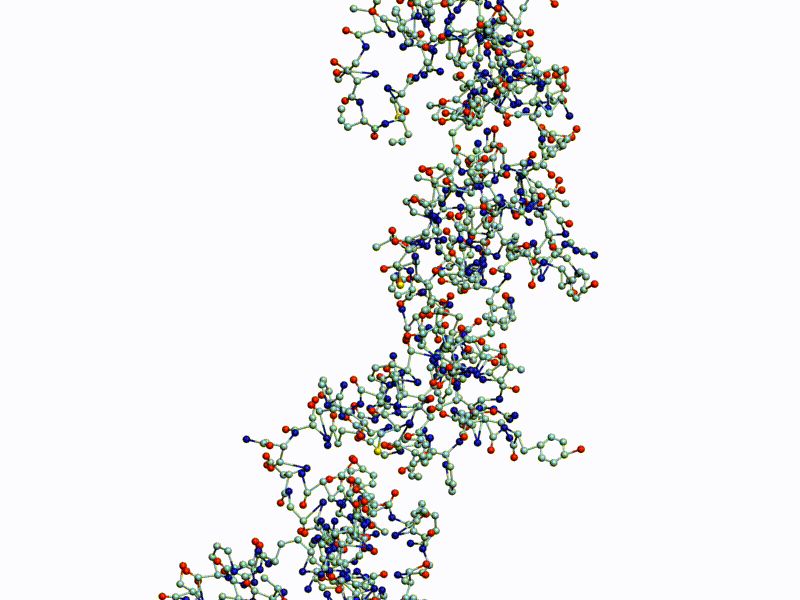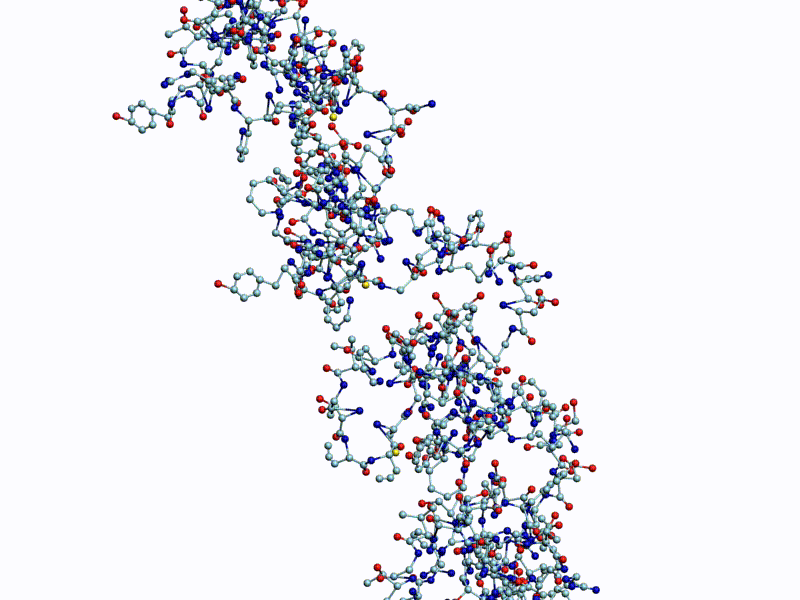
Полновесная анимация (avi, 27Mb): https://yadi.sk/i/g9qzgoDeH12mMA
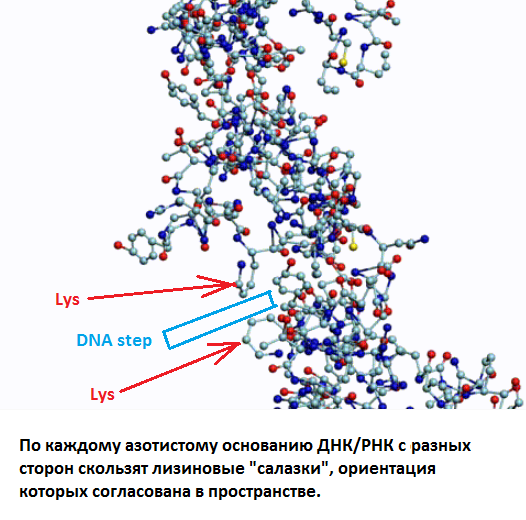
Обратите внимание на пары радикалов лизина (Lys).
По каждому азтистому основанию ДНК/РНК сользят лизиновые "салазки", ориентация которых согласована в пространстве.
Композиционный генетический код 4var, 6var, 7var, 8var и 9var
В Диграммах Пикотех применяются три вида композиционного генетического кода – коды 4var, 6var и 7var.
Код "2" (спираль - не спираль).
Код 4var более подробный (альфа-, бета-, пи-, 310-).
Код 6var ещё более подробный. В нём есть различие "одиночный альфа-код", "код альфа-спирали", "код 310-спирали", "Код метионина в составе спирали".
Код 7var различает одиночный код пи-спирали от кода в составе пи-спирали .
Код 8var различает одиночный код бета-спирали
Код 9var различает пролин в конце спирали
В настоящее время считается, что белок в неопознанной части структур, полученных методом РСА, не имеет определенной структуры, но пикотехнология показала, что это не так. В программе "Пикотех" вся структура определена.
Пикотехнология не может определить структуры, полученные не по программе рибосомой. Если после сборки белка он обработан ферментами, например, разрезан и переделан, то программа "Пикотех" об этом не даёт информации.
Построение 2D Диаграмм и структур 3D Пикотех
Алгоритм построения Пикотехнологических моделей белков основан на открытии Композичионного генетического кода, см. монографию А.КУшелева и В.Соколик "Геометрия живого наномира. Пикотехнология белков".
Структуры 2D Пикотех строятся с достоверностью 100% в автоматическом режиме. Структуры 3D Пикотех в зависимости от состава молекул строятся в автоматическом режиме (геометрический алгоритм) либо в ручном режиме (с учётом физико-химических взаимодействий и свойств сустава Pro). Метод построения структур 3D Пикотех на базе 2D структур Пикотех является ноу-хау Лабораторатории Наномир.
Пять надежных методов проверки Пикотехнологии
Первый - достоверная корреляция с данными РСА. Конечно, РСА не даёт высокой достоверности, но она часто выше 50%.
На таких белках, как инсулин, гемоглобин, окситоцин корреляция приближается к 100%, т.к. в окситоцине, например, есть замкнутые циклы через дисульфидные мостики. Это позволяет проверить и РСА, и Пикотех.
Второй - замкнутые через дисульфидные мостики циклы типа окситоциновых. Вероятность случайного замыкания шести циклов, насчитывающих десятки аминокислотных остатков из разных окситоцинов, порядка 1/30 000 000 000. Речь идёт о вероятности для каждого цикла - все шесть могут случайно замкнуться с вероятностью (1/30 000 000 000)^6.
Третий - самоповерка на примере сверхдлинных базовых и программных спиралей, где наблюдается 100%-ная корреляция между вторичной и первичной структурами.
Четвёртый - микроскопия. 3D модель "монстра" первой хромосомы хороша тем, что её можно проверить, рассматривая молекулу белка в флуоресцентный микроскоп. "Монстр" первой хромосомы такой крупный, что его видно в микроскоп в отличие от мелких белков. В частности, диаметр начальной фрактальной петли (правая часть) примерно 100 нанометров, т.е. 0.1 микрона.
Пятый - сопоставлени модельных экспериментов Пикотех и натурных экспериментов.
Модельные эксперименты с помощью 3D принтера показыват, что набором из 9 вариантов по три угла (композиционный, транспоцизионный и пролиновый) можно описать только часть белковых структур, например, прямой участок альфа-, 310-, пи-, бета-спиралей, произвольную последовательность композиционных (3D) кодов в том случае, если нет ограничений по водородным, вандерваальсовым и гуковским связям. В реальных белках, которые часто содержат программные спирали, такие ограничения есть, поэтому набором из 9 вариантов по 3 угла большинство белковых структур описать не удастся. Без учета физики, т.е. на уровне геометрического алгоритма. Учесть физику трудоёмко, но можно добавить базу данных для программных спиралей, что сильно расширит возможности геометрического алгоритма. И хотя программных спиралей великое множество, но ходовых программных спиралей не так уж много, а редкие программные спирали придётся исследовать с учетом физики. Пока в ручном режиме. Наиболее актуальной является интерактивная 3D-версия, где можно визуально наблюдать за появлением различных ограничений в процессе построения модели белка.
Модель девиации композиционного угла

Фрактальная программная спираль первой хромосомы (Нейробластома)






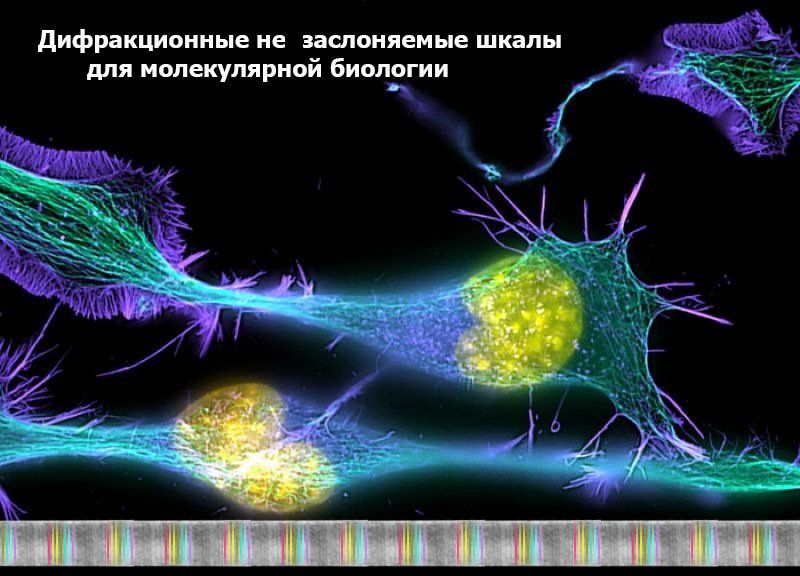
ДИФРАКЦИОННЫЕ НЕЗАСЛОНЯЕМЫЕ ШКАЛЫ ДЛЯ МОЛЕКУЛЯРНОЙ БИОЛОГИИ
Эффект обратной дифракции делает возможным создание незаслоняемой дифракционной шкалы на основе органических молекул, в частности, белковых. Это позволит произвести "геодезическую разметку" в микромире и точно измерить его объекты.
ПИКОТЕХНОЛОГИЯ КАК ДОПОЛНЕНИЕ К РЕНТГЕНО-СТРУКТУРНОМУ АНАЛИЗУ
Программа Пикотех не может полностью заменить РСА.
Однако она очень усиливает самые слабые стороны РСА, а именно достоверно показывает вторичную структуру и ближний порядок расположения атомов третичной структуры в момент сборки белковой молекулы рибосомой.
Чувствительность РСА такова, что он не замечает не только отдельных атомов, но и отдельных аминокислотных остатков. Более того, "хвосты" белковых молекул, которые не кристаллизуются, РСА вообще "не видит", а эти "хвосты" могут насчитывать до 50 аминокислотных остатков.
Особый класс образуют 97% белковых молекул, которые не кристаллизуются. Про них РСА просто ничего не знает, а программа Пикотех так же достоверно показывает их вторичную структуру и ближний порядок расположения атомов в третичной структуре.
КОЛЛАГЕН - НЕ ТРОЙНАЯ, А ПРОГРАММНАЯ 335-СПИРАЛЬ
Модель коллагена (программная 335-спираль) - яркая демонстрация несовершенства рентгеноструктурного анализа. Специалисты по РСА конструируют спираль коллагена из того, что им известно. А известна им альфа-спираль. Вот они и сконструировали модель из 3 альфа-спиралей. Программа Пикотех показывает, что это не тройная, а одинарная, но программная 335-спираль. Радикалы аминокислот действительно располагаются в виде трёх-заходной спирали, что и сбило с толку специалистов по РСА.
Другие программные спирали специалисты по РСА тоже ошибочно интерпретируют известными им "базовыми спиралями", но настало время показать, как устроены реальные, программные спирали белков.
Другими яркими примерами, демонстрирующими недостаточную информативность РСА, являются сверхдлинные, в том числе программные спирали.
Наконец, Пикотехнология в отличие от РСА имеет возможность самоповерки. В частности, самоповеркой является 100%-ная корреляция между первичной и вторичной структурой некоторых белков, например, сверхдлинных программных спиралей. Спирали длиной более 1000 витков исключают случайные совпадения, согласитесь. Например, вероятность случайного совпадения композиционных кодов для 310-спирали длиной более 3982 аминокислотных остатков не превышает 4^-3982=2.5*10^-2397, т.е. более двух тысяч нулей после запятой.
Подробнее об этом белке можно прочесть здесь.
https://www.ncbi.nlm.nih.gov/nuccore/902935724
>ENA|XP_013055863|102 nucleotides
cctggtccccctggtccccctggtccccctggtccccctggtccccctggtccccctggt
ccccctggtccccctggtccccctggtccccctggtccccct


ФРАКТАЛЬНАЯ ПРОГРАММНАЯ 257575-СПИРАЛЬ
https://www.ncbi.nlm.nih.gov/nuccore/523901990
Понятно, что здесь должны быть другими как минимум транспозиционные углы. Очевидно, что витки программной спирали должны взаимодействовать друг с другом, образуя более прочную фрактальную структуру. Дальний порядок восстановить ещё труднее. Видно, что полувитки программной 57-спирали поворачиваются на кодах "2" примерно на прямой угол, а значит участки программной спирали могут образовать реберный многогранник или аналогичную реберную конструкцию. Остатков Pro в структуре не встречается, поэтому скорее всего это - жесткая конструкция, хотя за счет транспозиционных связей может где-то и гнуться.

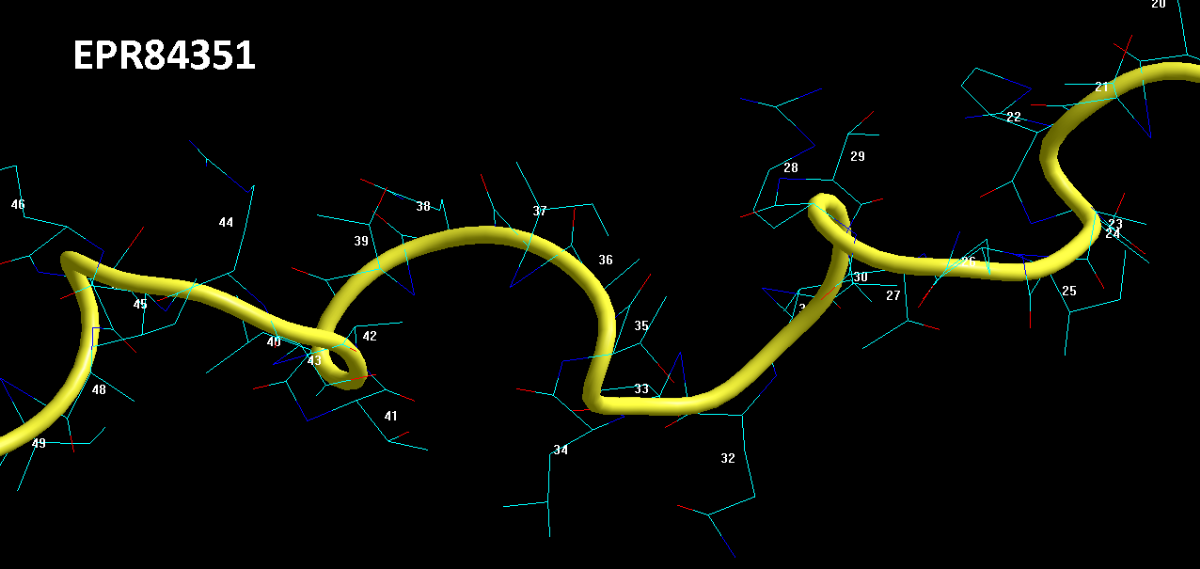
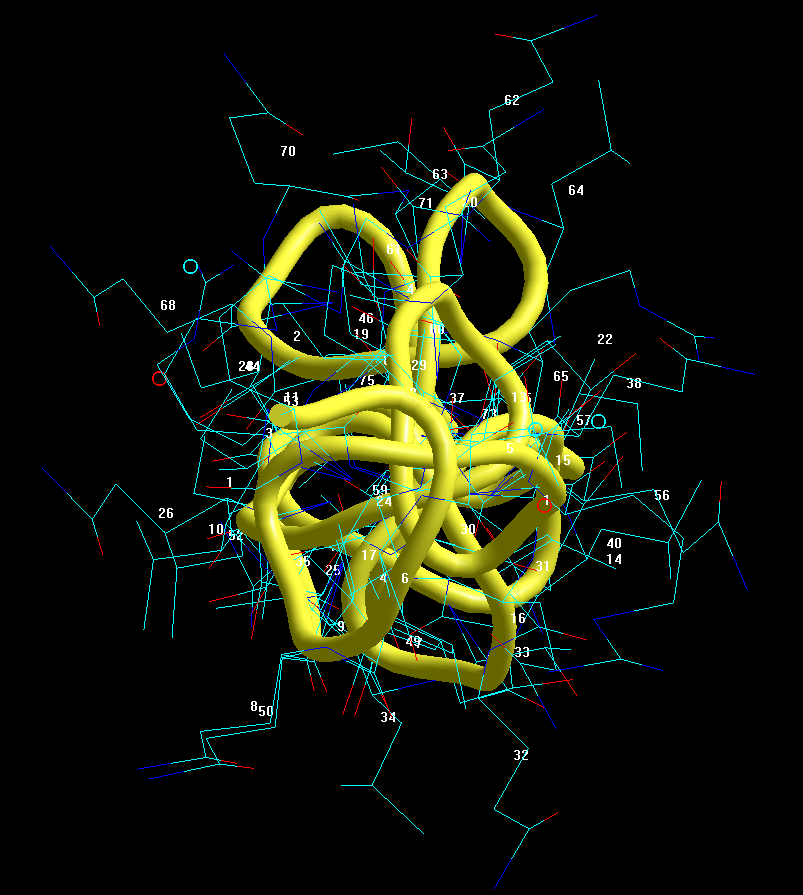
РЕНТГЕНО-СТРУКТУРНЫЙ АНАЛИЗ НЕ РАЗЛИЧАЕТ АЛЬФА-СПИРАЛИ И ПИ-СПИРАЛИ, А ТАКЖЕ ДРУГИЕ ДЕТАЛИ
Aequorea victoria isolate sGFP-206 green fluorescent protein (gfp) gene
https://www.ncbi.nlm.nih.gov/nuccore/JX472995.1?report=fasta
ДАННЫЕ 2D ДИАГРАММЫ ПИКОТЕХ
2D диаграмма Пикотех показывает, что в структуре есть 4 пи-спиральных (синий) и 2 альфа-спиральных (красный) участка. Есть ещё и виток 310-спирали (оранжевый), но такие "мелочи" РСА не замечает .
Бета-спиралей и тем более слоёв из бета-спиралей в белке GFP нет вообще. Это - ошибки РСА.

ИНТЕРПРЕТАЦИЯ РЕНТГЕНО-СРУКТУРНОГО АНАЛИЗА
Вторичная структура из пяти α-спиралей и одиннадцатb слоёв β-спирали.... Разве ?
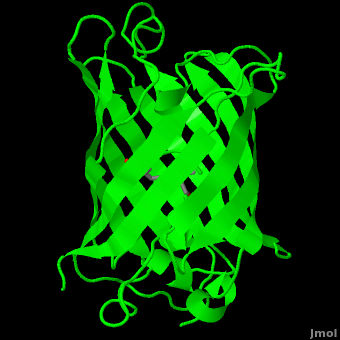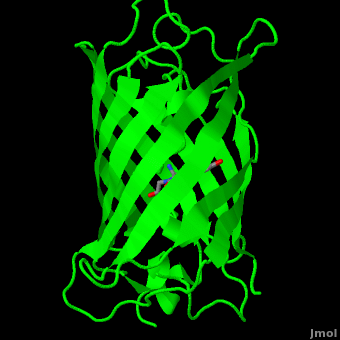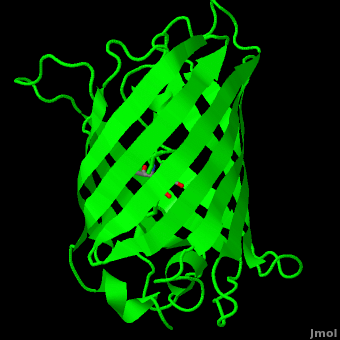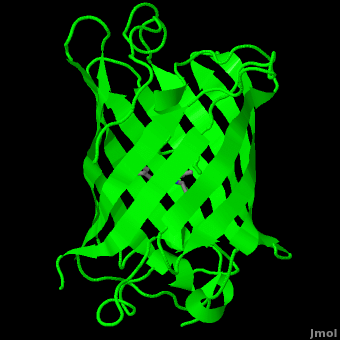


Human herpesvirus 3 strain Dumas transcriptional regulator
УВЕЛИЧИТЬ
ПИКОТЕХНОЛОГИЧЕСКАЯ МОДЕЛЬ ГИСТОННОГО КОМПЛЕКСА

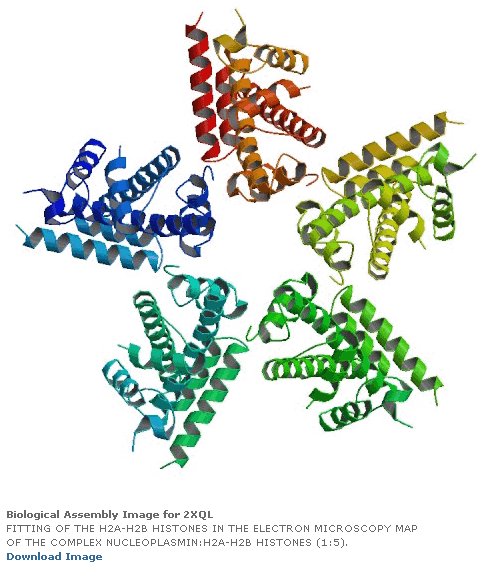
ПИКОТЕХНОЛОГИЧЕСКАЯ МОДЕЛЬ КОЛЛАГЕНА
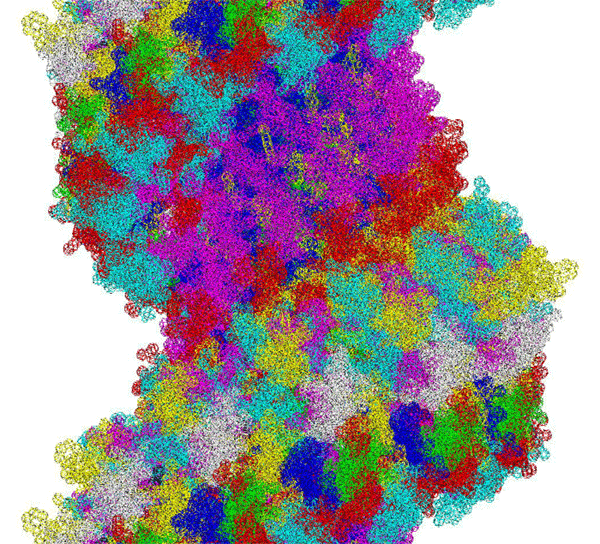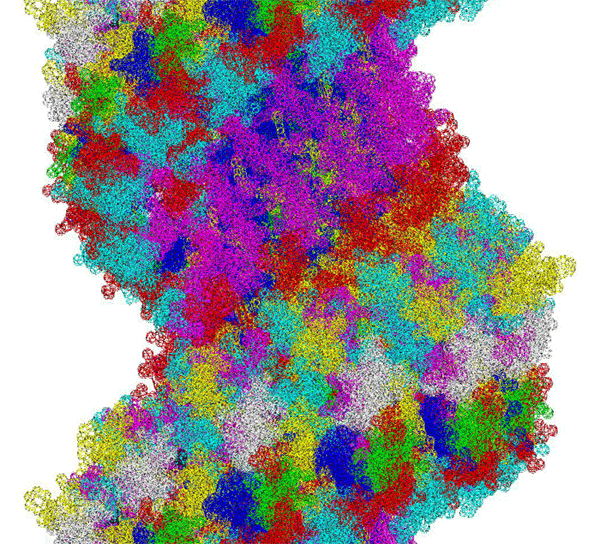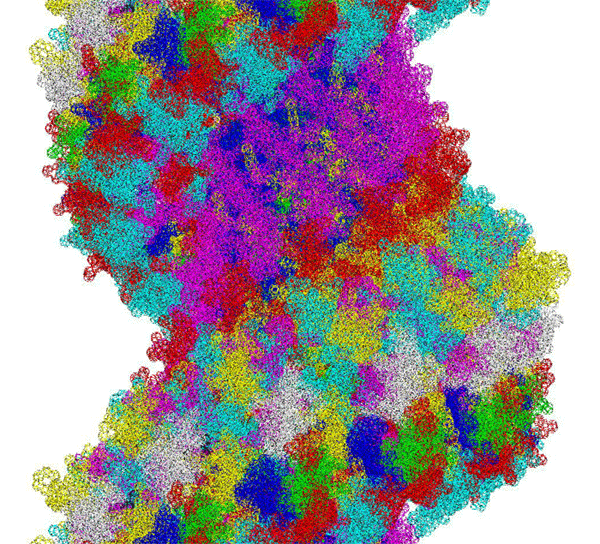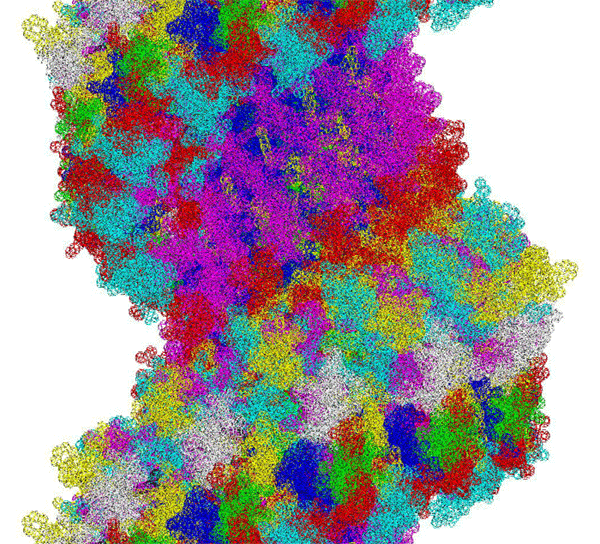

ПИКОТЕХНОЛОГИЧЕСКАЯ МОДЕЛЬ МОЛЕКУЛЫ ИНСУЛИНА
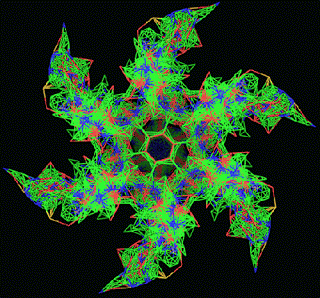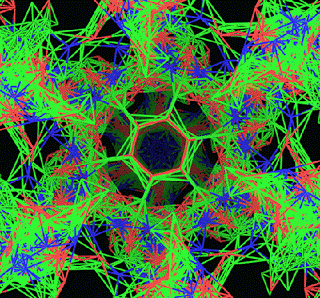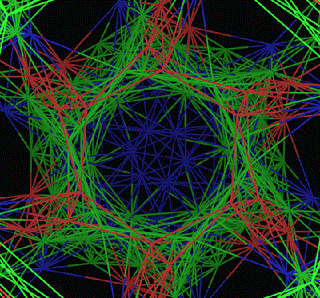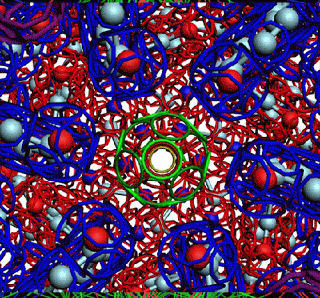

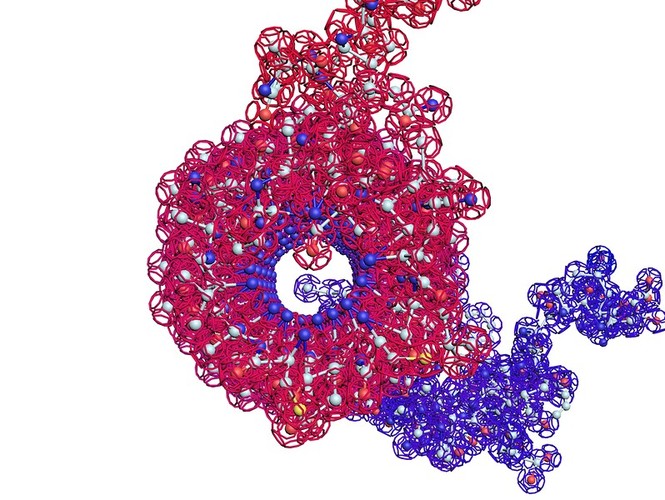
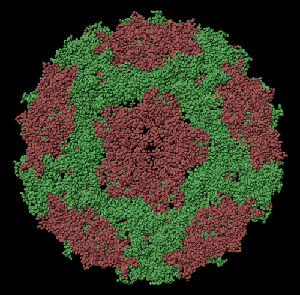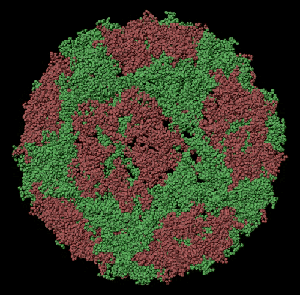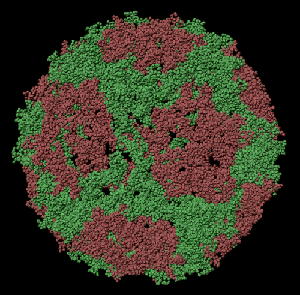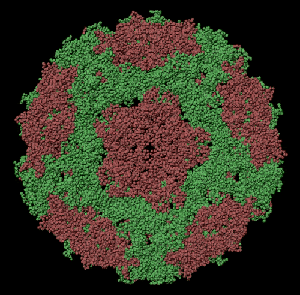
ПИКОТЕХНОЛОГИЧЕСКАЯ МОДЕЛЬ ОБОЛОЧКИ ВИРУСА

Литература
1.Монография А.Кушелев, В.Соколик "Геометрия живого наномира. Пикотехнология белков" https://www.morebooks.de/ru/search?utf8 … 0%BE%D0%B2
Предисловие рецензентов и авторов
Ознакомиться с началом монографии можно по ссылке
2. Кушелев А.Ю., Соколик В.В. Пикотехнология – новый подход в моделировании пространственной структуры белка / Заочная Международная научно-практическая конференция «Современная наука: тенденции развития» (24 января 2012), Краснодар: НИЦ Априори. – 2012. – С.203-207.
3. Соколик В.В. Предсказание пространственной структуры белка insilico на основе информации генома и геометрического алгоритма – альтернатива квантово-механическому подходу // Материалы Международной научной конференции «Математическое и компьютерное моделирование в биологии и химии. Перспективы развития» (28-30 мая 2012), Казань. – 2012. – С.155-158.
4. Sokolik V.V. Protein is coded in genome and synthesized in ribosomes as a structural template of a rotameric version sequence of peptide bound configuration // The International Moscow Conference on Computational Molecular Biology, МССМВ-11, Moscow. – 2011. – P. 347–348.
5. Sokolik V.V. Algorithm of protein structural template decoding according to its determined nucleotide sequence // Fist International Conference “Fundamental medicine: From scalpel toward Genome, Proteome and Lipidome”, Pax Grid Virtual Conferences, Kazan. – 2011. – P. 117–119.
6. Sokolik V.V. Modeling of the individual structural template of protein on determining it nucleotide sequences // VII Международная конференция по биоинформатике, регуляции структуры геномов и системной биологии. BGRS\SB-2010, Новосибирск. – 2010. – С. 275.
7. Соколик В.В. Способ моделирования пространственной структуры белка по детерминирующей его нуклеотидной последовательности // Биофизический вестник. – 2010. – Вып. 24 (1). – С. 31-45.