Виктория Соколик: 6-10 октября сего года в Киеве проходил очередной конгресс украинского биохимического общества, на котором был представлен мой доклад по пикотехнологии белков "Информация о структуре белка содержится в геноме".
Эта информация тезисно опубликована в материалах конгресса:
В.В. Соколик "Никакой дополнительной информации, большей, чем та, что содержится в ДНК, для сворачивания белка не требуется" Матеріали ХІ Укріїнського біохімічного конгресу (06-10 жовтня 2014), The Ukrainian Biochemical Journal, 2014, V. 86, №5 (Supplement 1), P. 37-38.
НИКАКОЙ ДОПОЛНИТЕЛЬНОЙ ИНФОРМАЦИИ, БОЛЬШЕЙ, ЧЕМ ТА, ЧТО СОДЕРЖИТСЯ В ДНК, ДЛЯ СВОРАЧИВАНИЯ БЕЛКА НЕ ТРЕБУЕТСЯ
СОЛОЛИК В.В.
ГУ «Институт неврологии, психиатрии и наркологии НАМН Украины», Харьков;
e-mail: Sokolik67@rambler.ru
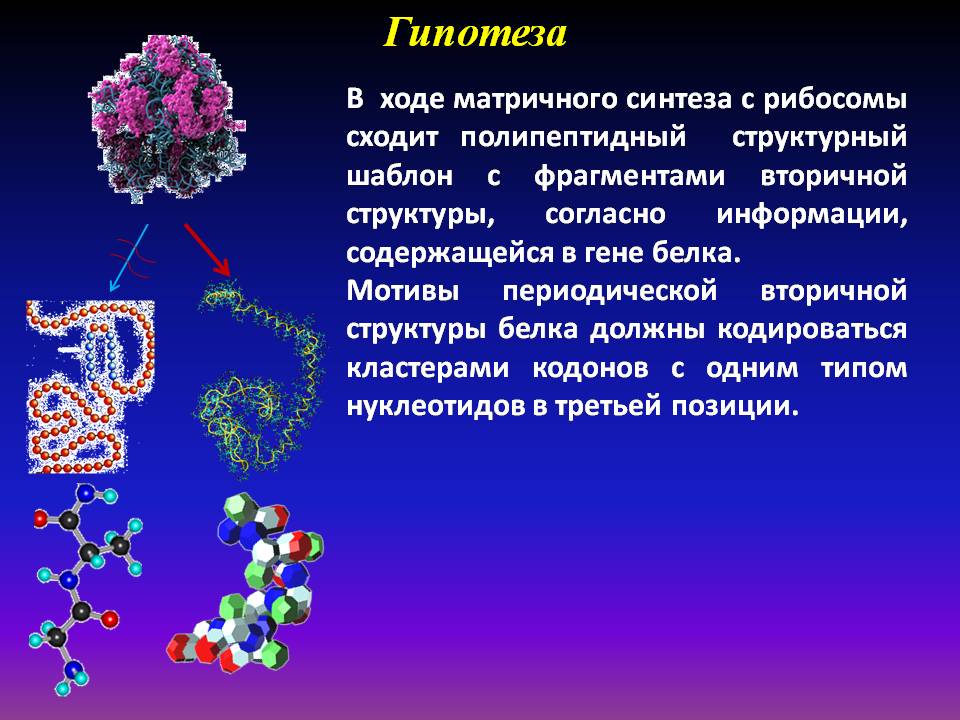
Фолдинг белка причисляют к пулу крупнейших научных проблем современности. Скорость сворачивания белка in vivo объясняют кооперативными эффектами – одновременным формированием «зародышей» вторичной структуры, благодаря чему молекула белка находит «кратчайший путь» на воображаемой гиперплоскости потенциальной энергии к точке, соответствующей нативной конформации. Как правило, реализуется один из трех сценариев: котрансляционное сворачивание новосинтезированного полипептида без шаперонов, с помощью молекулярных шаперонов или сопряженно с трансмембранной транслокацией. Один из подходов для описания рефолдинга денатурированных белков in vitro также постулирует, что сворачивание белка начинается с образования элементов вторичной структуры независимо от третичной или, по крайней мере, до завершения формирования последней. В обоих случаях (in vivo и in vitro) отправным пунктом успешного фолдинга является формирование первичного каркаса с мотивами вторичной структуры белка. Эти данные послужили основанием для предположения о том, что кроме аминокислотной последовательности в генах белков закодирована информация об их индивидуальных структурных шаблонах (каркасах) из фрагментов вторичной структуры. Цель исследования состояла в выявлении специфичности кодирования вторичной структуры в генах белков эукариот.
Дизайн исследования. Проанализировали случайную выборку из 100 белков эукариот, для которых в базе данных PDB представлены схемы вторичной структуры. Оценивали абсолютную и относительную частоту встречаемости кодонов вида XYC/G, XYА и XYТ и их кластеров (XYC/G)n, (XYА)n и (XYТ)n, n?3, в нуклеотидных последовательностях, детерминирующих спиральные фрагменты, ?-тяжи, повороты и неструктурированные мотивы белков. Результаты обрабатывали статистически. t-Критерий Стьюдента использовали для анализа производных показателей (абсолютная и относительная частота), убедившись в нормальности распределения. P-тест Фишера и четырехпольные таблицы – для выявления статистической зависимости между конформерами и разновидностями кодонов и/или их кластеров. Различие считали статистически значимым при p ? 0,05, тенденция к значимым отличиям при p ? 0,10.
Результаты. Установили, что правая спираль кодируется кластерами кодонов (XYC/G)n, n?3 (Р = 7.33?10-22, ?2 = 42.62). Повороты детерминированы кластерами кодонов (XYA)n, n?3. Эти данные согласуются с выявленным достоверным превышением относительной частоты встречаемости кластеров (XYC/G)n, n?3 и (XYA)n, n?3 в областях генов, кодирующих спиральные фрагменты и повороты, соответственно. ?-Тяжи менее жестко детерминированы кодонами XYT. Тест Фишера показал кодирование ?-тяжей кодонами XYT или их кластерами (XYТ)n, n?3: Р = 3.63?10-6 (?2 = 22.05) и Р = 3.43?10-2 (?2 = 4.56) для изучаемого пула белков. Обсуждается роль изоакцепторных тРНК с синонимичными кодонами в перекодировании информации третьего нуклеотида кодонов и их кластеров в ходе матричного синтеза белка. Создана программа Молекулярный конструктор, которая декодирует нуклеотидную последовательность с созданием pdb файла и визуализацией индивидуального структурного шаблона белка в качестве основы для дальнейших исследований методами молекулярной динамики.
Вывод. В геноме эукариот содержится информация о структуре белков.
1–Структура, властивості та функції біологічних макромолекул
Презентация самого доклада: