*
*
Правый столбец 2D Диаграммы Пикотех - нотная запись процесса сборки белковой молекулы
*
Ра
3D генетический код Пикотехнология белков
*
*G
Пространственная структура белка закодирована в его нуклеотидной последовательности
Высоко периодичные структуры белков
96.6 кб, 22 августа в 19:30
Популярные теги Все теги
Сотовая четвертичная структура белка OLQ02947
Вот, что известно о белке, в состав которого входит эта гиперспираль: https://www.uniprot.org/uniprot/Q54KA7
Цитата: Protein: Ankyrin repeat Molecular function: guanyl-nucleotide exchange factor activity
Цитата2: actin cytoskeleton organization
Кушелев: Автоперевод на русский: организация актинового цитоскелета
Ну что же, неплохо для начала. Цитоскелет из трёхгранных призм очень функционален. Теперь слово за структурными биологами, которые знают о структуре этого белка из "мокрых экспериментов".
В реальном белке три слоя, причём состоящих из более тонких слоёв. Первый слой состоит из 4 более тонких. Далее происходит смещение. Под альфа-спиралями (красный) оказываются пи-спирали (голубой). Этот второй слой в свою очередь состоит из 7 более тонких. Далее происходит второе смещение. Под пи-спиралями второго слоя (голубой) оказываются 310-спирали (оранжевый). Этот третий слой состоит из трёх более тонких. В итоге каждая грань каждой треугольной призмы состоит из спиралей трёх типов. Это значит, что параметры граней являются усреднёнными параметрами трёх типов спиралей. Это обусловливает высокую стабильность геометрии. Чем-то напоминает свойство пчелиных сот. Видимо, это очень важно для цитоскелета (скелета клетки) Symbiodinium microadriaticum.
*
*Посмотреть примеры пяти полных 2D Диаграмм Пикотех
В этом фрагменте белка присутствуют две программных спирали.
Программная 1111223553355532114135322332-спираль. Период 28 аминокислотных остатков.

Программная 111135332332-спираль. Период 12 аминокислотных остатков.


*
*
Высоко периодичные структуры белков
Пикотехнологические программные спирали белка
Модель коллагена (программная 335-спираль) - яркая демонстрация несовершенства рентгеноструктурного анализа. Специалисты по РСА конструируют спираль коллагена из того, что им известно. А известна им альфа-спираль. Вот они и сконструировали модель из 3 альфа-спиралей. Программа Пикотех показывает, что это не тройная, а одинарная, но программная 335-спираль. Радикалы аминокислот действительно располагаются в виде трёх-заходной спирали, что и сбило с толку специалистов по РСА.
Другие программные спирали специалисты по РСА тоже ошибочно интерпретируют известными им "базовыми спиралями", но настало время показать, как устроены реальные, программные спирали белков.
Другими яркими примерами, демонстрирующими несостоятельность РСА, являются сверхдлинные, в том числе программные спирали.
Наконец, Пикотехнология в отличие от РСА имеет возможность самоповерки. В частности, самоповеркой является 100%-ная корреляция между первичной и вторичной структурой некоторых белков, например, сверхдлинных программных спиралей. Спирали длиной более 1000 витков исключают случайные совпадения, согласитесь. Например, вероятность случайного совпадения композиционных кодов для 310-спирали длиной более 3982 аминокислотных остатков не превышает 4^-3982=2.5*10^-2397, т.е. более двух тысяч нулей после запятой.
Что такое программа "Пикотехнология белков", и как её грамотно использовать
Пикотехнология помогла найти ошибки рентгеноструктурного анализа

https://www.ncbi.nlm.nih.gov/nuccore/937513727#
>ENA|ALI34365|complement 1830 nucleotides
ttgagtcttacagaaaaatgtgatatttatgcagctcttcatgatgcaggaattaataga
gtggttaaacatcttatgcaacagcgcccgtctctttttaattttggtacttccttttta
gcatcaaatccaaatctattgtgtgaaaaaattatagctgtgcaagaagtaaacagacaa
cagaatcctcttttaacaatactagaaccaattcctgtcattggtctaccattaagccaa
tctagttcaaacaatttggcaataaattttgcaattcaactttctaaagctgaaatagat
ttccatgaagggaatgtatttcctttaccttcagaacttgtcccattggcaaatcaacgt
ttggctactcatttcaaagtttgtgccggcttggattgtctcggggacttgcctgttctt
agaaggcctccaaataccagaccgtctacttccaaactagaatgtttttgtctagatttg
tatgctataggtggatgcaaaatctctggattagcagaaaaccaaaaaatagggctcaaa
attgatgggatagagatagtggatttaaaaccccaaggattggaaaatgcaatcgaatgt
tatgcaaaatctgtacttaatcatggaatttttccccatgttgctgagattatttccaaa
ttgacatttgggataattactattcctccatccaatggtaacttccaagtatcaggaaat
ttgcagctttcaggaactacgaccgttcctagtaatcctgcaatcgaagatgatcaatta
aaaacatttatcaatttagacaatttggacttggatatcgtattctcaccaaatggtggc
ggtagtggcggtagtggcggtagtggcggcagtggcggcagtggcggtagtggcggcagt
ggaacaataacaagaacagtgaggccgcgatcaagaacaggcacctttgatctaaccgct
gctatatccgaaactacatttaaaaaaatatatggtgcccttcttgaaggatttaaattt
tcagtatcagacacgggcggctctggtatattctcagtaggttatacagtttcggctcat
ttagagaatggcacaatcgatctgcggaataatggttctattctaataagtgaattagac
gtgaaatgggacacattgcagcttaacataggcatagatattccaacacaaactattggt
ggcttttgcctcgttccaaatccatttggtggatgtttgattcgtgctccgtcaattgat
ctttttgaagcatcacctgatatttctttacccttgaacttgggtggactaataacatct
gaaattactttgagtgtaattccaaaggtgttttatggagttggctctggaattccaaac
agatggcaacttaccttagtccctacgcttccaatcgacttggatataattgacgtagca
gacacaattggagatttatttcataatctgattacaaatgcaattgtcagtcttcttagt
tctttgggactccctgattgggctatatcttttatagataccgtgcttggtggaatagaa
ggaataatccgaaacattctagatattggagacgatgtgggtgaatttattttaagtttg
attagtaatgttggtctattccaggatttgattgataagctggctcaattcatagcgctt
acgattctggaactcaaggatcccttggaagttcttgagggaaattctgccataccttta
attccagtaaagttaccaattgaatttttggatgttcggatcgatggtaatggtaatgag
ttgatccttgaaggggatgtgggtaattag
Результат:

Развёрнуто: https://img-fotki.yandex.ru/get/97884/1 … 0_orig.png

В исследуемом белке обнаружен переход программной 335-спирали, характерной для коллагена первого типа в программную 553-спираль, которые строятся под музыку с размером 3/8 (в темпе вальса, точнее в темпе коллагена). Далее программная спираль переходит в бета-спираль длиной 4 аминокислотных остатка.
Кушелев: Модель коллагена (программная 335-спираль) очень яркая демонстрация несовершенства рентгеноструктурного анализа. Специалисты по РСА конструируют спираль коллагена из того, что им известно. А известна им альфа-спираль. Вот они и сконструировали модель из 3 альфа-спиралей. Программа Пикотех показывает, что это не тройная, а одинарная, но программная 335-спираль. Радикалы аминокислот действительно располагаются в виде трёх-заходной спирали, что и сбило с толку специалистов по РСА.
Другие программные спирали специалисты по РСА тоже ошибочно интерпретируют известными им "базовыми спиралями", но настало время показать, как устроены реальные, программные спирали белков. Некоторые из них имеют очень необычные параметры. Такова, например, программная 35-спираль:

Другими яркими примерами, демонстрирующими несостоятельность РСА, являются сверхдлинные в т.ч. программные спирали/
Наконец, Пикотехнология в отличие от РСА имеет возможность самоповерки. В частности, самоповеркой является 100%-ная корреляция между первичной и вторичной структурой некоторых белков, например, сверхдлинных программных спиралей. Спирали длиной более 1000 витков исключают случайные совпадения, согласитесь. Например, вероятность случайного совпадения композиционных кодов для 310-спирали длиной более 3982 аминокислотных остатков не превышает 4^-3982=2.5*10^-2397, т.е. более двух тысяч нулей после запятой
где РСА о том, что на участке из 17 а.о. в АПФ (белок от diprospan) действительно 3/10 спираль
Кушелев: А тут дело уже не в РСА, а в ничтожной вероятности повторения одинаковых кодов "4", "1", "3" и "2".
Если бы коды "1" и "4" были бы равнозначными, то почему встречаются длинные последовательности 1111111111 и 444444444444 ? По-Вашей гипотезе это бессмысленно.
По моей это прямые альфа- и 310-спирали соответственно.
Кушелев: Вероятность случайного расположения 17 одинаковых кодов = 0.25^-17 = 5.8*10^-11
Вероятность случайного расположения 22 звеньев цикла = 0.25^-22 = 5.7*10^-14

Вероятность случайного расположения 22 одинаковых кодов = 0.25^-22 = 5.7*10^-14
http://www.rcsb.org/pdb/explore/explore … ureId=4BT9
http://www.uniprot.org/uniprot/P13674
http://www.ebi.ac.uk/ena/data/view/AAA36534
http://www.ebi.ac.uk/ena/data/view/AAA3 … play=fasta
>ENA|AAA36534|AAA36534.1 Homo sapiens (human) hypothetical protein
ATGATCTGGTATATATTAATTATAGGAATTCTGCTTCCCCAGTCTTTGGCTCATCCAGGC
TTTTTTACTTCAATTGGTCAGATGACTGATTTGATCCATACTGAGAAAGATCTGGTGACT
TCTCTGAAAGATTATATTAAGGCAGAAGAGGACAAGTTAGAACAAATAAAAAAATGGGCA
GAGAAGTTAGATCGGCTAACTAGTACAGCGACAAAAGATCCAGAAGGATTTGTTGGGCAT
CCAGTAAATGCATTCAAATTAATGAAACGTCTGAATACTGAGTGGAGTGAGTTGGAGAAT
CTGGTCCTTAAGGATATGTCAGATGGCTTTATCTCTAACCTAACCATTCAGAGACCAGTA
CTTTCTAATGATGAAGATCAGGTTGGGGCAGCCAAAGCTCTGTTACGTCTCCAGGATACC
TACAATTTGGATACAGATACCATCTCAAAGGGTAATCTTCCAGGAGTGAAACACAAATCT
TTTCTAACGGCTGAGGACTGCTTTGAGTTGGGCAAAGTGGCCTATACAGAAGCAGATTAT
TACCATACGGAACTGTGGATGGAACAAGCCCTAAGGCAACTGGATGAAGGCGAGATTTCT
ACCATAGATAAAGTCTCTGTTCTAGATTATTTGAGCTATGCGGTATATCAGCAGGGAGAC
CTGGATAAGGCACTTTTGCTCACAAAGAAGCTTCTTGAACTAGATCCTGAACATCAGAGA
GCTAATGGTAACTTAAAATATTTTGAGTATATAATGGCTAAAGAAAAAGATGTCAATAAG
TCTGCTTCAGATGACCAATCTGATCAGAAAACTACACCAAAGAAAAAAGGGGTTGCTGTG
GATTACCTGCCAGAGAGACAGAAGTACGAAATGCTGTGCCGTGGGGAGGGTATCAAAATG
ACCCCTCGGAGACAGAAAAAACTCTTTTGCCGCTACCATGATGGAAACCGTAATCCTAAA
TTTATTCTGGCTCCAGCTAAACAGGAGGATGAATGGGACAAGCCTCGTATTATTCGCTTC
CATGATATTATTTCTGATGCAGAAATTGAAATCGTCAAAGACCTAGCAAAACCAAGGCTG
AGCCGAGCTACAGTACATGACCCTGAGACTGGAAAATTGACCACAGCACAGTACAGAGTA
TCTAAGAGTGCCTGGCTCTCTGGCTATGAAAATCCTGTGGTGTCTCGAATTAATATGAGA
ATACAAGATCTAACAGGACTAGATGTTTCCACAGCAGAGGAATTACAGGTAGCAAATTAT
GGAGTTGGAGGACAGTATGAACCCCATTTTGACTTTGCACGGAAAGATGAGCCAGATGCT
TTCAAAGAGCTGGGGACAGGAAATAGAATTGCTACATGGCTGTTTTATATGAGTGATGTG
TCTGCAGGAGGAGCCACTGTTTTTCCTGAAGTTGGAGCTAGTGTTTGGCCCAAAAAAGGA
ACTGCTGTTTTCTGGTATAATCTGTTTGCCAGTGGAGAAGGAGATTATAGTACACGGCAT
GCAGCCTGTCCAGTGCTAGTTGGCAACAAATGGGTATCCAATAAATGGCTCCATGAACGT
GGACAAGAATTTCGAAGACCTTGTACGTTGTCAGAATTGGAATGA

Много обещающая альфа-спираль с 21 по 71!!!
А что нам покажет программа Пикотех?
https://img-fotki.yandex.ru/get/194503/ … 6_orig.gif
Такова однако точность РСА...

Эта периодическая структура может оказаться ... научным прорывом. Но это мы проверим уже завтра...
Victoriy пишет:
Похоже я открыла ящик Пандоры, научив А.Ю. пользоваться PDB .
Уважаемый Александр Юрьевич, давайте вместе проанализируем Ваш материал по кодированию различных видов спиралей на основе экспериментально показанных спиральных участков в белках 1TAG; 3SDH; 1GPB; 1PZ4; 1OBO; 1GS6; 1H1N; 1LRV и АПФ от Дипроспана. Структура последнего энзима мне лично не безразлично, т.к. его активность в сыворотке крови пациентов с лакунарными инсультами я определяла в этом году в одной из текущих НИР.
Предлагаю в скайпе голосом, а потом Вы выложите свой материал на форуме.
На форум интересно текст выложить, а не голос Давайте сначала разберёмся с самоповеркой пикотехнологии
Согласитесь, что таких случайностей просто не бывает
Кстати, эта тема имеет продолжение
Итак, вопрос для богини пикотехнологии, Виктории Соколик:
"Вы согласны, что структуры альфа-спирали, 310-спирали, фрагмента лизоцима из этой темы однозначно подтверждают:
1. Кодирование прямой альфа-спирали кодами -1111111111111111111111-
2. Кодирование прямой 310-спирали кодами -444444444444-
3. Программирование цикла из 22 аминокислотных остатков лизоцима" ?
Если есть возражения, то жду Ваших аргументов. Пока мы не придём к консенсусу по данному вопросу, более сложные вопросы, как то нюансы статистики, я обсуждать не готов.
http://www.rcsb.org/pdb/explore/explore … ureId=5KYN

http://www.uniprot.org/uniprot/Q15436
http://www.ebi.ac.uk/ena/data/view/CAA65774
https://img-fotki.yandex.ru/get/50623/1 … 8_orig.gif
Как видим, данные РСА опять не соответствуют даже типу белка. Указаны десятки протяженных альфа-спиралей, а в структуре белка их нет

Очень любопытный тип программной спирали

[21:58:49] Кушелев Александр Юрьевич: Вероятность случайного образования прямой альфа-спирали из 22 ао = как 44 раза монетка упадёт на "орла"
[21:59:14] Кушелев Александр Юрьевич: Если Вы думаете, что это вероятно, попробуйте
Skype:
[28.11.2016 20:25:50] *** Кушелев Александр Юрьевич отправил контактные данные каштан. ***
[16:42:43] каштан: у вас на этой картинке
что изображено ?
[17:36:39] Кушелев Александр Юрьевич: Кольцегранная и упрощённая (многогранная) динамическая модель аминокислоты
[17:37:48] каштан: пять атомов ?
[17:37:51] Кушелев Александр Юрьевич: Да. Это глицин
[17:38:10] каштан: какие пять атомов ?
[17:40:57] каштан: https://ru.wikipedia.org/wiki/%D0%93%D0 … 0%B8%D0%BD
здесь написано про шесть атомов
[17:41:02] Кушелев Александр Юрьевич: NCCOO
[17:41:09] Кушелев Александр Юрьевич: Да, это они
[17:42:00] каштан: глицин это NHHHOO
вы что-то другое написали
[17:42:33] Кушелев Александр Юрьевич: Водород не показан
[17:42:45] Кушелев Александр Юрьевич: Только атомы углерода, азота и кислорода
[17:43:01] Кушелев Александр Юрьевич: NCCOO
[17:44:45] каштан: почему атомы одинаковых размеров ?
[17:45:43] Кушелев Александр Юрьевич: Потому что разных
[17:45:47] Кушелев Александр Юрьевич:
[17:47:19] каштан: на этой картинке атомы под другому соединяются

[17:48:39] Кушелев Александр Юрьевич: Одни и те же атомы углерода могут соединиться в алмаз и графит
[17:49:06] каштан: в смысле вкладываются друг в друга
[17:50:35] Кушелев Александр Юрьевич: Нет
[17:50:50] каштан: кислород в полтора раза больше углерода - на вашей картинке этого не видно
[17:52:16] Кушелев Александр Юрьевич: Углерод может иметь разный размер в зависимости от условий. Он может быть многогранным и сопряжённым
[17:55:02] каштан: вы говорите о группе атомов
сами атомы одного элемента одинаковые с незначительными отличиями по изотопам
ещё раз
атом углерода в полтора раза меньше атома кислорода
[17:55:29] каштан: соколик вам об этом говорила
[17:55:37] Кушелев Александр Юрьевич: Один и тот же атом углерода может иметь разные размеры и форму
[18:04:37] каштан: ATG - это метионин ?
https://en.wikipedia.org/wiki/Methionine
в состав которого входит сера ?
[18:05:27] Кушелев Александр Юрьевич: Да
[18:05:57] каштан: атом серы в три раза больше атома углерода
[18:06:30] каштан: и в два раза больше кислорода с азотом
[18:06:42] Кушелев Александр Юрьевич: И так бывает
[18:07:03] каштан: в вашей модели все атомы одинаковые ?
[18:07:57] Кушелев Александр Юрьевич: Нет. Даже один атом углерода может иметь сильно разные размеры. Я уже писал выше
[18:09:23] каштан: атом углерода может иметь размеры атома серы и в результате программа строит модель из атомов одинаковых размеров ?
[18:13:12] Кушелев Александр Юрьевич: Какие же это одинаковые размеры?

[18:14:11] Кушелев Александр Юрьевич:
[18:15:25] Кушелев Александр Юрьевич: Зеленые и красные могут быть атомами углерода (+водород)
[18:20:45] каштан: в этой модели
все атомы выглядят одинаковыми
соколик говорила о том что ваша программа не учитывает межатомные расстояния (размеры атомов)
найти это сообщение не представляется возможным потому что у вас на форуме нет поиска
[18:22:08] Кушелев Александр Юрьевич: Во-первых, на форуме поиск есть, но для зарегистрированных пользователей.
[18:22:34] Кушелев Александр Юрьевич: Во-вторых, я Вам уже показал, что даже один атом углерода может иметь разные размеры. Причём сильно разные.
[18:24:36] Кушелев Александр Юрьевич: Виктория говорит о том, что в моей программе не учтены изменения радиусов внешних электронов. Это действительно так. Но радиусы колец-электронов, например, для ионов кислорода, хлора и фтора отличаются меньше, чем на процент. У Виктории размеры электронов/граней отличаются на порядок больше, т.е. ошибочно. Поэтому моя программа хоть и упрощённая, но раз в 10 точнее
[18:27:51] Кушелев Александр Юрьевич: Но это только по радиусам электронов. Если говорить о вторичной структуре, то тут уже различие в точности принципиальное. Ведь у Виктории по кодам "1" и "4" строится прямая спираль с шагом 3 аминокислотных остатка на виток. А большинство спиральных участков имеет 3.6 аминокислотных остатка на виток. При этом фазовый набег приводит к тому, что спиральные участки у Виктории повёрнуты не на те углы, поэтому все модели белков "в раскоряку"
Victoriy пишет:
Виктория слегка подпортила таблицу;
Кушелев: Тут два варианта. Либо Виктория слегка подпортила таблицу, либо Виктория легко кинет монетку на "орла" 44 раза подряд
Victoriy пишет:
У Виктории размеры электронов/граней отличаются на порядок больше, т.е. ошибочно.
Кушелев: Сначала я определил радиусы кольцевых электронов из рефрактометрических радиусов ионов кислорода, хлора и фтора (Работа Гольдшмидта 1923г). Для ионов кислорода, хлора и фтора отличие радиусов электронов не превышало 1%. Позднее радиусы электронов удалось уточнить масштабированием белковых молекул. Ведь макромолекулы белка могут иметь гигантские размеры в масштабах микромира. Я подобрал размер кольцевого электрона таким образом, чтобы размеры белковых молекул, определённых экспериментально совпали с кольцегранными моделями в программе PiMol и HyperChem.
Совпадение размеров макромолекул получилось при диаметре кольца-электрона 1.7А.
Действительно, атом серы в том же метионине
или цистеине существенно больше атома углерода, там внешние электроны раздвинуты предвнешней оболочкой, но это - радикал, который не влияет на параметры белковой "цепи". Ведь параметры белковой "цепи" определяются геометрией пептидной группы. Согласны?
*
*
http://www.myshared.ru/slide/643151/
Цитата: 15 МЕТИОНИН - незаменимая АМК. Необходима для синтеза белков, участвует в реакциях дезаминирования, является источником серы для синтеза цистеина. Метионил-тРНК участвует в инициации трансляции. Метильная группа метионина - мобильный одноуглеродный фрагмент, используемый для синтеза ряда соединений в реакциях переноса этой группы на соответствующий акцептор (реакция транс- метилирования) Метильная группа в молекуле метионина прочно связана с атомом S, поэтому непосредственным донором этого одноуглеродного фрагмента служит активная форма метионина - S-аденозилметионин (SAM)
16 S-аденозилметионин (SAM) - сульфониевая форма метионина, образующаяся при его присоединении к молекуле аденозина (продукта гидролиза АТФ) Реакция активации метионина Присутствует во всех типах клеток Структура (S + -CH 3 ) в SAM - нестабильная группировка, определяющая высокую активность метильной группы (отсюда термин «активный метионин»). Это уникальная реакция, единственная, в результате которой освобождаются все три фосфатных остатка АТФ. Отщепление метильной группы от SAM и перенос ее на соединение -акцептор катализируют метилтрансферазы в реакциях транс- метилирования. SAM в ходе реакции превращается в S-аденозилгомоцистеин (SAГ).
Конец цитаты.
Кушелев: Понятно. Связь Met-Cys косвенно подтверждается, т.к. Cys образуется из Met, а значит, что в мышином лизоциме тоже возможно превращение Met в Cys в процессе дозревания белка.

>NM_207361.5 Homo sapiens FRAS1 related extracellular matrix protein 2 (FREM2), mRNA
GGGATTTCCCTCAAAGCCCAGGGTCCGGGGACCTGGAAAGTAGAAGTGGAGGGATTCAATTCTCCGCGCG
ATTGAGGCGCTAGCGGCGGAGCTGGACGGCCTGGGAAGGCTTCGGCTCCTCGGCTGCGGCTCCAGCCCGG
ACGGCGCCGCGCAACTTTGCCATCCTTCTGGCCCAGCCCCGGCTACAGGAGGACCCCGCGGGCAACGCGC
GGAGTTCCTGGCACTTCCCGGCGGTGTCTCTTGTTGTCTGCCCGGGGACCGACTTCGCATGCTCTCAGGC
TGACCTGTCCAAGCCCGAACACCGGGACCATGCACTCAGCCGGGACTCCCGGGTTATCCTCGCGCCGGAC
AGGCAACTCCACCAGCTTTCAACCAGGACCGCCACCGCCGCCCCGGCTGCTGCTGCTGCTGCTGCTTCTC
CTGTCACTGGTAAGCCGCGTCCCGGCACAGCCCGCTGCCTTCGGCAGGGCGTTGCTGTCCCCTGGTCTCG
CGGGGGCTGCAGGGGTCCCTGCTGAGGAGGCCATAGTGCTGGCGAACCGCGGACTCCGGGTGCCTTTCGG
CCGTGAAGTCTGGCTGGATCCCCTGCATGACCTGGTGTTGCAGGTGCAGCCCGGGGACCGCTGCGCGGTT
TCGGTACTAGACAACGACGCACTGGCCCAGCGACCGGGCCGCCTGAGTCCCAAGCGCTTCCCGTGCGACT
TTGGCCCTGGCGAGGTGCGCTACTCTCACCTGGGCGCGCGCAGCCCGTCTCGGGACCGCGTCCGGCTGCA
GCTGCGCTATGACGCGCCCGGAGGGGCAGTAGTGCTACCACTGGTACTGGAGGTGGAGGTGGTCTTCACC
CAGCTGGAGGTTGTGACTCGGAACTTGCCTCTGGTCGTGGAAGAGCTGCTGGGGACCAGCAATGCCCTGG
ACGCGCGGAGCCTGGAGTTCGCCTTCCAGCCCGAGACAGAGGAGTGCCGCGTGGGCATCCTGTCCGGCTT
GGGCGCGCTGCCTCGCTATGGAGAACTCCTCCACTACCCGCAGGTCCCTGGAGGAGCCAGAGAGGGAGGC
GCCCCGGAGACTCTCCTGATGGACTGCAAAGCTTTCCAGGAACTAGGCGTGCGCTATCGCCACACAGCCG
CCAGTCGCTCACCAAACAGGGACTGGATACCCATGGTGGTGGAGCTGCGTTCACGAGGGGCTCCTGTGGG
CAGCCCTGCTTTGAAACGCGAGCACTTCCAGGTTCTGGTGAGGATCCGAGGAGGGGCCGAGAACACTGCA
CCCAAGCCCAGTTTCGTGGCCATGATGATGATGGAGGTGGACCAGTTTGTACTGACGGCCCTGACCCCAG
ACATGCTGGCAGCCGAGGATGCTGAGTCTCCCTCTGACCTGTTGATCTTCAACCTTACTTCTCCATTCCA
GCCTGGCCAGGGCTACTTGGTGAGCACCGATGATCGCAGCCTGCCCCTTTCCTCCTTCACTCAGAGGGAT
CTGCGGCTCCTGAAGATTGCCTACCAGCCCCCTTCTGAAGACTCTGACCAGGAGCGCCTCTTTGAACTGG
AATTGGAGGTAGTGGATCTAGAAGGAGCAGCTTCAGACCCTTTTGCCTTCATGGTAGTGGTGAAGCCCAT
GAACACAATGGCTCCGGTGGTCACCCGGAATACCGGTCTTATTCTCTATGAGGGTCAGTCTCGGCCCCTC
ACAGGCCCTGCAGGCAGTGGTCCGCAAAACTTGGTCATCAGCGATGAGGATGACCTAGAAGCAGTGCGGC
TAGAGGTGGTGGCTGGGCTCCGGCATGGTCACCTTGTCATTCTGGGTGCTTCCAGTGGCAGCTCTGCTCC
CAAGAGCTTTACAGTGGCTGAGCTGGCAGCCGGCCAGGTGGTCTACCAGCATGATGACAGAGACGGCTCG
CTGAGCGACAACCTGGTGCTTCGCATGGTGGATGGAGGAGGCAGGCACCAGGTACAGTTTCTGTTCCCCA
TCACCTTAGTGCCTGTGGATGACCAGCCACCTGTTCTCAATGCCAACACGGGGCTGACACTGGCAGAGGG
TGAAACAGTGCCCATCCTGCCCCTTTCCCTGAGTGCAACTGACATGGATTCAGATGATTCTCTGCTGCTT
TTTGTGCTGGAGTCACCCTTCTTAACTACGGGGCATCTGCTTCTCCGCCAAACTCACCCTCCCCATGAGA
AGCAGGAACTTCTCAGAGGCCTTTGGAGGAAGGAGGGGGCATTTTATGAGCGAACAGTGACAGAGTGGCA
GCAGCAGGACATAACAGAGGGCAGGCTGTTCTATAGACACTCTGGGCCCCATAGTCCTGGGCCAGTCACA
GACCAGTTCACATTTAGAGTCCAGGATAACCATGACCCTCCTAATCAGTCCGGGCTACAGCGGTTTGTGA
TTCGTATCCATCCTGTGGATCGCCTCCCTCCGGAGCTGGGCAGTGGCTGTCCCCTTCGTATGGTGGTACA
GGAATCCCAGCTCACACCACTGAGGAAGAAGTGGCTGCGCTACACTGACCTGGACACAGATGACCGAGAA
CTACGTTACACAGTGACTCAGCCCCCCACAGACACAGACGAAAATCACCTGCCAGCCCCACTGGGTACCT
TGGTCTTGACTGACAACCCCTCAGTCGTGGTGACCCATTTTACCCAAGCCCAGATCAACCATCATAAAAT
TGCTTACAGACCCCCGGGTCAAGAACTGGGCGTGGCTACTCGAGTGGCCCAGTTCCAGTTCCAGGTGGAA
GACCGAGCTGGGAATGTGGCTCCAGGTACCTTTACCCTTTACTTGCATCCCGTGGACAACCAGCCACCTG
AGATCCTCAACACCGGCTTCACTATTCAGGAGAAGGGTCACCACATCCTGAGTGAGACAGAGTTGCACGT
GAATGATGTAGACACTGATGTTGCCCATATCTCTTTCACTCTCACTCAGGCACCCAAACATGGCCACATG
AGAGTGTCTGGACAGATCCTGCATGTAGGGGGTCTCTTCCACTTGGAGGACATAAAACAGGGCCGAGTTT
CCTATGCCCATAATGGGGACAAGTCCCTGACTGATAGCTGCTCCTTGGAAGTCAGTGACAGACATCATGT
GGTGCCCATCACTCTCAGAGTAAATGTCCGGCCAGTGGATGATGAAGTGCCCATACTGAGCCATCCTACT
GGCACTCTGGAGTCCTATCTAGATGTCTTAGAAAATGGGGCTACTGAAATCACTGCCAATGTTATTAAGG
GGACCAATGAGGAAACTGATGACTTGATGTTGACTTTCCTCTTGGAAGATCCACCTTTGTATGGGGAAAT
CTTGGTCAATGGCATTCCAGCAGAGCAGTTTACTCAAAGGGACATCTTGGAGGGCTCTGTTGTATATACC
CACACCAGTGGTGAGATAGGCCTATTGCCTAAAGCGGATTCTTTTAACCTGAGTCTGTCAGATATGTCTC
AAGAATGGAGAATTGGTGGCAATACTATCCAAGGAGTTACTATATGGGTGACCATCCTGCCTGTTGATAG
CCAGGCCCCAGAAATCTTTGTAGGTGAACAGTTGATAGTAATGGAAGGTGATAAAAGTGTTATAACATCA
GTGCATATAAGTGCTGAAGATGTCGACTCCCTGAATGATGACATCTTGTGCACTATAGTTATTCAGCCTA
CTTCAGGTTATGTTGAAAACATTTCTCCAGCACCAGGCTCTGAGAAATCAAGAGCAGGGATTGCCATAAG
TGCTTTCAACTTGAAAGATCTCAGGCAGGGCCACATAAACTATGTCCAGAGTGTCCATAAAGGGGTGGAA
CCTGTGGAGGACCGATTTGTATTTCGTTGTTCTGATGGCATTAACTTTTCAGAGAGACAGTTCTTCCCCA
TTGTAATCATTCCCACCAATGATGAACAGCCAGAGATGTTTATGAGAGAATTTATGGTGATGGAAGGCAT
GAGTCTGGTAATTGATACACCCATTCTCAATGCTGCTGATGCTGATGTTCCCCTGGATGATTTAACTTTC
ACTATTACCCAATTCCCCACTCATGGTCACATCATGAATCAGCTGATAAATGGCACGGTTTTGGTCGAAA
GCTTCACCTTGGATCAGATCATAGAGAGTTCCAGCATTATTTATGAGCATGATGACTCCGAGACCCAGGA
AGACAGTTTTGTGATTAAACTAACAGATGGGAAGCACTCTGTGGAAAAGACGGTCCTCATTATAGTTATC
CCTGTTGATGATGAGACGCCCAGAATGACTATCAATAATGGACTAGAAATAGAAATTGGGGATACCAAGA
TTATCAACAACAAAATATTAATGGCAACAGATTTAGATTCAGAAGACAAATCTTTGGTTTATATTATTCG
TTATGGGCCAGGACATGGCTTATTACAGAGACGAAAACCTACTGGTGCCTTTGAAAATATCACACTGGGC
ATGAATTTTACCCAGGATGAAGTAGACAGAAACTTAATTCAGTATGTCCATTTGGGGCAAGAGGGCATTC
GGGACCTAATTAAATTTGATGTGACTGATGGAATAAATCCCCTCATAGATCGTTACTTTTATGTGTCCAT
CGGGAGCATTGACATTGTCTTCCCTGATGTGATAAGTAAGGGAGTGTCCTTGAAAGAAGGTGGCAAAGTC
ACTCTTACAACAGACCTACTAAGCACTAGTGACTTGAACAGTCCTGATGAAAACTTGGTTTTTACCATCA
CCAGGGCTCCCATGCGAGGTCACCTGGAATGCACGGATCAGCCTGGTGTGTCCATCACGTCTTTCACTCA
GCTGCAACTGGCTGGAAACAAAATCTACTACATCCACACAGCTGATGATGAAGTGAAAATGGACAGTTTT
GAGTTTCAAGTCACCGATGGACGTAACCCTGTCTTTCGGACATTCCGTATCTCCATTAGCGATGTGGACA
ATAAAAAGCCAGTGGTCACCATCCACAAGCTGGTTGTCAGTGAAAGTGAAAACAAGCTGATTACTCCTTT
TGAGCTCACTGTCGAAGACAGAGATACTCCTGACAAGCTCCTGAAATTCACTATCACCCAGGTGCCTATT
CATGGCCATCTCCTATTCAACAATACCAGACCTGTCATGGTTTTTACCAAGCAAGACTTGAATGAAAACT
TAATCAGCTACAAACATGATGGCACTGAGTCAAGTGAAGATAGCTTCTCCTTCACAGTGACTGATGGCAC
CCATACAGACTTCTATGTTTTTCCTGATACGGTGTTTGAAACAAGGAGACCCCAAGTGATGAAGATCCAG
GTCTTGGCTGTTGACAACAGTGTCCCCCAAATCGCAGTGAATAAGGGGGCCTCTACACTTCGCACTCTAG
CCACTGGCCACTTGGGGTTCATGATCACAAGCAAAATATTGAAAGTGGAGGACAGAGACAGCTTACACAT
TTCTCTTAGATTTATCGTGACAGAGGCCCCTCAACATGGATATCTTCTCAACCTGGACAAAGGCAACCAC
AGCATCACTCAGTTCACACAAGCTGACATTGATGACATGAAAATATGCTATGTCTTAAGAGAAGGGGCTA
ATGCCACAAGTGATATGTTCTATTTTGCAGTTGAAGATGGTGGTGGAAACAAGTTAACGTACCAGAATTT
TCGTCTGAATTGGGCATGGATCTCCTTTGAAAAGGAATATTACCTGGTCAATGAGGACTCCAAATTTCTA
GATGTTGTTCTTAAACGTAGAGGTTACTTGGGAGAAACTTCTTTTATAAGTATTGGCACAAGAGACAGAA
CTGCAGAAAAAGACAAAGACTTCAAGGGCAAAGCACAGAAACAAGTGCAGTTCAACCCAGGCCAGACCAG
GGCCACATGGCGAGTGCGGATCCTGAGTGATGGGGAGCATGAGCAGTCTGAAACCTTTCAGGTGGTACTC
TCAGAGCCCGTGCTGGCTGCCTTGGAATTCCCCACAGTCGCCACTGTTGAGATCGTTGATCCAGGAGATG
AGCCAACTGTGTTTATTCCCCAGTCCAAATACTCCGTTGAAGAAGATGTTGGTGAGCTGTTCATTCCCAT
CAGGAGGAGCGGAGATGTGAGCCAGGAGTTGATGGTGGTCTGTTATACCCAACAAGGAACAGCAACTGGA
ACTGTGCCGACTTCCGTGTTGTCTTACTCTGATTACATATCCAGGCCTGAGGACCACACCAGTGTTGTCC
GCTTTGACAAAGATGAACGGGAGAAACTGTGTCGGATAGTCATAATTGATGACTCTTTGTACGAGGAGGA
GGAAACCTTCCATGTCCTTCTGAGCATGCCCATGGGGGGAAGAATCGGATCAGAGTTCCCAGGGGCTCAA
GTTACAATCGTTCCTGACAAAGATGATGAACCCATCTTTTACTTCGGTGATGTGGAATACTCTGTGGATG
AGAGTGCTGGCTATGTGGAAGTGCAGGTGTGGAGAACGGGCACTGACCTGTCCAAGTCTTCTAGTGTCAC
AGTGAGGTCTCGGAAAACAGATCCTCCCTCTGCAGATGCTGGAACAGACTATGTGGGCATCAGCCGTAAT
TTAGATTTTGCACCTGGAGTCAACATGCAGCCTGTTCGTGTTGTCATTCTGGATGACCTTGGACAACCAG
CGCTGGAGGGAATTGAGAAATTTGAACTGGTGCTTCGCATGCCTATGAACGCAGCCCTTGGCGAGCCCAG
CAAAGCCACAGTGTCCATAAATGACTCTGTCTCCGATTTGCCTAAGATGCAATTCAAAGAACGAATATAT
ACTGGCAGCGAAAGTGATGGGCAGATAGTTACAATGATCCATAGGACTGGGGATGTCCAGTACAGATCTT
CAGTGAGATGCTACACCCGGCAGGGGTCTGCACAGGTGATGATGGACTTTGAAGAACGCCCAAACACTGA
TACCTCCATCATCACATTCCTCCCTGGTGAGACAGAAAAGCCCTGCATTCTTGAGCTGATGGACGATGTG
CTCTATGAGGAGGTAGAGGAGCTCCGCCTGGTACTCGGCACTCCACAAAGCAACTCTCCCTTTGGGGCTG
CAGTTGGTGAACAAAATGAAACTCTCATAAGGATCCGAGATGATGCTGATAAGACTGTTATTAAATTTGG
AGAAACCAAATTTAGTGTCACTGAACCCAAAGAACCTGGAGAGTCGGTGGTTATAAGAATTCCAGTGATT
CGCCAAGGAGACACTTCAAAGGTTTCCATTGTGAGAGTCCACACCAAGGATGGCTCGGCCACCTCTGGAG
AAGACTACCACCCTGTGTCAGAAGAAATTGAGTTTAAGGAAGGGGAAACCCAGCACGTGGTTGAAATCGA
AGTTACCTTTGACGGGGTGAGAGAGATGAGAGAGGCCTTCACTGTTCACCTAAAACCTGATGAAAATATG
ATAGCAGAGATGCAGTTGACGAAAGCCATTGTGTACATAGAAGAAATGAGCAGCATGGCAGATGTCACTT
TTCCTTCTGTCCCTCAAATTGTATCCCTGTTGATGTATGACGACACTTCCAAAGCTAAGGAGAGTGCTGA
ACCCATGTCTGGCTATCCTGTCATCTGTATCACAGCTTGCAACCCCAAATATTCAGACTACGATAAAACA
GGCTCTATCTGTGCAAGTGAGAACATCAATGACACTTTGACGCGGTACCGGTGGCTGATTAGTGCACCTG
CGGGCCCTGACGGTGTGACCAGCCCTATGAGAGAAGTGGACTTCGACACCTTTTTTACGTCATCCAAGAT
GGTCACACTGGACTCCATATACTTTCAGCCTGGCTCCCGGGTACAGTGCGCAGCTCGTGCTGTGAACACC
AATGGGGATGAAGGCCTGGAGCTCATGAGCCCTATTGTAACCATCAGCAGAGAAGAAGGTCTTTGTCAGC
CCCGTGTACCTGGGGTTGTTGGAGCAGAGCCGTTCTCAGCTAAATTGCGCTACACAGGCCCTGAGGATGC
AGACTACACAAACCTTATCAAGCTCACTGTCACAATGCCACACATAGATGGCATGCTCCCCGTGATCTCC
ACTAGAGAGCTTTCCAACTTTGAGCTCACCCTCAGCCCTGATGGCACAAGAGTTGGAAACCACAAGTGCT
CCAACCTCCTGGATTATACTGAAGTGAAGACTCATTATGGTTTCTTGACTGATGCTACCAAAAATCCAGA
AATAATTGGAGAGACATATCCTTACCAGTACAGCTTGTCCATCAGAGGTTCCACTACCTTGCGCTTCTAC
CGGAACCTGAACCTAGAGGCCTGTTTATGGGAGTTCGTTAGCTACTATGACATGTCAGAACTCCTTGCTG
ACTGTGGTGGCACCATTGGAACAGATGGACAGGTCCTAAACCTAGTGCAGTCCTATGTGACCCTTCGAGT
CCCTCTGTATGTTTCCTACGTGTTCCATTCCCCCGTGGGGGTAGGAGGCTGGCAGCATTTTGACTTGAAG
TCAGAGCTTCGTCTAACTTTTGTGTACGACACCGCCATCTTGTGGAATGATGGAATTGGCAGCCCCCCAG
AGGCTGAACTTCAAGGTTCTCTCTATCCAACCAGCATGCGCATCGGTGATGAGGGGCGCTTGGCCGTGCA
CTTCAAGACAGAGGCTCAGTTCCATGGCTTATTTGTGCTGTCACATCCCGCATCCTTTACAAGCTCAGTG
ATCATGTCAGCTGATCATCCAGGCCTGACATTTTCCCTCCGCCTCATAAGGAGTGAACCAACCTATAACC
AGCCAGTACAGCAGTGGAGCTTTGTCTCTGACTTTGCCGTACGTGACTACTCAGGGACCTATACTGTGAA
GCTGGTGCCATGCACTGCCCCATCACATCAGGAATACCGCCTGCCAGTCACCTGCAACCCCAGAGAACCT
GTCACCTTTGACCTTGACATCCGATTCCAACAGGTCAGTGATCCAGTGGCTGCTGAGTTTAGCTTGAACA
CCCAAATGTACCTGCTCTCTAAGAAGAGTCTCTGGTTGTCTGATGGATCCATGGGATTCGGGCAAGAGAG
TGATGTTGCTTTTGCAGAAGGTGATATAATTTATGGTCGTGTCATGGTGGATCCTGTCCAGAATCTGGGT
GACTCCTTTTACTGCAGCATTGAGAAGGTGTTTCTATGCACTGGAGCTGATGGCTATGTTCCCAAGTATA
GTCCAATGAATGCAGAATATGGCTGCTTAGCCGACTCTCCTTCACTCTTATATAGATTTAAAATTGTGGA
CAAAGCTCAGCCAGAGACACAAGCGACCAGTTTTGGAAATGTCCTATTTAATGCCAAACTAGCAGTGGAT
GACCCTGAAGCCATTCTCTTAGTGAATCAGCCTGGATCTGATGGATTTAAAGTCGACTCAACACCACTCT
TTCAGGTCGCTCTAGGCCGAGAATGGTATATACATACGATCTATACAGTGAGATCGAAAGACAATGCCAA
TCGAGGTATTGGCAAAAGAAGTGTGGAGTACCATTCTCTGGTGAGTCAAGGAAAGCCCCAATCCACCACC
AAGAGCCGGAAGAAGAGAGAGATCAGGAGCACACCCTCACTGGCATGGGAGATTGGTGCTGAAAACAGTC
GAGGAACAAACATCCAGCACATTGCCCTGGACCGCACCAAGAGGCAGATCCCCCATGGGAGAGCACCTCC
AGATGGCATCCTCCCCTGGGAGCTCAACAGCCCCAGCTCTGCAGTCAGCCTGGTCACTGTGGTGGGAGGC
ACCACGGTAGGGTTACTCACCATCTGCCTCACTGTCATTGCAGTGCTGATGTGCAGGGGCAAGGAAAGTT
TCAGGGGGAAGGATGCCCCGAAAGGCTCCAGCAGCAGTGAGCCCATGGTGCCCCCACAGAGCCATCACAA
TGACAGCTCAGAAGTTTGATGACTGCAGGTAGAATTCAACCTTTTCCGTAAGTGCCTCGGAAAAGATCAC
AATGGAACCTTAAATACTTCTGGTAAACCATAGAGAATGGAGGATGGCTGTGATGAAGCTGCTTAGAGAA
TATGACTGTACTAATGGAGTTTGATTTGAATTCTCCATACTCTTTTTTGCATCAAAGGACAAATTAAGGC
ATCTTTCCTCTTGCCCCAAAGGCAGCAACAACGTACTCATATGTACACAGAGCCATGATGTGAGGAATGT
ACTCCTCATTTTAGACATATTCTCTATGCAGTGGAGATAAATCTATTAAAAGGTGCTAACAGACTCTCTT
ACAAGTGTAAGAGGAATCTACTGTTGCTATGTTAGTGTGAATGTTGAAGGTGCAATTATCACATTGTTTA
TTAATTGTATAACAGATTATTACTAGAAAGGTTTTTGTTGCTAGTCTGGAAAACTGGTGAACACACTGTT
TATTATGACAAGTTTTCAAATAGGTGAAGACAGCATAGAATTATGACCAGGGTGGCTCAACCCACAAATC
AACTGATCTACTACTTGGGAGACAGTGGAAGGAAAGATAGAGGGAAGGAAGTTCACTCACTTGAATTTGA
ATTGCTTCTCATTCTCATCAGACTCTCCTTGTTTTGTTTGCATATTTAATTTTAAATTAAACCAAAGAAT
ATGTTAATTCTGAAAGAGATTTTTAAGAGATGCTGCTCTGTTTTGGCATATAGTGAGTAATGGTAGAGCT
CTTGCATGGTAATATACCTTATTGGTGCTCAACATTTGTGGGAAATTAGAGGGTTGGTGAGATTTTGGTG
ATGAATAAATGCCATAGAGTTAGAGTCACTACAAGACAATGTTCTCTAAGAACTGAGCCTAGATGTTTGC
AGTATTGAACCCATAAATGATAATAAGAAACATTGTTACAGATGGTGAAGGGAGAAAGTGGTATTTATTA
ATATAATGTGTATAATCAGAGTGCCTCTTATACATACTTTATAGAATAAAGGAACATTTTGACAGATGAG
GTTATCCCTCGGAGTAGCGTAATCACACAATAACATTTAGAACTTAAATTGGCTACAGGACATTTTATTA
GCTTTCTATCCAGCATCTGGTCCAAACGATGGGACTCTCTGCAACTATCATTCCAGAAATGGTCTTGGAG
GTGGCCAGAATCATTGACCATATTTTTATTTGGTTCAAGAAGACATCGCTCTGGCATCCCCAGTTCAGTG
AACTGGTACAATGTGGTCCCTCTCAATATTTAAAAATATAATTTGTAACAGTGCTTACCATTCACCTCCC
AAAGCCAAGGAACTTGAGTGAGCTTCCTTGGCAAGCCTCCCATTGCCTTTCTGCATCAATGTGTTTGTTA
CCAAATAGCAGAAATTTCGCTTAGAAGGGGAAAACTTCAGCTTTCCAAAAGCTAGTAAACATGTTTCAAA
GAAAAATAAGGTCAGGAGAAAAAGAATAATGCCTACAGATTATCCTTCAGATGCTGGCTAGAATCCCCTT
TGACTTCTAGCCATAAGCTGGCCACATTGGACATCTTGGTCACTGCAGAATTTTAAAAGGCAAACTTCTC
AGAACAGATGTGCACATCTTAATTTGGTGTCTGTAAGTATCAACCATTTCTTGATCAAGATAAGGAAGCA
ACACTGATACTGAGAGAGAGGTCATATGTCCAATAGTTGCCTTTCTTCTCTTAGGCAGCTCTGCACTTCA
TGTTCAGTTTTTTAAAAATAGACTGTAGTATCCTTTCATTGGAGAATTTACAAAAATATCATCTACAGCT
CAAAAGTCCCACTAACGGCTTATATGAACAGAAAGTACTGTGTAGCCCAGGAGTTTATTTAGCAAGATAA
ATAGTGGTAATTTCTTAATCATTCCCCTTTCCTTTGCTTTACCTTCTGGCAGATGCTTAGCTACTTCTTT
GGCTACCTCATAATTTGTATGGGAATGAACATGAATTAGGGAACTTCAAATAGTGCGTAGACTTTTCCCT
ACTTACCATTGGGTTCAATTGATGTAGACTGTTAGAGCAAGTATACTCAATCAACTTGCTGATATTTTAG
TAGTTAACAGCTTCCGAGCCTATCATGCAATTTGGAGTCTGTCAATGAATATAGCAATCGGTGAAGCATT
ACTGTACGAAGTCTGTTTTCAGGCTTTGGGTCAAGCTGGTTTTATACCAAATTTCACTCTACAATGCATA
TTCTAATGAAGCTACTTTATATAAATTGGAGAGCTTATAAAATGCAGAACATTTCTATCCTATCCTACCA
AATATTTATCCTTCTCTGTCCTCTATTCTCTTACTCTCCTCTCTCCCTTTTTCCTTCCCTTTATTCCCTT
GAAAAAAGAGAATCTGTGGGAAATAGGCAGATATAGCTTTTTAAAATATAAAGTACTTGGGATTTTGCAT
TATTTTTCTTAATATCTATTTACTAAGTATGTAATTTAATATACATTAATTATTTTTTGAAACTCTTGTT
AGTGGGAAGAATATGGTAAATTTTTTGTTAAATAAAATAGACCCTTATGTTTAGCATTTTGTTTTTAGAG
AACTATTCTGGTACTATCAGAACAAATACATAAAATAACTTCCCATAGAGAACAGGATATAGCAATAATA
GCTCCTTAGATACTCAGTGGCTTCTGACTCCAATCAAGGTCTTGTTGATATTATATAGTAAAAATAAAAC
CAAAAATAAATATTATTCAAGTGGCTCTTCTAAGCATGTGAATCATGAAGCACTGAAATATGTATTTTAA
TGATGATCTTATTTATTCCCATTTTTGCCCTTAGTTAACATTTACTGGTGCTCACCTAGGATTGGCTATT
CTGAGGGATTGCATAGAAACCAAGCTCCACTTGCTGTCCTTGGGAAGGTTATAACTGAATGCAGCTCTTT
ATTTGGACTAAAGTGTCAGGATATGCATTAGATTCTCTCCTGAACCAAAAACACAACAGTCATTATCTGT
GAACCATAATTTAAAAATCTTTCTAGAATAACAACAGCAGACTCCACTCTTGTTTGTCTAAAAGAGCCCT
ACTGGGTATGGATCATTCTGATGACAGATTTATACAAAATGATTCAAACCAGTAACTTAGTAAAATTGAC
CTTCGCAAAACCTCACTGGGGGAGTGCCTTGTAGAGCTGTGGGTGGGACTGCACATTCTTTTCCTCTTAG
TAAAAGATAGGCCCACTTTATTCCAAGAATAACACTTAGCACATAAACTCTTCTTCCAGCTCGTTAGCAG
CATTAGCACCTTCTGAATTCCACCCTCTCAGAAGAATCCACAGTGTTTGAACAATTTGCATAAAGGTCAG
CTAGCATCCTGCTGCCAAGCCACTGCATAGCATTTGTGATAAGAAGGACCAACTCTAGGCTCAATATGAA
GGGATTTAGTTCTGTAAGCAGCAAAAAAGCTTCTTTATCAAGTCATCTTACCTCTAATTCTTTTCCAGTG
TGCCAACTCCAAAGTCAACATTAAAAATGTAAATGGACCTGTGTAAATATCACAGAGAGCTTTTCCTTAT
ACATCTCAATGCTGAGAGTTAAAATATTCCCAGGTTAAAATTTTTTTAAAGTACCAATAATAGAGCTAAA
TACAATGACATTTGCTTTTAAAAGGTGGATATTTTATTTCTGCTTTTTGAAAATACTTATTTAGTATTGA
CTTGGAAGCCAATTTGGTCCTTTAATAAGTAAAGAAAATAATATGTTTAAAAATGTAAATGTTTTACAAA
TTTGAAACTTTCATAATTGTATTAATCAGAAAACAAGCACATTGCCATTCTTTGAAACTCATGTTTCTAG
ACATGACAGCAGTAATAAAAGGATGAAAACAAGTGTCTTCACTAAGCGTATGGCCAATAAATGGGACCCA
AACGTTCAATCTGTTCAGTTTACCAAGGTTCAGAAATACGTAATTTAGCAGGAAACTATAAATACCAGTG
CTATCACAGCCACACATACACACACACAGACATAAAATAACCAAACATCTCATTTCTAGGAAAGAGATAA
CACTAAAGGCATCATAGGTTTAACTGAAATACGTTATATGAAGTTTTACAAAAAGGTCAACAGAAAGCTC
ATTTGTGAAAACATACTCTCATGGGAGCTTCTTTAACATTAGTTCAGAGGTTAATATATTTCCTGGAGGT
GTTTTCCTAGAATTGATTGCACTATTGCATGGTAATAACATTTAATTGTTAAGGAAACATTATATATAGG
TTCAAATTATCCCTTAATGTTGATTTCTCCCCTTTTCCATGGATTTTGATACTAAGAAACAAAATGCTTT
GAGATTTTGGTAACTATTTTGATTTTGATAAAACATGTTAAAATAGAAGGACATGATATTTTTCTATAGT
TTCCATCAGGAAGAGTACATCAGAAACTTCTCCATAAGGAAAGAAAACTGACTCTCTCTTGAACTAGTGT
TGACAAAATACACTAATGTCTTTCTTAATTTTATTTTATTAGGAGAAAAATCAGAATATTAAATTTGCAA
ACTTTTGAACAGCAGTAATAGCTCCTTAGACACTCAGTATCTTTCTTCCCTATCTTTCAAGCACATTTAA
TTTCTTTCCCCTGGTATTTGTTTGCTTTGTGTTTTTTCAATATTTTGTTTGCTAAAGAACAATAACAACA
ACAAAGCTGGTCCAGCCCTGACACCTACTTTTGCTTTTTTTCTTTCTTTCTTTTAATTTGTTGAACTATA
GGTTTCTTTCTGAGATGTATTTTTCATTCTTCCAATAATGCTCTCTTTTTCATTTCAAAACCATTACCAC
TTTCAATATGTAATTAACATCCTCATTCATCAGAAAGCTGGTAAGTCCACACAGCATTTGTGCAGTATCC
CTTCTGTGTAAAATGTTAAGGCTTTCAGATGAAAGAAAAAAGAACTTCCTGCAAAAGATCAAAAGCATCA
TACTAAATAAAATGATGACAATATGTGCCTTGATTTTTCTCTCAATGGAGGTTCAACATTGACATTTAAG
ATGTAAAAAAAATTACTTTGATGGTTTAATTAAGAATAATAAGTCAAATATCACATGTATGTAGTTCTTT
TGGATGACCGAATACCTTAAAATGAACTAAATAACTGAAATAAAGTGTTAAAATGTTAACAGCGTTGAAG
TTTTTAAAATTATAATTATCAAATGCACAATGTTCTTTTACTATGCCAAATGTTGCCTACACTGATTACA
TGATTGTCACAGTTTAAGTGAATTTCTACTATGGATCTTTATTTTCCCCAATGTTTGGATCATCCTCTCA
GATAAGAATTGTCAGTATTGCACCTCAAGAAAGAAGAAAAAATAAGAATGTAAATTTTTAAAAGCTAGCA
TTAAAAGTAAAGAGCAGTACAATAAAAATCACTTAAAATAGAGAAAGGCAACTACTGAGTAAGAAGAAAA
GATAATTTCTGTAAAAGCTAAATTTATGCACATTACAAAAAGTAGTCAAGTCAGTCTTCCATTGTGACTT
CAGGAATTCAAATGCAAGTTGTTGGCAAAATATATATATACACACACACACACATACACACACATATATG
TACATACATATATATATAGAGAGAGAGAGATAGATTTTTTTTTTTTGAGACAGAGTTTCGCTCTTGTTGC
CCAGGCTGGAGTGCAATGGAATGATCTTGGCTCACCGCAACCTCCGCCTCCAGGGTTCAAGTGATTCTCC
TGCCTCAACCTCCTGAGTAGCTGGAATTACAGGTGCCCACTACCATGCCTGGCTAGTTTTTGTATTTTTA
GTAGAGACAGGGTTTCTCCATGTTGGTCAGGCTGATCTCGAACTCCCAACCTCAGATGATCCTCCCGCCT
CCACCTCCCAAAGTACTGGAATTACAATGGCGTGAGCCACCACAAGATATTTTTAAAGCCCATTTGTCTA
TAAAAGAAAACTCTTGGACAAGGAGCTTTGTGTTTTATAATTGTATTACGTGCCTGCTTCCTTACCAAGT
GCCCTCCATAACACTAAGTAAATTTATATTTATCTAGCCTCTGAGTGACATATCTGTGTGTAATGGAATT
ATTTGCAGAATTTAATAGCCTTTTTTATTTTCGCATGAGAATTTTTCAAAAACTATCAGGGTCTAGTTTT
ATCATACATTTACTAAGTTACTACATATTGGTGGATATTGACGTTTATTCCTGGGAATCTAATTTTGCAA
ACAATTTGGAGCCATTCAGTCATAAATTCATGTCAACAATAAGTCTTGCTGCCGCTTGTGTTTCATAACT
ACGCTTGCTTTCCTTCAAAATATACCAAGTGTGTAATATAAATAAAGCCCATATCAATATAAAAAAAAAA
AAAAAAAAA
Композиционный код 6:
5,3,5,5,3,5,5,3,4,4,1,1,3,2,3,5,5,2,5,3,3,5,5,3,5,5,3,5,5,2,3,2,5,3,5,5,3,5,3,3,4,4,4,1,1,1,2,5,5,5,
2,3,5,2,1,1,1,1,1,1,1,1,1,1,1,4,4,2,5,5,2,3,3,5,3,4,4,4,3,5,5,5,3,5,5,2,5,3,5,3,3,1,1,4,1,3,2,1,4,1,
1,4,1,6,1,2,5,5,3,5,5,3,1,4,1,4,2,1,1,1,1,1,3,3,2,2,5,2,4,4,1,4,4,4,4,4,4,4,3,5,5,2,5,2,1,1,1,4,2,5,
5,3,1,1,1,1,4,1,4,1,3,3,4,4,4,3,2,5,5,3,3,5,5,5,2,4,4,4,1,1,2,4,4,4,3,5,5,3,3,4,4,4,3,5,5,3,1,4,4,1,
1,4,1,1,4,1,1,1,4,3,5,2,2,5,5,5,2,5,5,5,2,4,1,1,4,3,1,1,1,1,4,1,1,3,5,3,1,1,4,1,1,3,1,4,1,4,1,1,4,3,
4,1,1,1,4,4,1,4,1,3,5,5,5,2,5,2,2,5,2,2,5,2,4,1,4,1,4,1,1,1,1,4,1,3,5,3,5,5,5,3,4,4,4,3,1,4,4,4,1,1,
3,1,4,1,4,4,1,4,1,1,1,1,1,1,1,2,1,1,1,1,4,1,1,4,1,1,1,1,4,4,3,5,3,2,3,1,1,1,1,4,1,1,3,2,2,5,3,5,2,1,
1,4,1,3,1,4,6,1,1,3,3,5,5,3,2,5,5,5,3,5,5,2,5,5,3,5,2,2,1,1,1,1,2,1,6,4,4,1,4,3,2,2,5,3,3,5,5,5,3,3,
5,3,1,1,1,1,1,3,4,4,1,1,2,2,4,1,1,1,3,2,5,5,5,3,1,4,1,6,6,6,6,1,4,1,1,3,2,4,4,1,4,1,2,4,4,4,2,5,5,3,
3,5,3,5,3,1,4,1,1,1,1,3,3,3,2,5,5,3,1,1,1,1,1,4,1,1,3,3,1,1,4,1,3,5,5,5,3,5,5,3,4,4,1,4,1,3,1,1,1,1,
3,3,3,5,3,1,1,1,1,1,3,3,5,3,5,5,2,5,3,2,3,2,2,3,2,5,3,3,5,5,5,2,4,4,1,1,6,1,2,5,3,4,4,1,1,4,3,5,3,3,
3,5,3,5,3,5,3,5,5,5,2,5,3,2,5,3,3,5,3,1,1,1,1,1,3,5,3,5,2,3,2,5,5,2,4,4,4,3,4,4,4,3,3,5,3,5,3,5,3,3,
5,3,5,5,3,3,5,5,5,3,2,5,3,5,5,2,1,1,1,4,1,1,1,3,3,5,3,1,1,4,4,1,1,1,4,4,3,4,4,4,3,2,2,1,1,1,1,2,5,3,
4,1,1,1,1,3,5,3,5,3,5,5,2,3,3,5,3,1,1,4,4,4,2,5,2,5,3,3,2,4,1,1,4,1,3,5,5,3,2,3,5,5,3,2,3,3,3,5,5,3,
3,5,5,5,2,5,5,3,3,5,5,3,5,3,5,5,3,3,5,3,5,3,5,5,5,3,3,5,3,5,3,1,1,1,1,4,2,3,3,5,2,2,5,2,1,1,1,1,1,1,
2,2,1,1,1,4,1,3,3,5,3,5,5,3,3,3,5,2,5,2,5,5,5,2,3,3,5,5,3,5,3,5,3,3,3,4,4,4,2,5,5,3,5,3,3,5,3,3,5,3,
5,5,3,4,1,4,1,3,5,3,5,3,3,5,5,2,5,3,5,5,5,2,2,4,1,1,1,1,4,1,1,3,5,5,5,2,3,5,2,3,2,3,5,2,5,3,5,5,5,2,
5,2,5,3,3,5,5,2,5,2,5,3,1,1,1,1,3,5,5,5,2,1,4,4,1,3,3,5,3,1,1,1,1,3,3,3,3,3,5,3,5,5,3,3,3,4,4,4,3,3,
2,4,1,1,1,1,1,1,4,3,5,2,3,5,3,5,3,2,3,5,3,5,3,5,5,3,1,4,1,1,1,2,3,1,1,1,1,1,1,1,3,3,5,5,5,3,1,1,1,4,
3,5,2,1,1,1,4,3,3,2,5,3,3,3,5,3,5,3,5,3,5,3,5,2,5,3,3,5,5,5,3,5,3,2,4,4,4,3,2,5,3,1,1,1,1,1,1,2,3,5,
5,2,3,5,3,5,3,3,4,1,1,1,4,3,3,1,1,1,1,3,5,3,5,3,3,3,4,4,1,1,3,5,3,2,3,5,5,2,5,3,3,3,5,5,2,5,5,3,3,3,
5,3,5,5,5,3,2,3,5,3,3,3,5,3,3,3,5,3,5,3,3,3,5,5,5,3,5,3,3,3,1,1,6,1,3,5,5,5,3,3,2,3,5,3,5,3,5,5,5,3,
5,3,2,2,5,5,3,3,3,1,1,1,1,1,1,3,3,2,3,5,5,5,3,3,5,2,5,2,5,3,3,5,3,3,3,5,5,3,5,2,3,5,3,3,3,5,3,3,3,5,
3,3,5,3,2,3,3,2,1,4,1,1,4,3,3,3,5,5,5,2,3,5,3,2,3,3,5,5,2,2,5,3,3,3,3,3,3,2,2,2,5,3,2,3,3,3,3,1,1,1,
4,3,3,1,1,1,1,3,2,3,3,5,3,3,2,3,3,3,3,5,3,3,2,2,2,5,3,5,3,2,3,2,5,3,5,2,3,3,5,5,5,3,3,1,1,1,1,1,2,5,
3,5,5,3,5,3,3,5,5,3,3,5,5,5,2,3,2,3,3,3,3,3,5,3,5,3,2,5,3,1,1,1,1,3,2,5,3,5,5,3,3,3,5,2,5,5,3,5,3,3,
3,5,5,5,3,5,5,3,5,2,3,3,2,5,3,5,3,3,3,3,3,3,3,5,5,3,3,3,3,5,3,3,5,3,5,5,3,3,3,5,5,5,3,5,5,2,3,5,5,3,
5,5,3,1,1,1,1,3,5,5,2,5,3,5,5,3,3,3,5,3,3,1,1,1,1,1,3,5,3,3,5,3,3,2,2,3,5,5,5,3,5,3,1,4,1,1,3,2,3,5,
3,3,3,3,5,5,5,3,5,3,5,3,3,2,2,3,2,3,3,5,3,5,5,3,5,5,5,3,2,3,5,2,2,3,3,3,2,3,5,3,3,5,3,3,3,3,3,3,5,2,
2,3,5,3,3,5,3,2,3,3,3,3,5,3,3,3,5,2,5,5,5,3,3,5,5,3,3,2,5,3,5,3,3,5,3,5,3,5,5,3,5,5,3,5,5,2,3,3,3,3,
5,3,3,2,2,3,5,5,2,3,3,5,3,3,4,1,1,4,1,3,5,3,5,5,3,3,5,2,3,5,2,5,5,5,3,3,3,5,3,5,3,3,2,2,5,2,2,5,3,3,
5,5,5,3,3,3,3,5,5,3,3,1,1,1,1,3,5,5,2,3,5,5,3,5,5,3,5,3,3,4,1,1,4,3,5,3,5,5,3,5,3,2,5,3,1,1,1,1,1,2,
3,3,3,3,5,3,5,5,3,3,5,3,3,5,5,3,2,3,5,3,5,3,5,2,5,3,5,5,3,5,3,5,5,3,3,5,2,4,1,1,1,1,1,4,3,5,3,3,3,3,
4,4,4,3,3,3,3,5,5,3,5,3,5,3,3,3,3,1,1,1,4,3,5,3,1,1,1,4,3,3,3,5,3,5,2,5,5,3,5,3,3,5,5,3,3,5,5,3,5,5,
3,3,5,3,5,5,5,3,3,3,5,3,5,2,3,3,3,1,1,1,1,2,5,3,3,5,5,3,2,5,5,3,3,3,3,3,5,5,3,3,2,5,3,5,3,4,6,1,1,1,
1,1,3,3,5,5,3,5,5,3,5,2,5,3,5,5,5,3,2,3,5,3,2,5,3,1,1,1,4,1,6,1,2,5,3,2,5,3,5,5,5,3,5,5,3,5,3,3,3,3,
3,5,5,2,5,5,3,3,3,2,3,3,1,1,4,1,3,1,1,1,1,1,3,5,5,2,3,3,5,3,3,5,5,3,2,5,3,5,3,3,3,5,3,3,5,2,3,3,5,5,
3,3,2,3,3,3,3,3,2,5,5,3,5,5,5,3,3,3,5,3,5,2,5,5,5,3,3,5,3,3,5,5,5,3,5,5,5,3,3,2,3,3,3,3,3,3,3,3,5,5,
2,3,3,3,3,2,3,3,5,2,3,5,3,3,2,3,3,5,3,1,1,1,1,3,2,5,3,3,4,1,1,1,2,1,1,1,1,1,2,5,2,4,4,1,4,3,3,5,5,3,
5,5,3,3,5,3,5,5,2,5,2,1,1,4,4,3,5,5,3,5,5,2,5,5,3,3,5,5,3,3,2,2,3,5,2,3,5,3,3,5,5,5,3,5,5,3,3,3,3,3,
3,5,5,5,3,1,1,1,1,1,2,3,4,1,1,1,1,6,4,1,3,3,5,3,3,2,2,2,3,2,3,5,5,3,5,5,5,3,5,3,3,5,2,5,5,3,1,1,1,1,
3,3,5,5,3,5,3,3,3,5,5,3,5,3,5,2,5,2,3,3,5,3,1,1,1,1,1,3,5,5,3,5,3,4,1,6,1,6,4,2,3,5,2,2,5,5,2,5,3,3,
3,2,5,3,3,5,3,3,3,3,5,5,3,5,5,3,3,5,3,5,3,5,3,5,3,3,5,3,5,3,4,1,4,1,3,5,5,3,1,4,1,1,3,3,3,5,2,5,5,3,
5,3,2,3,3,5,3,2,3,3,2,2,5,3,4,1,1,1,3,3,3,3,3,2,3,2,1,1,6,1,3,3,3,3,5,3,5,3,5,3,2,3,2,5,5,5,2,3,5,3,
3,3,5,5,3,5,5,3,5,5,2,5,3,1,1,1,1,3,5,2,5,5,2,3,5,3,5,5,3,5,3,5,5,3,5,3,3,2,2,3,3,5,5,3,3,3,5,5,2,3,
2,5,5,3,5,3,5,3,5,5,5,3,3,2,5,3,1,1,1,4,1,4,3,2,1,4,6,6,1,3,3,3,5,2,5,3,3,1,1,1,1,2,5,5,3,3,5,2,3,5,
5,5,3,3,1,4,6,1,3,5,5,3,5,5,2,1,1,1,1,4,2,5,5,3,2,3,5,5,3,5,3,5,3,2,3,3,3,3,3,3,3,5,2,5,5,2,3,3,3,3,
5,3,3,3,3,3,2,3,5,3,3,3,5,3,3,5,3,3,3,2,4,4,4,3,2,3,3,2,5,3,5,3,2,5,3,2,5,3,5,3,5,3,1,1,1,1,3,1,4,1,
1,3,2,3,5,5,5,3,5,2,3,3,3,5,3,5,3,5,3,1,1,1,4,3,3,5,3,3,5,3,4,4,4,3,5,5,3,5,5,5,3,3,5,2,3,3,3,3,3,5,
2,2,1,6,1,1,4,3,5,3,5,5,2,3,3,6,1,1,6,2,3,5,3,3,3,3,5,3,3,3,2,1,4,1,6,3,5,5,3,5,3,3,5,5,3,3,3,5,5,3,
5,3,3,5,5,3,5,2,3,5,5,5,3,3,2,5,5,3,3,2,5,3,5,3,2,3,5,5,5,3,5,3,1,4,4,1,4,1,4,3,3,2,3,5,5,3,5,3,5,5,
5,3,5,3,3,4,1,1,1,1,3,3,5,2,1,1,6,1,2,5,5,5,2,5,3,5,3,4,4,4,2,5,5,2,3,3,3,5,5,5,3,5,3,3,1,4,1,1,6,1,
3,3,2,5,5,5,3,3,3,3,3,3,5,5,3,2,3,5,3,3,2,2,5,5,5,2,3,3,5,5,5,2,5,3,5,3,2,5,5,2,5,3,5,5,5,3,5,2,5,2,
5,2,3,1,6,1,1,4,1,1,3,3,5,3,5,5,3,1,1,1,1,1,3,3,5,2,3,3,2,1,1,1,1,1,1,1,4,3,3,3,3,5,5,3,3,3,3,5,5,3,
3,3,5,3,3,2,3,2,3,2,5,2,3,3,1,1,1,1,1,1,1,3,3,5,3,1,1,1,1,1,4,1,4,1,2,5,5,3,3,5,5,5,3,5,5,3,5,5,2,3,
5,3,3,5,3,3,5,5,3,2,2,3,2,5,5,2,5,2,5,5,5,3,5,5,3,2,5,3,5,3,3,1,1,4,1,3,1,1,4,4,2,2,5,5,5,3,3,5,5,5,
2,5,3,3,2,3,3,4,1,1,1,1,1,1,1,3,3,2,3,5,5,5,2,5,3,3,3,3,3,3,5,3,2,1,1,6,1,1,3,3,1,4,1,1,1,4,1,1,1,2,
5,3,5,5,3,5,3,3,5,5,2,3,5,2,5,3,2,5,2,5,5,5,2,3,3,3,2,5,5,2,3,1,1,1,1,2,5,3,3,2,5,3,5,5,2,2,1,1,1,1,
3,5,3,5,3,5,2,3,5,5,2,5,5,3,3,4,1,4,4,2,5,3,5,2,2,3,5,3,4,4,4,2,1,1,1,1,1,3,3,3,5,5,3,5,3,5,5,2,5,3,
5,5,3,3,2,5,3,3,5,3,1,1,1,1,3,6,1,4,1,3,5,5,3,5,5,5,3,3,2,5,5,2,5,5,3,5,3,3,3,3,3,2,3,3,3,2,3,3,3,3,
4,4,4,3,3,5,5,3,5,3,5,5,3,5,5,5,3,4,4,4,3,2,5,3,2,3,3,5,3,3,5,5,3,3,2,5,3,2,3,3,5,5,3,5,5,3,3,2,5,3,
3,3,3,3,3,5,5,3,3,5,2,5,2,3,5,5,3,3,2,3,5,2,3,3,5,3,2,2,5,3,5,3,3,5,3,5,3,5,3,5,3,2,3,3,2,3,3,5,5,2,
2,2,5,3,5,5,3,2,5,2,3,5,3,2,3,5,5,3,2,5,3,5,3,5,3,5,3,2,3,3,5,3,3,3,5,5,5,3,3,5,5,3,3,2,5,5,3,1,1,1,
1,1,4,1,1,3,1,1,1,1,2,5,2,5,2,5,5,3,3,3,3,5,3,2,2,2,1,1,1,1,3,1,4,1,1,1,1,1,1,1,1,3,5,3,2,3,2,3,1,1,
1,1,1,1,1,1,1,1,1,3,2,1,1,4,1,3,5,5,2,4,4,4,2,5,3,1,1,1,1,1,3,5,3,2,4,4,6,1,1,1,1,3,3,1,1,4,1,3,5,5,
3,1,1,1,1,3,1,1,6,4,1,2,5,5,3,5,3,5,5,2,3,3,1,1,4,1,2,3,5,5,3,5,3,5,5,5,2,3,3,5,3,2,5,3,3,3,3,3,3,2,
5,3,5,5,3,5,3,3,3,3,3,5,3,2,1,4,1,1,1,3,5,3,5,3,5,3,3,3,3,5,5,3,2,3,3,5,2,3,1,1,1,1,3,5,5,3,3,3,3,3,
5,5,2,5,2,5,3,5,3,3,3,3,3,3,3,3,3,5,3,2,5,5,2,3,2,3,3,3,3,5,3,5,3,5,5,3,5,5,3,2,5,5,2,3,5,3,3,3,5,5,
5,3,3,5,3,3,3,5,5,3,5,3,3,3,2,3,5,3,5,5,2,5,5,3,3,3,3,5,3,3,3,3,5,5,3,2,3,5,5,3,5,3,3,3,6,1,1,4,3,3,
5,2,3,3,4,1,1,1,1,2,5,3,2,5,3,3,3,5,5,3,3,5,2,3,3,3,3,5,3,5,3,5,5,3,5,5,5,3,3,5,3,3,5,3,5,3,3,2,5,3,
5,3,3,5,3,5,3,3,5,3,5,5,3,3,5,2,3,3,1,1,1,1,1,3,5,5,2,2,5,3,3,3,3,3,3,5,5,3,3,3,4,4,4,3,3,5,5,5,2,3,
5,2,5,3,5,2,2,3,5,3,3,5,5,3,5,5,2,3,3,5,3,3,3,5,2,3,3,3,5,3,3,3,5,3,1,1,1,1,3,5,2,3,3,3,2,5,3,2,5,3,
5,3,3,2,3,3,3,5,3,3,2,2,3,1,1,6,1,3,5,5,2,2,5,5,2,5,3,5,3,5,5,3,3,5,2,5,5,3,3,3,3,5,3,2,2,3,3,2,5,3,
5,3,5,5,5,3,3,5,3,3,3,3,2,3,5,5,3,3,5,2,3,3,3,5,3,3,3,5,5,3,5,2,5,2,3,5,5,3,3,2,6,1,1,1,2,2,3,3,3,2,
3,3,5,3,3,5,3,2,3,5,3,5,5,3,3,5,5,3,5,5,2,5,5,2,5,3,5,5,5,3,5,3,3,5,3,5,5,3,3,5,3,3,5,3,5,5,5,3,2,3,
3,3,3,3,3,2,5,3,3,5,5,5,3,3,3,2,5,3,2,3,3,3,2,3,3,5,5,3,5,3,5,3,2,5,2,5,5,2,3,5,2,3,5,2,5,5,3,3,3,5,
3,3,3,5,5,3,3,2,3,3,3,5,5,3,2,3,5,5,3,1,1,1,1,2,5,5,3,1,1,1,1,1,3,3,3,5,5,3,3,5,3,3,3,3,1,1,1,1,3,2,
3,2,3,3,5,3,5,3,2,2,5,3,3,5,3,3,5,3,3,1,1,1,1,2,3,5,2,2,5,3,3,5,5,3,5,5,3,2,5,5,5,3,3,5,3,3,3,5,5,5,
3,3,3,3,5,3,5,3,5,3,1,1,1,1,6,3,5,5,5,3,5,5,5,2,3,3,5,2,5,5,5,3,5,2,3,2,3,3,5,5,3,3,5,5,3,2,5,3,2,3,
3,2,5,3,5,5,5,3,3,3,2,3,5,5,3,3,3,3,5,5,3,5,5,2,2,5,3,5,5,3,5,3,3,3,5,3,5,5,5,3,3,2,2,3,5,3,2,5,3,5,
2,5,5,3,3,5,3,3,5,3,2,5,3,3,2,5,5,2,3,3,2,3,5,3,3,3,3,5,5,5,3,3,5,2,5,5,3,2,3,5,3,5,3,3,2,5,3,5,3,3,
5,5,3,3,3,5,3,3,5,3,3,3,3,3,3,5,2,3,5,5,2,5,3,3,3,2,5,3,3,5,3,3,2,3,3,5,3,2,3,3,5,5,2,3,3,3,2,5,3,3,
3,3,5,3,5,5,3,5,2,3,2,5,5,3,3,3,5,2,3,5,5,3,5,3,5,3,5,3,3,5,5,3,3,3,3,5,3,2,3,2,5,3,5,5,2,5,3,2,2,3,
5,3,3,5,5,3,3,3,5,5,3,5,3,2,1,1,1,1,1,2,3,3,3,3,3,3,5,3,3,3,3,3,2,3,5,3,3,5,3,5,3,3,3,2,5,3,3,2,3,5,
5,5,3,3,3,5,2,3,5,5,3,3,5,3,5,5,5,2,3,5,3,5,3,3,5,2,5,3,3,5,3,3,2,3,3,5,3,5,5,3,2,3,3,5,5,3,3,3,2,2,
5,3,5,3,5,3,5,3,1,1,1,1,3,5,2,2,5,3,5,3,3,2,5,3,3,3,3,3,3,2,2,2,2,5,5,3,3,3,3,2,3,1,1,1,1,2,5,5,3,5,
5,2,3,3,5,3,5,3,3,5,3,5,3,2,3,5,5,5,2,3,3,4,4,4,3,1,1,1,1,3,4,1,1,1,2,5,3,5,3,5,5,5,2,5,2,3,3,5,5,3,
5,3,5,5,2,5,3,3,4,4,4,3,2,2,3,2,3,3,3,5,5,3,5,3,3,5,5,3,3,5,2,3,5,2,1,1,1,1,3,3,3,5,5,2,5,3,5,3,3,3,
5,5,5,3,5,5,3,3,3,3,5,3,5,5,3,5,5,3,3,5,5,3,5,5,2,3,1,1,1,1,3,5,3,5,2,3,2,5,2,2,3,3,6,1,6,1,4,3,3,3,
5,3,5,3,5,3,2,3,5,3,3,5,3,5,3,3,5,5,3,3,3,3,5,3,5,3,3,3,3,3,5,3,5,3,5,3,4,4,4,3,3,3,3,5,3,3,3,3,3,3,
3,2,5,3,5,5,2,3,5,5,3,3,5,5,3,3,5,3,3,5,3,3,3,3,3,2,3,5,3,3,2,5,5,3,5,2,3,5,5,5,3,5,3,3,5,5,2,3,5,5,
2,1,1,1,1,1,1,3,3,5,5,3,3,4,4,4,3,5,3,5,5,2,5,3,3,2,5,3,2,3,5,3,3,2,3,2,3,3,3,2,2,5,3,3,5,5,5,3,5,5,
5,2,5,2,5,2,5,2,3,5,2,5,5,2,3,2,2,5,3,1,1,1,1,3,3,5,3,5,5,3,5,3,3,3,3,3,3,3,3,3,5,3,3,3,5,5,3,3,3,5,
5,2,3,3,3,2,3,3,5,5,3,2,3,1,4,1,4,3,5,5,3,5,5,5,3,5,5,2,2,2,3,3,3,5,2,5,3,2,3,5,5,3,3,5,3,3,3,3,5,5,
3,3,2,5,3,5,2,2,3,5,3,5,3,5,3,3,3,5,3,3,3,3,3,3,2,3,5,3,3,5,2,5,3,3,3,2,3,1,1,1,1,3,2,2,3,3,3,2,5,2,
5,3,3,5,3,1,1,1,1,3,5,3,3,2,2,5,3,5,3,3,3,3,3,5,5,3,3,5,3,3,3,3,2,5,3,5,2,2,2,2,3,3,5,2,5,3,5,3,3,5,
3,3,2,5,2,3,3,5,3,5,5,2,3,3,5,3,5,3,3,3,3,3,3,5,5,3,2,2,2,2,2,1,4,1,1,1,1,1,2,3,5,3,3,5,3,3,3,3,3,5,
5,5,3,5,3,3,3,5,5,3,3,2,5,5,3,5,5,3,3,3,3,5,3,5,3,5,3,5,3,5,5,3,2,5,2,5,5,3,5,5,2,3,2,5,5,3,3,2,2,5,
3,5,3,3,5,3,5,3,2,3,3,3,3,1,1,1,4,3,5,5,3,5,5,2,2,3,3,5,5,2,2,3,5,5,3,3,5,2,5,5,3,3,3,5,3,3,3,3,3,3,
3,5,3,3,3,3,3,5,3,3,5,2,3,5,2,3,5,5,5,3,5,5,5,3,5,3,3,5,5,5,2,3,3,2,5,3,5,3,3,5,5,3,3,3,3,3,3,2,3,5,
3,5,2,5,5,3,5,3,5,3,3,5,2,5,5,3,3,3,3,5,3,3,3,3,3,2,2,5,5,3,3,5,5,3,3,1,1,1,1,2,3,5,3,3,5,3,5,5,2,3,
5,5,3,3,5,3,3,3,3,3,3,3,5,3,3,3,3,5,5,5,3,3,3,5,3,3,5,3,5,2,3,3,5,5,3,3,5,2,3,3,2,3,3,3,3,5,5,3,2,3,
3,3,3,5,3,3,2,5,5,3,5,3,5,3,1,1,1,1,2,3,3,2,3,2,5,2,5,2,5,2,5,2,3,2,3,2,3,2,3,3,5,3,5,2,3,3,3,3,5,3,
5,3,5,3,3,3,5,5,3,2,5,3,5,3,3,3,5,2,5,3,3,5,3,2,4,1,1,1,1,1,1,1,1,1,4,3,5,5,3,5,5,5,2,2,2,5,5,3,3,3,
3,3,3,3,3,5,5,3,3,2,3,5,2,5,5,3,2,3,5,5,5,3,1,1,1,1,1,1,1,2,5,5,5,3,5,3,1,4,1,1,1,1,2,3,3,5,5,3,3,2,
5,5,3,3,3,2,3,2,3,5,3,3,2,3,2,3,3,5,5,5,3,5,5,5,3,2,5,5,5,2,3,3,3,3,3,1,1,1,4,2,3,5,3,2,5,3,3,3,2,3,
3,3,5,3,3,3,3,5,5,3,3,3,3,3,2,2,5,3,3,3,5,2,3,3,2,3,5,5,2,5,5,3,3,5,3,3,5,5,3,3,3,3,2,5,3,5,5,2,5,3,
3,3,5,5,2,2,2,3,3,3,5,3,3,3,2,5,2,5,5,3,5,5,3,2,5,3,5,3,3,2,3,3,5,3,5,3,2,3,3,3,3,3,
Вторичная структура (Пикотех-2017) весит около 6 мегабайт в стандарте PNG: https://cloud.mail.ru/public/KKjH/goMgmMGAn
На яндекс-фотки не грузится. Но самый интересный фрагмент удалось вырезать в программе Paint:
Сиреневым цветом отмечен виток необычной метиониновой спирали. В отличие от альфа-спирали у неё более длинный шаг. Метиониновая спираль отличается от обычной альфа-спирали примерно как резьба с более крупным шагом от резьбы с менее крупным шагом:
Шаг метиониновой спирали примерно на 25% крупнее шага обычной альфа-спирали. При этом на один виток приходится тоже 3.6...3.7 аминокислотных остатка Met.

Ещё один интересный участок этого белка 3916...4164. Он содержит все типы композиционных кодов: 1,2,3,4, но ... не содержит ни одного витка спирали! Интересно оценить вероятность случайной комбинации композиционных кодов такого типа. Напомню, что спираль возникает при трёхкратном (или более) повторении кодов 1, 4 или их комбинации. При этом третий код должен быть "4", либо спиральных кодов (1,4), идущих подряд, должно быть больше 3.

Белковая молекула кончается витком полилизиновой пи-спирали, похожей на винт Архимеда.
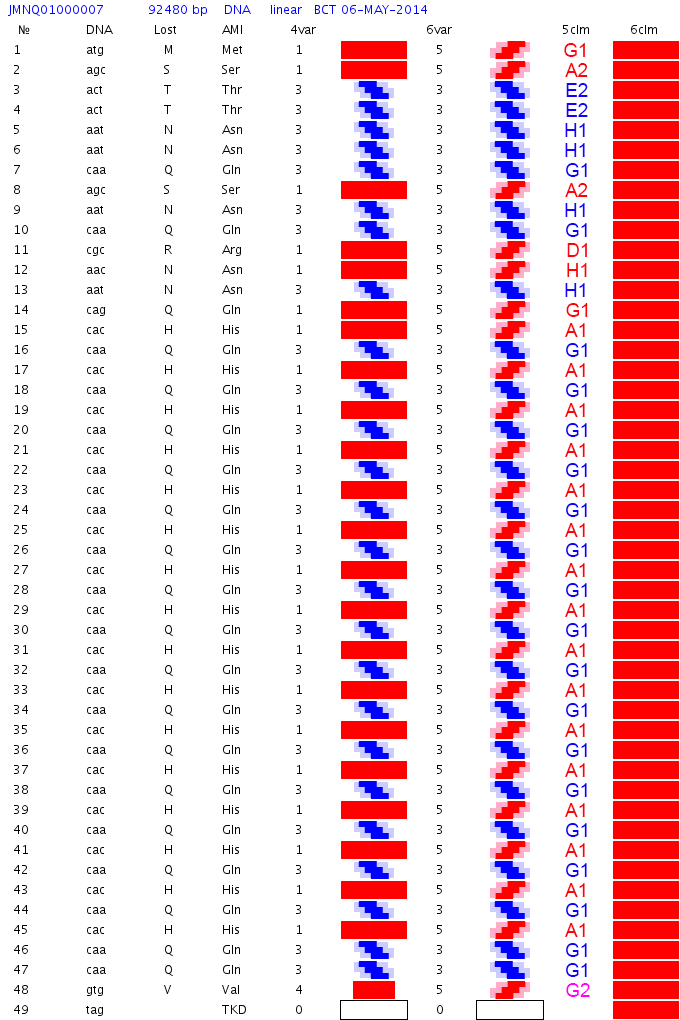
2D диаграммы Пикотех белковых структур, исследованных методом РСА, версия 2017 года 6var.
СТРУКТУРА ДИАГРАММЫ ПИКОТЕХ
СОКРАЩЁННАЯ ДИАГРАММА
Красный - альфа-спираль.
Оранжевый - 310-спираль.
Розовый - одиночный код альфа/310 спиралей.
Голубой - пи-спираль.
Зеленый - бета-спираль.
Сиреневый - метиониновая спираль. У неё более крупный шаг "резьбы", чем у обычной альфа-спирали.
Черный в сокращённом представлении и белый в развернутом означают либо неизвестный код, либо конец трансляции.
Циклическое повторение цветов - программная спираль.
ПОЛНАЯ ДИАГРАММА
Содержание столбцов
1 - порядковый номер аминокислотного остатка в белковой молекуле
2 - триплетный код
3 - однобуквенный код аминокислотного остатка
4 - трёхбуквенное обозначение аминокислотного остатка
5 - упрощённый композиционный код
6 - графическая интерпретация упрощенного композиционного кода
7 - композиционный код
8 - графическая интерпретация композиционного кода
9 - нота, которая звучит при установке данной аминокислоты в растущую белковую цепь
10 - графическое изображение ноты (или ударного инструмента)
Для новой версии Пикотех 2018 разработан Композиционный код 7var
Программные спирали - повторение последовательности композиций. Например, один код альфа-спирали, затем один код пи-спирали. n(35) задаёт программную спираль, а n3 или n5 - простые спирали (пи-спираль и альфа-310-спираль).
1111111111111111111 - прямая альфа-спираль
4444444444444444444 - прямая 310-спираль
3333333333333333333 - прямая пи-спираль
2222222222222222222 - прямая бета-спираль
232323 - программная 23-спираль
141414 - программная 14-спираль
1.
https://www.ncbi.nlm.nih.gov/Structure/ … p;uid=5TUD
Structural insights into the extracellular recognition of the human serotonin 2B receptor by an antibody
Ishchenko A, Wacker D, Kapoor M, Zhang A, Han GW, Basu S, Patel N, Messerschmidt M, Weierstall U, Liu W, Katritch V, Roth BL, Stevens RC, Cherezov V
https://www.ebi.ac.uk/ena/data/view/CAA … play=fasta
>ENA|CAA54513|CAA54513.1 Homo sapiens (human) 5-HT2B serotonin receptor
ATGGCTCTCTCTTACAGAGTGTCTGAACTTCAAAGCACAATTCCTGAGCACATTTTGCAG
AGCACCTTTGTTCACGTTATCTCTTCTAACTGGTCTGGATTACAGACAGAATCAATACCA
GAGGAAATGAAACAGATTGTTGAGGAACAGGGAAATAAACTGCACTGGGCAGCTCTTCTG
ATACTCATGGTGATAATACCCACAATTGGTGGAAATACCCTTGTTATTCTGGCTGTTTCA
CTGGAGAAGAAGCTGCAGTATGCTACTAATTACTTTCTAATGTCCTTGGCGGTGGCTGAT
TTGCTGGTTGGATTGTTTGTGATGCCAATTGCCCTCTTGACAATAATGTTTGAGGCTATG
TGGCCCCTCCCACTTGTTCTATGTCCTGCCTGGTTATTTCTTGACGTTCTCTTTTCAACC
GCATCCATCATGCATCTCTGTGCCATTTCAGTGGATCGTTACATAGCCATCAAAAAGCCA
ATCCAGGCCAATCAATATAACTCACGGGCTACAGCATTCATCAAGATTACAGTGGTGTGG
TTAATTTCAATAGGCATTGCCATTCCAGTCCCTATTAAAGGGATAGAGACTGATGTGGAC
AACCCAAACAATATCACTTGTGTGCTGACAAAGGAACGTTTTGGCGATTTCATGCTCTTT
GGCTCACTGGCTGCCTTCTTCACACCTCTTGCAATTATGATTGTCACCTACTTTCTCACT
ATCCATGCTTTACAGAAGAAGGCTTACTTAGTCAAAAACAAGCCACCTCAACGCCTAACA
TGGTTGACTGTGTCTACAGTTTTCCAAAGGGATGAAACACCTTGCTCGTCACCGGAAAAG
GTGGCAATGCTGGATGGTTCTCGAAAGGACAAGGCTCTGCCCAACTCAGGTGATGAAACA
CTTATGCGAAGAACATCCACAATTGGGAAAAAGTCAGTGCAGACCATTTCCAACGAACAG
AGAGCCTCAAAGGTCCTAGGGATTGTGTTTTTCCTCTTTTTGCTTATGTGGTGTCCCTTC
TTTATTACAAATATAACTTTAGTTTTATGTGATTCCTGTAACCAAACTACTCTCCAAATG
CTCCTGGAGATATTTGTGTGGATAGGCTATGTTTCCTCAGGAGTGAATCCTTTGGTCTAC
ACCCTCTTCAATAAGACATTTCGGGATGCATTTGGCCGATATATCACCTGCAATTACCGG
GCCACAAAGTCAGTAAAAACTCTCAGAAAACGCTCCAGTAAGATCTACTTCCGGAATCCA
ATGGCAGAGAACTCTAAGTTTTTCAAGAAACATGGAATTCGAAATGGGATTAACCCTGCC
ATGTACCAGAGTCCAATGAGGCTCCGAAGTTCAACCATTCAGTCTTCATCAATCATTCTA
CTAGATACGCTTCTCCTCACTGAAAATGAAGGTGACAAAACTGAAGAGCAAGTTAGTTAT
GTATAG
Программа Пикотех показывает следующую детализированную структуру.
Вторичная структура белка начинается с программной 35-спирали длиной 8 аминокислотных остатков. 8-ой остаток Ser является последним остатком программной 35-спирали и первым остатком пи-спирали, которая состоит из 4-х аминокислотных остатков. Далее идёт последовательность композиционных кодов 5233553, а за ней один виток альфа-спирали: 1111, образованный аминокислотными остатками LQST. Остатки SIP под номерами 38,39,40 образуют виток бета-спирали. Затем последовательность EEMKQI образует примерно половину витка программной 35-спирали. Остатки с 81 по 86 образуют гибридную альфа-310-спираль. Далее 87-90 идет пи-спираль. 94-98 - 310-спираль (полтора витка). 120-123 - один виток альфа-спирали. Участок 124-334 структурирован одиночными композиционными кодами всех типов. 335-341 - программная 355-спираль. 342-346 - программная 23-спираль. 346-351 - пи-спираль (примерно полтора витка). 360-363 - один виток 310-альфа-спирали. 378-383 - прямая альфа-спираль (полтора витка). 414-418 - альфа-310-спираль (примерно 1.3 витка).
431-435 - программная 23-спираль. 440-443 - гнутая метионином альфа-спираль (примерно 1.2 витка). 467-471 пи-спираль (примерно 1.2 витка). 469-480 - программная 3335-спираль.
2.
https://www.ncbi.nlm.nih.gov/Structure/pdb/5V56
Crystal structure of a multi-domain human smoothened receptor in complex with a super stabilizing ligand
Zhang X, Zhao F, Wu Y, Yang J, Han GW, Zhao S, Ishchenko A, Ye L, Lin X, Ding K, Dharmarajan V, Griffin PR, Gati C, Nelson G, Hunter MS, Hanson MA, Cherezov V, Stevens RC, Tan W, Tao H, Xu F
http://www.rcsb.org/pdb/explore/explore … ureId=5V56
https://www.ebi.ac.uk/ena/data/view/AAA … play=fasta
>ENA|AAA23367|AAA23367.1 Desulfovibrio vulgaris hypothetical protein
ATGCCCAAAGCCCTCATCGTCTACGGTTCCACCACAGGCAACACGGAATACACCGCCGAA
ACCATCGCACGTGAACTTGCCGATGCAGGGTACGAAGTCGATAGCCGGGACGCGGCCTCT
GTCGAGGCTGGCGGTCTCTTCGAAGGCTTCGACCTCGTCCTTCTCGGATGCTCGACGTGG
GGTGACGACTCCATCGAACTGCAGGACGACTTCATTCCCCTTTTCGACTCCCTCGAAGAG
ACGGGGGCGCAGGGCCGCAAGGTGGCCTGCTTCGGCTGCGGCGACAGTTCCTACGAGTAC
TTCTGCGGGGCTGTCGACGCCATCGAAGAGAAGCTCAAGAACCTCGGTGCCGAAATCGTT
CAGGACGGTCTTCGCATCGATGGCGACCCCCGCGCCGCCCGGGACGACATCGTCGGCTGG
GCGCATGACGTGAGGGGCGCCATCTAG
Результаты программы Пикотех
4-8 - альфа-спираль
49-53 - альфа-спираль
57-60 - альфа-спираль
62-65 - альфа-спираль
67-71 - альфа-спираль
75-78 - альфа-спираль
80-95 - альфа-спираль
97-103 - альфа-спираль
105-108 - альфа-спираль
110-115 - альфа-спираль
115-121 - программная 35-спираль
120-127 - программная 3355-спираль
128-141 - альфа-спираль
143-148 - альфа-спираль
3.
https://www.ncbi.nlm.nih.gov/Structure/pdb/5W0P
Identification of Phosphorylation Codes for Arrestin Recruitment by G Protein-Coupled Receptors
Zhou XE, He Y, de Waal PW, Gao X, Kang Y, Van Eps N, Yin Y, Pal K, Goswami D, White TA, Barty A, Latorraca NR, Chapman HN, Hubbell WL, Dror RO, Stevens RC, Cherezov V, Gurevich VV, Griffin PR, Ernst OP, Melcher K, Xu HE
https://www.ebi.ac.uk/ena/data/view/CAD … play=fasta
>ENA|CAD97623|CAD97623.1 Homo sapiens (human) hypothetical protein
ATGAATGGCACAGAAGGCCCTAACTTCTACGTGCCCTTCTCCAATGCGACGGGTGTGGTA
CGCAGCCCCTTCGAGTACCCACAGTACTACCTGGCTGAGCCATGGCAGTTCTCCATGCTG
GCCGCCTACATGTTTCTGCTGATCGTGCTGGGCTTCCCCATCAACTTCCTCACGCTCTAC
GTCACCGTCCAGCACAAGAAGCTGCGCACGCCTCTCAACTACATCCTGCTCAACCTAGCC
GTGGCTGACCTCTTCATGGTCCTAGGTGGCTTCACCAGCACCCTCTACACCTCTCTGCAT
GGATACTTCGTCTTCGGGCCCACAGGATGCAATTTGGAGGGCTTCTTTGCCACCCTGGGC
GGTGAAATTGCCCTGTGGTCCTTGGTGGTCCTGGCCATCGAGCGGTACGTGGTGGTGTGT
AAGCCCATGAGCAACTTCCGCTTCGGGGAGAACCATGCCATCATGGGCGTTGCCTTCACC
TGGGTCATGGCGCTGGCCTGCGCCGCACCCCCACTCGCCGGCTGGTCCAGGTACATCCCC
GAGGGCCTGCAGTGCTCGTGTGGAATCGACTACTACACGCTCAAGCCGGAGGTCAACAAC
GAGTCTTTTGTCATCTACATGTTCGTGGTCCACTTCACCATCCCCATGATTATCATCTTT
TTCTGCTATGGGCAGCTCGTCTTCACCGTCAAGGAGGCCGCTGCCCAGCAGCAGGAGTCA
GCCACCACACAGAAGGCAGAGAAGGAGGTCACCCGCATGGTCATCATCATGGTCATCGCT
TTCCTGATCTGCTGGGTGCCCTACGCCAGCGTGGCATTCTACATCTTCACCCACCAGGGC
TCCAACTTCGGTCCCATCTTCATGACCATCCCAGCGTTCTTTGCCAAGAGCGCCGCCATC
TACAACCCTGTCATCTATATCATGATGAACAAGCAGTTCCGGAACTGCATGCTCACCACC
ATCTGCTGCGGCAAGAACCCACTGGGTGACGATGAGGCCTCTGCTACCGTGTCCAAGACG
GAGACGAGCCAGGTGGCCCCGGCCTAA
Программа Пикотех показывает
8-14 - альфа-спираль.
21-26 - альфа-спираль.
28-31 - альфа-спираль.
35-44 - альфа-спираль.
46-70 - 310-альфа-спираль.
72-78 - альфа-спираль.
83-87 - альфа-спираль.
90-97 - альфа-спираль.
102-107 - альфа-спираль.
112-115 - альфа-спираль.
117-120 - альфа-спираль.
124-139 - альфа-спираль.
141-151 - альфа-спираль.
153-156 - альфа-спираль.
158-168 - альфа-спираль.
172-186 - альфа-спираль.
189-201 - альфа-спираль.
204-216 - альфа-спираль.
215-225 - программная 355-спираль.
224-233 - альфа-спираль.
235-239 - альфа-спираль.
238-248 - программная 255-спираль.
247-259 - альфа-спираль.
261-271 - альфа-спираль.
273-283 - альфа-спираль.
285-290 - альфа-спираль.
295-302 - альфа-спираль.
307-326 - альфа-спираль.
336-348 - альфа-спираль.
4.
https://www.ncbi.nlm.nih.gov/Structure/pdb/5XEZ
Structure of the full-length glucagon class B G-protein-coupled receptor
Zhang H, Qiao A, Yang D, Yang L, Dai A, de Graaf C, Reedtz-Runge S, Dharmarajan V, Zhang H, Han GW, Grant TD, Sierra RG, Weierstall U, Nelson G, Liu W, Wu Y, Ma L, Cai X, Lin G, Wu X, Geng Z, Dong Y, Song G, Griffin PR, Lau J, Cherezov V, Yang H, Hanson MA, Stevens RC, Zhao Q, Jiang H, Wang MW, Wu B
http://www.rcsb.org/pdb/explore/explore … ureId=5XEZ
http://www.uniprot.org/uniprot/P47871
https://www.ebi.ac.uk/ena/data/view/AAC … play=fasta
>ENA|AAC52063|AAC52063.1 Homo sapiens (human) glucagon receptor
ATGCCCCCCTGCCAGCCACAGCGACCCCTGCTGCTGTTGCTGCTGCTGCTGGCCTGCCAG
CCACAGGTCCCCTCCGCTCAGGTGATGGACTTCCTGTTTGAGAAGTGGAAGCTCTACGGT
GACCAGTGTCACCACAACCTGAGCCTGCTGCCCCCTCCCACGGAGCTGGTGTGCAACAGA
ACCTTCGACAAGTATTCCTGCTGGCCGGACACCCCCGCCAATACCACGGCCAACATCTCC
TGCCCCTGGTACCTGCCTTGGCACCACAAAGTGCAACACCGCTTCGTGTTCAAGAGATGC
GGGCCCGACGGTCAGTGGGTGCGTGGACCCCGGGGGCAGCCTTGGCGTGATGCCTCCCAG
TGCCAGATGGATGGCGAGGAGATTGAGGTCCAGAAGGAGGTGGCCAAGATGTACAGCAGC
TTCCAGGTGATGTACACAGTGGGCTACAGCCTGTCCCTGGGGGCGCTGCTCCTCGCCTTG
GCCATCCTGGGGGGCCTCAGCAAGCTGCACTGCACCCGCAATGCCATCCACGCGAATCTG
TTTGCGTCCTTCGTGCTGAAAGCCAGCTCCGTGCTGGTCATTGATGGGCTGCTCAGGACC
CGCTACAGCCAGAAAATTGGCGACGACCTCAGTGTCAGCACCTGGCTCAGTGATGGAGCG
GTGGCTGGCTGCCGTGTGGCCGCGGTGTTCATGCAATATGGCATCGTGGCCAACTACTGC
TGGCTGCTGGTGGAGGGCCTGTACCTGCACAACCTGCTGGGCCTGGCCACCCTCCCCGAG
AGGAGCTTCTTCAGCCTCTACCTGGGCATCGGCTGGGGTGCCCCCATGCTGTTCGTCGTC
CCCTGGGCAGTGGTCAAGTGTCTGTTCGAGAACGTCCAGTGCTGGACCAGCAATGACAAC
ATGGGCTTCTGGTGGATCCTGCGGTTCCCCGTCTTCCTGGCCATCCTGATCAACTTCTTC
ATCTTCGTCCGCATCGTTCAGCTGCTCGTGGCCAAGCTGCGGGCACGGCAGATGCACCAC
ACAGACTACAAGTTCCGGCTGGCCAAGTCCACGCTGACCCTCATCCCTCTGCTGGGCGTC
CACGAAGTGGTCTTCGCCTTCGTGACGGACGAGCACGCCCAGGGCACCCTGCGCTCCGCC
AAGCTCTTCTTCGACCTCTTCCTCAGCTCCTTCCAGGGCCTGCTGGTGGCTGTCCTCTAC
TGCTTCCTCAACAAGGAGGTGCAGTCGGAGCTGCGGCGGCGTTGGCACCGCTGGCGCCTG
GGCAAAGTGCTATGGGAGGAGCGGAACACCAGCAACCACAGGGCCTCATCTTCGCCCGGC
CACGGCCCTCCCAGCAAGGAGCTGCAGTTTGGGAGGGGTGGTGGCAGCCAGGATTCATCT
GCGGAGACCCCCTTGGCTGGTGGCCTCCCTAGATTGGCTGAGAGCCCCTTCTGA
Программа Пикотех показывает
147-173 - наиболее протяженный участок альфа-310-спирали.
Весь белок представлен преимущественно участками альфа-310-спиралей.
5.
https://www.ncbi.nlm.nih.gov/pubmed/17962520
https://www.ncbi.nlm.nih.gov/Structure/ … ;uid=60314
High-resolution crystal structure of an engineered human beta2-adrenergic G protein-coupled receptor.
Cherezov V1, Rosenbaum DM, Hanson MA, Rasmussen SG, Thian FS, Kobilka TS, Choi HJ, Kuhn P, Weis WI, Kobilka BK, Stevens RC.
https://www.rcsb.org/pdb/explore/explor … ureId=2RH1
http://www.uniprot.org/uniprot/P00720
https://www.ebi.ac.uk/ena/data/view/CAA28212
>ENA|CAA28212|CAA28212.1 Enterobacteria phage T4 hypothetical protein
ATGAATATATTTGAAATGTTACGTATAGATGAACGTCTTAGACTTAAAATCTATAAAGAC
ACAGAAGGCTATTACACTATTGGCATCGGTCATTTGCTTACAAAAAGTCCATCACTTAAT
GCTGCTAAATCTGAATTAGATAAAGCTATTGGGCGTAATTGCAATGGTGTAATTACAAAA
GATGAGGCTGAAAAACTCTTTAATCAGGATGTTGATGCTGCTGTTCGCGGAATTCTGAGA
AATGCTAAATTAAAACCGGTTTATGATTCTCTTGATGCGGTTCGTCGCTGTGCATTGATT
AATATGGTTTTCCAAATGGGAGAAACCGGTGTGGCAGGATTTACTAACTCTTTACGTATG
CTTCAACAAAAACGCTGGGATGAAGCAGCAGTTAACTTAGCTAAAAGTATATGGTATAAT
CAAACACCTAATCGCGCAAAACGAGTCATTACAACGTTTAGAACTGGCACTTGGGACGCG
TATAAAAATCTATAA
Версия 2016 года -6var
Версия 2018 года - 7var
Темно-синим цветом показаны пи-спирали, бирюзовым - одиночные АО попи-коду. Белок состоит преимущественно из пи-спиралей.
Этот пример демонстрирует, что РСА не от личает пи- от 310- спиралей.
Фолдинг есть, но на уровне третичной структуры белка. На уровне вторичной структуры его нет. Программная аллотропия белков опровергает гипотезу фолдинга
Технология трансляции генетического кода в структуру белка
Высоко периодичные структуры белков
Письмо, которое нужно разослать по лабораториям РСА
В лаборатории Наномир создана новая технология определения структуры белковых молекул, которая работает примерно в миллиард раз быстрее РСА. Наша задача в кратчайшие сроки проверить новую технологию, т.к. от её внедрения человечество может получить фантастический эффект.
Проще всего проверить новую технологию на простых структурах типа поливалина:
https://img-fotki.yandex.ru/get/509292/ … 5_orig.png
ETC64880: https://www.ncbi.nlm.nih.gov/nuccore/564568708
OXS06857: https://www.ncbi.nlm.nih.gov/nuccore/1226034760
KOF67455: https://www.ncbi.nlm.nih.gov/nuccore/918287724
OUM69688: https://www.ncbi.nlm.nih.gov/nuccore/1198313040
PIS11988: https://www.ncbi.nlm.nih.gov/nuccore/1277217132
KOF75295: https://www.ncbi.nlm.nih.gov/nuccore/918302080
OLP77457: https://www.ncbi.nlm.nih.gov/nuccore/1129165616
KYM86846: https://www.ncbi.nlm.nih.gov/nuccore/1009361734?&fmt_mask=65536
OTF86159: https://www.ncbi.nlm.nih.gov/nuccore/CM007906.1?report=fasta&sat=37&satkey=307832265&itemID=1362
KKF17324: https://www.ncbi.nlm.nih.gov/nuccore/808866584?&fmt_mask=65536
XP_002382247: https://www.ncbi.nlm.nih.gov/nuccore/238502026
OLP98873: https://www.ncbi.nlm.nih.gov/nuccore/1129194414
CDW76368: https://www.ncbi.nlm.nih.gov/nuccore/678335661
AHB99005: https://www.ncbi.nlm.nih.gov/nuccore/564747871?&fmt_mask=65536
OAQ27448: https://www.ncbi.nlm.nih.gov/nuccore/1032646064
OUM69730: https://www.ncbi.nlm.nih.gov/nuccore/1198313040
OGL53990: https://www.ncbi.nlm.nih.gov/nuccore/1084158629
EIE92391: https://www.ncbi.nlm.nih.gov/nuccore/76151980
XP_001590524: https://www.ncbi.nlm.nih.gov/nuccore/156049114
OTG33229: https://www.ncbi.nlm.nih.gov/nuccore/CM007891.1?from=7600734&to=7601591&sat=37&sat_key=307832447
OLQ06943: https://www.ncbi.nlm.nih.gov/nuccore/1129204435
XP_001895277: https://www.ncbi.nlm.nih.gov/nuccore/170580473
XP_001584576: https://www.ncbi.nlm.nih.gov/nuccore/156030497
KOF84884: https://www.ncbi.nlm.nih.gov/nuccore/918315370
Возможно ли методом РСА определить структуру этих участков белковых молекул? Если да, то нужен прайс.
С уважением, Руководитель лаборатории Наномир, Александр Кушелев
Кушелев Александр Юрьевич: Мы с Викторией соколик стали обсуждать фолдинг, и тут выяснилось, что она перепутала углы поворота транспортной РНК и белковой спирали
Отсюда и странные углы спиралей...
2017.12.08 11:31:07
активное втюхивание того, что может сделать программа Кушелева (2D-схему белка), вместо того, что хотят видеть заказчики 3D-модель белка, не приведёт к желаемому результату.
Кушелев: Тут есть ряд нюансов. Во-первых, нужно объяснить всем, кто хочет видеть 3D-модели белков, что рентгеноструктурный анализ (РСА) в действительности не может достоверно показать даже вторичную структуру белка. Что касается третичной и четвертичной, то РСА показывает их условно, т.е. взаимное расположение неких крупных ассоциаций, например, спиральных участков. При этом РСА часто не может отличить разные типы спиралей, например, пи-спираль от альфа-спирали, альфа-спираль от 310-спирали. А большинство спиральных участков гибридные, т.е. представлены чередованием композиций альфа- и 310-спиралей. С помощью РСА об этом не удалось узнать за многие десятилетия исследований. Поэтому доверие к интерпретации данных РСА держится на недостатке знаний в этой области, т.е. грубо говоря на невежестве профессионалов по молекулярной биологии
Во-вторых, программа Пикотех 3D позволяет показать и проконтролировать достоверность 3D моделей для некоторых участков белковых молекул. Например, для циклов, замкнутых через дисульфидные мостики. Речь идёт о циклах лизоцимов, где методом структурного анализа надежно установлены факты замыкания циклов через дисульфидные мостики:

http://nanoworld88.narod.ru/data/285.htm
http://nanoworld88.narod.ru/data/586.htm
Цитата: В общей сложности построены модели 6 замкнутых через дисульфидные мостики участков.
1. Cys 95 - Cys 113 в человеческом лизоциме.
2. Cys 96 - Cys 117 в альтернативном человеческом лизоциме.
3. Cys 78 - Cys 82 в альтернативном человеческом лизоциме.
4. Cys 94 - Cys 98 в лизоциме куриного яйца
5. Met 1 - Cys 13 в мышином лизоциме
6. Met 1 - Cys 24 в лизоциме куриного яйца
Другой пример - длинные фундаментальные (альфа-, 310-, пи-) и программные спирали:

Кушелев: Если Вы признаёте существование программных спиралей, то
1. Во-первых, почему они не изображаются у Вас на уровне 2D-структуры?
2. О каком фолдинге может идти речь, если 3D модель программных спиралей строится по таблице композиционного кода?
Victoriy пишет:
Специфика 3/10, пи- и альфа-спиралей формируется уже в ходе процесса фолдинга на основе простейшего структурного шаблона 3/10 спирали.
Кушелев: А тут подробнее, пожалуйста.
Вот вторичная структура двух полилизинов:
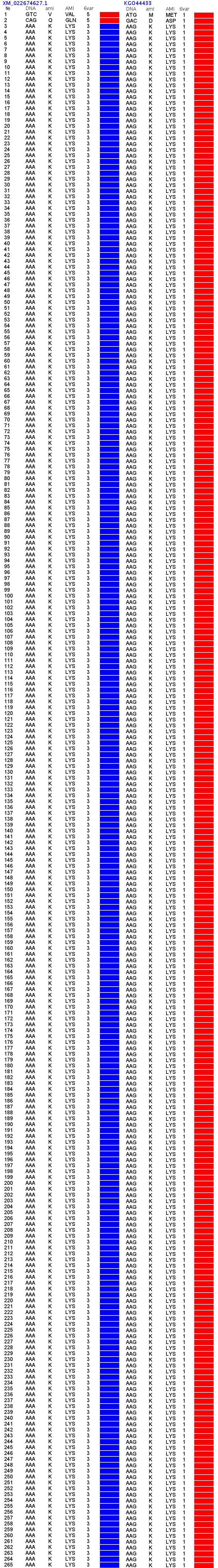
Вот, что показывает для пи-спирали алгоритм Кушелева и Соколик:

Ваш алгоритм 2D ничего не показывает. Откуда при 3D моделировании у Вас появляется пи-спираль? Каков конкретный алгоритм Вашей 3D программы, который строит пи-спираль по коду:
>XM_022674627.1 PREDICTED: Astyanax mexicanus protein FAM133-like (LOC111193520), partial mRNA
GTCCAGAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAA
Victoriy пишет:
декодированные спиральные структуры белка не обязательно сохраняются в полном объёме в его функциональной конформации после фолдинга: нередко они полностью трансформируются в бета-тяжи и петлевые структуры/повороты. Пересмотрите примеры в монографии "Пикотехнология белков", там много таких белков.
Кушелев: Вот мне и интересно, как эти два белка (пи-спиральный и альфа-спиральный) моделируются в Вашей программе. Можно пошагово показать, как по коду n(AAA) Ваша программа получает пи-спираль, а по коду n(AAG) - альфа-спираль. И ещё очень интересно посмотреть Вашу 3D модель программной 35-спирали, когда коды AAA и AAG чередуются. По моей таблице композиционного кода получается такая модель:

Пример программной 35-спирали (компактное 2D-представление)
Пример программной 35-спирали (развернутое 2D-представление)

Подробнее: https://subscribe.ru/archive/science.ne … 3210.html/
3D-модель белковой молекулы с программной 35-спиралью.
Victoriy пишет:
программа "Молекулярный конструктор" Виктории Соколик способна выдавать как 2D-схему, так и 3D-модель структурного шаблона белка по детерминирующей его нуклеотидной последовательности.
Кушелев: Это очень здjрово. Но хотелось бы уточнить, как конкретно, т.е. по шагам, получается, например, 3D модель этого белка:

2D структуру алгоритм, реализованный в программе Prosolver не показывает, но и "не очень-то хотелось". Интересно посмотреть по шагам, как программа Молекулярный конструктор строит 3D-модель этого белка, т.е. пи-спираль. Ну и сравнить с постройкой альфа-спирали из тех же остатков лизина.
Сможете показать по шагам работу алгоритма?
2017.12.08 12:01:24
Victoriy пишет:
Единственно что необходимо для полноценного удовлетворения заказчика, это виртуально в соответствующих программах (типа молекулярная динамика - МД) оптимизировать полученную структуру шаблона белка с учетом ближнего физ-хим. окружения его молекулы, т.е. смоделировать его фолдинг как бы в природных условиях, но виртуально.
Кушелев: Гипотеза фолдинга опровергается программной аллотропией (композиционным кодированием) структуры белка:

Что касается программ оптимизации, то стандартные программы не учитывают кольцевую форму электрона, поэтому непригодны для оптимизации пикотехнологических моделей.
Victoriy пишет:
... любая заказываемая модель белка необходима не просто так для красоты и любопытства, а для её дальнейшего изучения на предмет эффективности связывания (образования комплекса) с целевыми лигандами - виртуального докинга. Последний шаг и есть практическое внедрение в фармакологии, биотехнологии и др. областях.
Поэтому на этапах молекулярная динамика/докинг лаборатории Наномир понадобятся услуги опытного молекулярного биолога либо специального он-лайн сайта (такие принимали участие в CASP), а не кучи программистов, о которых говорит А.Ю. Либо же предлагать лабораториям структурный шаблон белка и оговаривать, что они должны над ним ещё сами поработать вышеописанным способом (обычно специалистов по МД и докингу в таких лабораториях достаточно).
Кушелев: Конечно, можно пытаться оптимизировать пикотехнологическую модель нанотехнологическими алгоритмами, но это неэффективно. Это примерно то же самое, что собирать айфон методом лепки из глины.
Для тех, кто не видит картинки, я буду подписывать: "Айфон, слепленный из пластилина".
Другое дело - создать программы оптимизации пикотехнологического уровня. Это безусловно сложнее, но и результат будет соответствующий:

Цикл лизоцима, замкнутый через дисульфидный мостик.
Нужно обратить внимание на то, что 3D модели некоторых участков белков получаются с пикотехнологической точностью даже без оптимизации, т.е. на уровне геометрического алгоритма. Согласитесь, что точность на уровне пикометров впечатляет по сравнению с тем, что РСА не может отличить пи-спираль от альфа-спирали.
aest пишет:
А к чему вся эта дискуссия по альфа спиралям сейчас ?
Кушелев: А при том, что альфа-спираль является жесткой структурой. Ее можно повернуть на суставе Pro относительно другой спирали:
А Виктория предлагает менять вторичную структуру. А это равносильно "размягчению костей"

Victoriy пишет:
Обратите Ваше пристальное внимание на то, что углы композиции между аминокислотными остатками задаются внутри рибосомы, а вот водородные связи, дополнительно фиксирующие их взаимное расположение уже на выходе из неё.
Кушелев: С чего бы это? Если аминокислота соединена с предыдущей, водородная связь образуется за миллисекунду:

Подробнее о протонной релаксации водородных связей: http://crm-en.ics.org.ru/uploads/kim3/crm09307.pdf
Victoriy пишет: В рибосоме водородные связи не образуются, только на выходе из неё за те же миллисекунды.
Кушелев: С чего Вы это взяли? В канале рибосомы умещаются даже все радикалы альфа-спирали. Это значит, что там та же среда, что и за пределами рибосомы. Те же молекулы воды, протоны, образующие водородные связи. Как только присоединена очередная аминокислота (0.2 ... 0.6 сек), так уже через миллисекунду образуется водородная связь, т.е. до установки следующей аминокислоты. Другого варианта нет.
Victoriy пишет: Вы уже заложили свою таблицу композиционного кода и углы для альфа-спирали и Вам проще отстаивать эту ошибку
Кушелев: Зачем отстаивать? Эксперимент показывает, что для замыкания дисульфидных мостиков фолдинг не нужен
Цикл, собранный по геометрическому алгоритму замыкается и без него:
2017.12.09 20:14:49
Кушелев: Изменение нескольких углов может привести к тому, что цикл из 22 аминокислот не замкнется вообще, т.е. не с вероятностью 1/30 000 000 000, а с вероятностью строго равной нулю (=0).
Поэтому я и пишу, что замыкание 6 циклов из разных лизоцимов не может быть случайностью. Эти замыкания показывают, что таблица композиционного генетического кода и композиционные углы правильные. Не все, конечно, а только те, которые принимают участие в формировании этих циклов.
2017.12.11 12:37:24
Victoriy пишет:
статистический анализ приведенный в монографии "Пикотехнология белков", на основе которого сделана моя таблица композиционного кода, не показал специфику кодирования альфа- и 3/10 спиралей разными кодонами. Вашей статистики на этот счет нет, поэтому такое утверждение голословно, и ненаучно его отстаивать.
Кушелев: Если анализ не показал, то это не означает, что повторный анализ не покажет
Мы с Вами знаем, что специалисты по рентгеноструктурному анализу редко отличают 310-спираль от альфа-спирали. Более того, за многие десятилетия существования РСА протяженные участки пи-спиралей вообще оказались незамеченными...
А они есть!

Так что в следующей книге "Пикотехнология белков-2" у Вас есть возможность продемонстрировать и 6 фрагментов лизоцимов, замкнутых через дисульфидные мостики без фолдинга, и сверхдлинные фундаментальные и программные спирали белковых молекул, структура которых определена по таблице композиционного кода, где Ваши данные для альфа- и пи-спиралей полностью совпадают с моими. Вы ещё хотели показать экспериментальные данные, с которыми совпадают Ваши альфа- и пи-спирали длиной более 63 витков:

Оригинал: https://img-fotki.yandex.ru/get/892397/ … 5_orig.png
А если этих экспериментальных данных еще нет, то что мешает их получить? В институте белка мне сообщили, что определить структуру с помощью РСА могут в Курчатовском институте. Давайте вместе предложим им определить две структуры полилизина, закодированные триплетами n(AAG) и n(AAA).
2017.12.09 20:50:04
Кушелев: На иллюстрации показано, что вторичная структура, построенная по алгоритму Виктории Соколик на 100% совпадает со вторичной структурой, полученной по композиционной таблице Кушелева. Это только в программе от Prosolver коду AAA ничего не соответствует, а по алгоритму Виктории Соколик код AAA соответствует левой спирали, т.е. пи-спирали. Вам осталось подправить углы пи-спирали, чтобы не пришлось получать "те же результаты другим способом" уже в 3D версии программы
Если я не ошибаюсь, Вы предлагаете строить модель пи-спирали в два этапа. На первом построить неправильную левую спираль (3 АО на виток), т.е. с неправильными водородными связями, соединяющими n-ый АО с (n+3)-им, а на втором этапе переделать неправильную левую спираль в правильную пи-спираль, т.е. разорвать все водородные связи n ... (n+3), изменить все углы между аминокислотными остатками со 120 градусов до 87, после чего сделать заново все водородные связи, но уже не с третьим АО, а с пятым, т.е. n ... (n+5). Другими словами, неправильно построить, потом всё сломать и построить правильно. А почему нельзя сразу сделать правильно по Вашей же таблице композиционного кода, которая в этом месте на 100% совпадает с моей? Просто сразу взять правильные углы пи-спирали.




Альфа-спираль Бета-спираль Пи-спираль 310-спираль
Подробнее можно посмотреть в 287-ом выпуске рассылки: http://nanoworld88.narod.ru/data/287.htm
2017.12.10 14:04:17
Когда Виктория Соколик, писала книгу "Пикотехнология белков", научных открытий, о которых сегодня идет речь, ещё не было. Поэтому эти иллюстрации не могли попасть в книгу из будущего:


Но буквально вчера выяснилось, что в программе от Prosolver вторичная структура показана не так, как должна быть показана согласно таблице композиционного кода от Виктории Соколик.
Там, где программа показывает отсутствие вторичной структуры, в действительности по таблице Виктории Соколик вторичная структура есть. Это структура левой спирали, т.е. пи-спирали:

Так что разногласий во вторичной структуре нет. Разногласие в величине угла, т.е. на уровне пространственной модели, т.е. 3D версии программы.
Виктория считает, что рисобома собирает левую спираль с углами по 120 градусов (три АО на виток), после чего некий "фолдинг" ломает эту структуру, т.е. разрывает водородные связи n ... (n+3), меняет все углы со 120 на 87 градусов и создаёт все водородные связи заново, т.е. уже n ... (n+5). А я считаю, что рибосома строит пи-спираль с первого раза. Без мистического фолдинга.
aest пишет:
лизоцим не является жесткой фигурой, подобной треугольнику, там вполне возможно согласованно поизменять углы без разрыва дисульфидного мостика, подобно тому, как это можно сделать в параллелограмме
Кушелев: Только он замкнулся через дисульфидный мостик без изменения углов. А это значит, что фолдинга нет на уровне вторичной структуры. Кстати, Вы можете "поизменять углы" и выложить на форум результат своего колдовства. Интересно посмотреть, что за углы Вы будете менять, и удастся ли Вам поменять их согласованно?
Ваши "просто слова" против модельного эксперимента ровным счётом ничего не значат
Сначала измените согласованно углы, но не в параллелограмме, а в модели фрагмента лизоцима, а мы посмотрим, замкнётся ли он после этого или нет.
Victoriy пишет:
Вы строите свою таблицу кодирования разновидностей спиралей опираясь на свои фантазии и отмахиваясь от результатов статистики.
Кушелев: Вообще-то я сразу показал, как я строил свою таблицу композиционного генетического кода
Я воспользовался данными РСА для наиболее изученных белков. При этом на пластмассовых моделях я получил параметры альфа-спирали, 310-спирали, пи-спирали и бета-спирали. Модельные эксперименты показали, что составленная таблица композиционного кода работает без ошибок. Позднее, когда сборка моделей белка была автоматизирована, обнаружился цикл лизоцима, замкнувшийся через дисульфидный мостик без фолдинга, т.е. строго по таблице композиционного кода.
Ещё позднее я нашел ещё 5 фрагментов разных лизоцимов, замнувшихся без фолдинга.
Более того, в процессе этого исследования я обнаружил замыкание дисульфидных мостиков не только между цистеинами, но и между цистеином и метионином, что явилось научным открытием, согласитесь. Вам известно, что дисульфидные мостики могут образовываться между цистеином и метионином?
Здесь программа от Prosolver показывает отсутствие вторичной структуры по композиционному коду пи-спирали, но мы уже выяснили, что таблицы композиционного кода по части пи-спирали совпадают, поэтому в действительности эти циклы лизоцима обязаны замкнуться и по алгоритму Виктории Соколик
Кстати, эти модели совпадают и с экспериментальными данными, если не считать дисульфидных мостиков между цистеином и метионином. Но тут уж дело времени. Должны появлиться экспериментальные данные и по дисульфидным мостикам между метионином и цистеином. Рано или поздно экспериментаторы должны обнаружить эти мостики...
Kushelev пишет:
Меняйте согласованно углы. Но не забывайте, что эти углы не лежат в одной плоскости
aest пишет: Возьмите металлическую цепь о тысячи звеньях, замкните. Бросьте комком на землю. Сколько там углов между звеньями согласованно изменились не лежа при этом в одной плоскости? Цепь не разомкнулась. Мистика...
Кушелев: Жаль, что Вы не отличаете механизм белковой молекулы от механизма цепи. В молекуле белка вращение происходит вокруг некоторых осей:

В учебнике молекулярной биологии есть такой термин: "вращение затоможено". Это определяется как раз структурой электронной оболочки молекулы. Так же, как рука может гнуться в локте не по любому направлению. Сустав - это не шарнир.
Victoriy пишет:
абсолютно не соответствующие реальному механизму трансляции белка.
А соответствует очередь протонов на образование водородных связей в альфа-спирали на выходе из канала рибосомы?
Кушелев: Вы согласны, что в канале рибосомы присутствуют протоны? Если согласны, то что им мешает через миллисекунду после установки очередного аминокислотного остатка образовать водородную связь? Т.е. ещё до того, как тРНК отпустит аминокислотный остаток, который она держит 4-мя вандерваальсовыми связями:
Подробнее: http://nanoworld88.narod.ru/data/216.htm
Подробнее: http://nanoworld88.narod.ru/data/247.htm

Кушелев: Судя по этой схеме, Виктория отмеряет углы поворота tRNA вокруг оси белковых спиралей.
Увжаемая Виктория! А Вас не смутил угол 0 градусов? Или Вы его "смело заменили" на 180 градусов?
Кстати, эту ошибку легко исправить.
Достаточно организовать "фолдинг" таблицы композиционных углов от Виктории Соколик, чтобы получилась таблица композиционных углов от Александра Кушелева.
Сами структуры белков фолдировать не нужно. Достаточно фолдировать таблицу углов
А я никак не мог понять, откуда Вы такие углы берете? Это можно назвать "антифолдинг" таблицы композиционных углов. Осталось добавить "фолдинг" таблицы композиционных углов, "и всех делов" .
Получается, что Вы не показывали Prosolver иллюстрации, где показано, вокруг каких осей вращаются модели аминокислотных остатков?
Есть же описание алгоритма от Евгения Неделько, которое находится в открытом доступе с 1998 года.
В программе Неделько выводились только координаты центров атомов, а углы были взяты у Рамачандрана. Это уже в более поздней версии Дениса Савина программа скрипт стала выводить не только координаты центров атомов, но и кольцегранные оболочки. Помню Вы там ещё ошибку нашли. Правда, программа правильно заработала после исправления второй ошибки. Оказалось, что Денис Савин перепутал в одном месте координату в уравнениях. Потом мы с Валентином поменяли зеркальные отражения аминокислотных остатков на правильные и промасштабировали модели по экспериментальным данным.
Кстати, в программе Дениса Савина вращение тоже реализовано не вокруг одной оси, как было бы естественно, а последовательно вокруг трёх декартовых осей. Это уже связано с вычислительной средой. При этом сначала оказалось, что все спирали строятся правильно, а изломы спиралей неправильно. Выяснилось, что правильные углы между спиралями разных типов получаются при правильном задании начала отсчета углов. Другими словами, нужно поставить аминокислоту в правильную позицию, правильно сориентировать по всем трем осям, после чего получаются корректные модели белков. Но это только геометрический уровень.
2017.12.11 09:59:42
aest пишет:
Вы приводите в качестве примеров жесткие фигуры, у которых грани как раз треугольники. Но масса таких фигур ничтожна по сравнению с массой нежестких фигур. Так что основной, имеющий важность для науки вывод не опровергнут: фолдинг есть. А вы можете и дальше его не замечать, пусть он тут для других зияет
Кушелев: Вы очень настойчиво не хотите изучать азы философии. Кстати, Ваше заявление по поводу "массы таких фигур", применительно к белкам, тоже ошибочно. Подавляющее большинство белков содержат альфа-спиральные участки. На этих иллюстрациях они показаны красным цветом. Их можетбыть много, может быть мало.


Альфа-спиральные участки присутствуют в подавляющем большинстве белков. Их отсутствие является редким исключением. Так что Вам срочно нужна новая ошибка "для оправдания"

Попобуйте, поколдуйте, например, над жёсткостью альфа-спирали.
2017.12.11 10:15:37
aest пишет:
фолдинг происходит благодаря наличию у белка "суставов", т.е. мест, где он легко может гнуться, о чем и говорит Виктория. Так что, повторюсь, основной, имеющий важность для науки вывод не опровергнут: фолдинг есть, а пикотехнология Кушелева ошибочна
Кушелев: Вы, вероятно, не поняли моего объяснения. На суставах может гнуться третичная структура белка, т.е. состоящая, например, из жёстких альфа-спиральных участков. Действительно, на уровне третичной структуры есть и фолдинг, т.е. однократная "укладка жестких участков вторичной структуры на суставах Pro", и динамический "фолдинг", т.е. по существу функционирование пико-механизмов. Я даже показал динамические модели белков, демонстрирующие этот динамический "фолдинг":


Однако Виктория пишет не об этом "фолдинге третичной структуры", а именно о мистическом фолдинге вторичной структуры, когда этот мистический "фолдинг" должен сначала разрушить построенные рибосомой "шаблоны", например, левую спираль с углами между аминокислотными остатками в 120 градусов, разорвать все водородные связи n ... (n+3), изменить все углы со 120 на 87 градусов и замкнуть заново все водородные связи уже по схеме n ... (n+5). Вот о чём дискуссия, а не о том, что угол между альфа-спиральными участками может меняться на Pro.
aest пишет:
Напоминаю слова Виктории:
"Кстати фолдинг это не кардинальное изменение закодированного структурного шаблона, а лишь его "косметическая" оптимизация."
Кушелев: А за этими словами кроется разрушение всех водородных связей, изменение всех углов и создание всех водородных связей заново. Вот такая "травматическая косметика"


. . . . . Было . . . . . . . . . . Стало
aest пишет:
Если я не ошибаюсь, альфа спираль у Виктории получается в результате фолдинга.
Кушелев: Совершенно верно! Рибосома по композиционному коду строит сначала 310-спираль (по Виктории):


310-спираль альфа-спираль
после этого мистический фолдинг (будем называть вещи своими именами) разрушает все водородные связи n ... (n+3), меняет все углы между аминокислотными остатками с 120 градусов (3 АО на виток) до 100 градусов (3.6 АО на виток), после чего создает все водородные связи заново. "Травматическая косметика" в двух словах
Victoriy пишет:
А как насчет Вашего обобщения частного случая лизоцима до всей когорты белков? Это ли не грубейшая философская ошибка, ведь дисульфидные мостики имеет менее 1 % белков.
Кушелев: Давайте разберёмся. Если 1% белков строится по композиционному генетическому коду, то какой вывод можно сделать о принципах сборки белков? Могут ли белки, в которых есть дисульфидные мостики строиться по иным принципам, чем остальные белки? Может ли альфа-спираль собираться по композиционному коду, а пи-спираль без композиционного кода?
Если уж композиционный код обнаружен, значит он работает для всех белков рибосомальной сборки. Не может же рибосома "тут работать, а там не работать". Это же абсурд!
Конечно есть нюансы. Рибосомы митохондрий работают по слегка другой таблице композиционного кода:

Более того, в некоторых организмах используется своя таблица генетического кода, но это нюансы. Если рибосома работает, то она работает одинаково при сборке любых рибосомальных белков.
Victoriy пишет:
что Вы носитесь с этими дисульфидными мостиками лизоцима. Никто же не спорит о закодированности структуры белка в разной степени сохраняющейся в конформации белка после фолдинга последнего.
Кушелев: Если пространственная структура сохранилась в циклах лизоцимов на все 100%, то фолдингтне нужен.
Victoriy пишет:
для лизоцима можно предположить замыкание находившихся по соседству в пространстве (а не в линейной последовательности аминокислот) SH-групп цистеинов в дисульфидные мостики ещё на этапе синтеза его структурного шаблона, что способствовало сохранению пространственного взаиморасположения КЛЮЧЕВЫХ узлов в этом ферменте (в частности активного центра).
Кушелев: Так по соседству атомы серы могут оказаться только в одном случае. Если все остатки установлены строго по композиционному коду. Случайно это невозможно. Для одного цикла из 22 АО вероятность случайного "расположения по соседству" равна 1/30 000 000 000, а для всех 6 циклов разных лизоцимов эту вероятность нужно ещё в 6-ю степень возвести. Получится (1/30 000 000 000)^6 = 1,37e-63 Просто нереальная вероятность.
Victoriy пишет:
Гомологичные лизоцимы унаследовали найденный природой ход своего уникального сворачивания для формирования активного центра,
Кушелев: Если структура в окончательном варианте построена по коду, её уже невозможно свернуть. Она уже "свёрнута". Вы пытаетесь "фолдингом" положить в карман то, что там уже лежит.
В процессе эволюции композиционный код подстраивался под физико-химические взаимодействия, поэтому после денатурации белки иногда могут полностью ренатурировать. Но рибосома по композиционному коду собирает их не денатурированными, согласитесь. Известно же, что рибосома собирает белки быстро, а ренатурируют они медленно. Так что фолдинг тут неуместен.
Victoriy пишет:
остальные несущественные детали
Кушелев: Кстати, о "несущественных деталях". В процессе исследования циклов разных лизоцимов, которые замыкаются через дисульфидные мостики, мне удалось обнаружить, что эти самые дисульфидные мостики возникают не только между цистеинами, но и между цистеином и метионином! Это научное открытие тоже прошло незамеченным, а зря...
Victoriy пишет:
моя программа Молекулярный конструктор по коду AAG построит шаблон правой спирали полилизина, а по коду ААА - соответственно левой спирали. А далее виртуальный фолдинг с поиощью МД придаст им вид альфа- и пи-спиралей, если конечно их устойчивость выиграет в противостоянии с влиянием микроокружения. Вы же в курсе, что закодированные спирали сохраняются в конформации белка в лучшем случае на 80-90%, а в худшем - на 10-20 %.
Кушелев: Итак, в процессе дисскуссии выяснилось, что после исправления программы от Prosolver вторичные структуры полилизинов, построенных по кодам n(AAA) и n(AAG) по Соколик и Кушелеву полностью совпали:

Однако, Виктория Соколик утверждает, что рибосома соединяет водородными связями остатки левой спирали через 3 АО, а потом некий "фолдинг" разрушает все эти водородные связи и создаёт заново, но уже через 5 АО. Согласитесь, что это абсурд.

Алгоритм определения вторичной структуры полилизина у Вас оказался правильный, т.е. точно такой же, как у меня. Просто в программе от Prosolver он не был реализован.
Осталось исправить таблицу углов, т.е. чтобы по коду пи-спирали строилась именно пи-спираль, а не полуфабрикат, который потом нужно переделывать в пи-спираль
Victoriy пишет:
это не совпадение с Вашими моделями, а соответствие экспериментальным данным.
Кушелев: Я бы рад ошибиться и признать свою ошибку, особенно, если Вы сможете показать экспериментальные данные по белку: XM_022674627, который является прямой пи-спиралью длиной 263 аминокислотных остатка, т.е. 263/4.1=64 витка прямой пи-спирали.
И это при том, что официально считается: http://info-farm.ru/alphabet_index/p/pi-spiral.html
Цитата: Длинная ливозакручена π-спираль вряд наблюдается в белках
Конец цитаты.
https://ru.wikipedia.org/wiki/%D0%92%D1 … 1%80%D0%B0
Цитата: π-спираль, или спираль 516, — спираль с широкими витками, в результате в центре спирали остаётся пустое пространство. В белках встречается редко, обычно не более одного витка.
aest пишет:
Виктория утверждает, что замыкание происходит во время синтеза структурного шаблона, а потом происходит фолдинг.
Кушелев: Вы точно ничего не напутали? Если дисульфидный мостик замкнулся, то пространственная структура белка зафиксирована. Это всё равно, что закрыть дверь на ключ.
Гипотеза фолдинга заключается в том, что из рибосомы выходит типа линейная цепочка аминокислотных остатков, которая сама, т.е. уже без рибосомы, сворачивается в объёмную конструкцию.
А я на примере циклов лизоцимов показал, что углы между аминокислотными остатками запрограммированы в таблице композиционного генетического кода, т.е. рибосома по композиционному коду сразу собирает вторичную структуру белка. Например, если код n(AAG), то из рибосомы выходит прямая альфа-спираль с водородными связями между n-ой и n+4-ой пептидными группами. Если код n(AAA), то выходит прямая пи-спираль с водородными связями между n-ой и n+5-ой пептидными группами. Сворачивать уже ничего не требуется. Либо нужно сначала разломать закодированную вторичную структуру, а потом снова сделать, что абсурдно, согласитесь. Открытие композиционного кода опровергло гипотезу фолдинга. Другое дело - укладывание вторичной структуры белка в третичную. Если между спиральными участками попадаются суставы Pro, то спиральные участки могут двигаться, как кости скелета на суставах. И тут уже вступают в действия силы притяжения и отталкивания между участками спиралей. Этот процесс вполне можно назвать фолдингом, но ... фолдингом уровня третичной структуры, которая складывается из жестких спиральных (и не очень) участков вторичной структуры. Циклы лизоцимов - это законченные третичные структуры, которые фолдингу не подлежат, т.к. дисульфидный мостик уже замкнулся и зафиксировал пространственную форму. И случилось это в процессе рибосомальной сборки, а не в процессе мистического фолдинга уровня вторичной структуры. Ведь дисульфидный мостик замкнулся уже на уровне геометрического алгоритма, т.е. композиционных кодов и композиционных углов. Это значит, что никаких взаимных движений на суставах Pro не было. Как построили, так и стоит.
Поймите, что нельзя построить (фолдингом или чем угодно) то, что уже построено. Либо надо ломать и строить заново, что абсурдно.
Victoriy пишет:
и левая спираль или поворот (NNА)
Кушелев: А это уже ближе к делу. Значит Ваша программа по коду AAA действительно построит не правую, а левую спираль. Но реальная пи-спираль имеет 4.1 аминокислотных остатка на виток: http://info-farm.ru/alphabet_index/p/pi-spiral.html .
Аминогруппа одной аминокислоты формирует водородную связь с C = O группой аминокислоты на пять остатков ранее
А это значит, что Ваш "фолдинг" должен разорвать все водородные связи с третьими аминокислотными остатками и создать новые водородные связи с пятыми аминокислотными остатками, что сами понимаете, нереально.
По коду AAA рибосома сразу ставит аминокислоту так, чтобы водородная связь замкнулась на пятый аминокислотный остаток. И не надо ничего переделывать
Но самое интересное, что пи-спираль в Вашей программе так же однозначно закодирована, как и в моей программе Пикотех, т.е. в Вашей таблице AAA - тот же композиционный код пи-спирали, а код AAG - тот же композиционный код альфа-спирали.
Вы просто ввели в заблуждение отсутствием вторичной структуры в программе от Prosolver:

Пи-спираль по коду AAA (Кушелев) и отсутствие вторичной стуктуры по коду AAA (Соколик)
Я так понял, что эту ошибку Вы частично исправили в программе "Молекулярный конструктор", т.е. теперь по коду AAA программа показывает не отсутствие вторичной структуры, а левую спираль, но ещё не 4.1 аминокислотных остатка на виток, а 3. Но это тоже дело поправимое. Ведь в следующей версии можно изменить 3 на 4.1. И не нужно никаких чудо-"фолдингов" для превращения всех неправильных углов пи-спирали в правильные
2017.12.09 13:33:17
На основе структурного шаблона правой спирали в ходе фолдинга могут быть смоделированы все три основных разновидностей правых спиралей: 3/10-, альфа- и пи-.

Кушелев: Так-так-так... Пи-спираль вообще-то левая, 4.4 остатка на виток. Но я уже писал, что бог с ними, с этими переделками 310-спирали в разные спирали белков. Вы скажите, откуда "фолдинг" узнает, во что переделывать "310-шаблон". Ведь информация о том, что полилизин будет пи-спиралью, содержится в кодах: n(AAA), а информация о том, что полилизин будет альфа-спиралью, содержится в кодах n(AAG). В шаблоне информации типа AAA/AAG уже не содержится. Верно? Так откуда "фолдинг" узнает, во что переделывать шаблон? Где информацию брать?
Рассмотрите пи-спираль полилизина, построенная по триплетам AAA и альфа-спираль полилизина, построенная по триплетам AAG.
фолдинг этих самых структурных шаблонов в энергетически устойчивую функциональную конформацию
Кушелев: Уважаемая Виктория! Как Вы прокомментируете открытие программных спиралей белковых молекул?

Здесь Вы видите 19 вторичных структур поливалина. На схеме вторичной структуры представлены фундаментальные спирали:
альфа-, 310-, пи-, бета-
и программные:
14-, 35-, 352-, 144-, 25-, 22225-, 411412-, 255-, 2444-, 1144-, 11444444-, 23-, 335(коллагеновая)-
Помните, как мы с Вами дискутировали по поводу лизиновых "хвостов", т.е. участков пи-спиралей длиной более 10 аминокислотных остатков? Вы ещё писали, что это всё гипотетические структуры...
А как Вы прокомментируете тему: "Рекорды сверхдлинных спиралей белковых молекул"?
Там встречаются фундаментальные спирали (альфа-, 310-, пи-, бета-) длиной более 3000 аминокислотных остатков. И программные спирали, в т.ч. фрактальные более 100 периодов, например, по 35 аминокислотных остатков в периоде, т.е. более 3500 аминокислотных остатков в спирали.
Если бы была верна гипотеза фолдинга, то зачем было бы кодировать все эти спиральные структуры?
Антигомология = программная аллотропия опровергает гипотезу фолдинга. Согласны?
*
Модель 2012 года, версия 4 var
http://molbiol.ru/forums/index.php?show … &st=50
А размеры Вы можете привести? Например, у дырки какие? И, например, у всего коннексона в поперечине?
Кушелев: Давайте вместе посчитаем. Шаг альфа-спирали известен - 0,54 нм
Считаем число витков, и умножаем на 0.54 нм
Если я не ошибся, то внизу получилась масштабная линейка с шагом 0.54 нм. Считаем габаритный размер и размер отверстия. Габаритный размер 0.54*22=~12нм. Это грубая оценка. Если нужно, то можно вывести точный мастшаб в 3DS Max smile.gif Диаметр отверстия 0.54*(6...7)=~3...4 нм
В этом ракурсе видно, что соседние коннексины должны входить в зацепление и двигаться до внешнего изгиба альфа-спирали соседнего коннексина. Четвертичную структуру удобнее собирать из 3D-распечаток субъединиц. В виртуальном пространстве сборку четвертичной структуры делать значительно сложнее. Хотя, если есть суперкомпьютер, специальные программы, шлем, перчатки, то может быть удобнее будет именно в виртуальном пространстве...
Вот тут-то и проблемка...
По РСА размер самой узкой части отверстия 1,4 нм, ошибка больше, чем в два раза. А это самая важная характеристика, связанная с пропускной способностью контакта. И точность РСА Вас тут уже не спасет.
Кушелев: В данном случае ошибка в два раза - вполне нормально. Я просто ошибся при сборке четвертичной структуры. Если зацепить коннексины, чтобы альфа-спираль одного коннексина двигалась внутри другого, то размер отверстия как раз совпадёт. Кроме того, точные размеры можно получить не на упрощённой модели, где один аминокислотный остаток показан 14-гранником, а на более точной модели, где один электрон показан одним кольцом.
Так что не торопитесь с выводами. Сначала нужно правильно сложить субъединицы, а потом уже сравнивать
В случае зацепления коннексинов размер отверстия коннексона получается 1.5-2 нм, но нужно учитывать, что в этой модели не показаны радикалы аминокислотных остатков. А радикалы могут уменьшить диаметр отверстия на 0.5...1 нм. Так что имеет смысл построить менее упрощённую пикотехнологическую модель четвертичной структуры коннексона.
Кстати, я не понял, что означает размер Entrance diameter 35A ? Это размер отверстия при максимальном увеличении диафрагмы-коннексона?
Кушелев: Тогда получается, что отверстие не может уменьшиться до нуля, как я пытаюсь изобразить в анимации? Тогда понятно, почему альфа-спирали налезают друг на друга. Они просто не могут до такой степени сблизиться, чтобы отверстие уменьшилось до нуля.
А с выводами Вы зря торопитесь. Модель ещё не доделана. Есть только пикотехнологическая модель третичной структуры, которая построена автоматически.
И вообще, я бы сказал, что общий вид коннексона, полученного с помощью Вашей модели, совершенно не похож на реальный.

Кушелев: Все так считают?
В случае зацепления коннексинов размер отверстия коннексона получается 1.5-2 нм, но нужно учитывать, что в этой модели не показаны радикалы аминокислотных остатков. А радикалы могут уменьшить диаметр отверстия на 0.5...1 нм. Так что имеет смысл построить менее упрощённую пикотехнологическую модель четвертичной структуры коннексона.
Программа Пикотех не делает четвертичную структуру. Я же Вам объяснил, что четвертичную структуру приходится собирать вручную. При этом делать это в виртуальном пространстве 3DS Max без виртуальных перчаток, стереошлема и специальной программы очень неудобно. Поэтому с первого раза угадать четвертичную структуру коннексона не удалось. Да и со второго раза тоже, т.к. альфа-спирали налезают друг на друга. Похоже, что субъединицы должны быть несколько повёрнуты по другим осям.
Кстати, я не понял, что означает размер Entrance diameter 35A ? Это размер отверстия при максимальном увеличении диафрагмы-коннексона?
Тогда получается, что отверстие не может уменьшиться до нуля, как я пытаюсь изобразить в анимации? Тогда понятно, почему альфа-спирали налезают друг на друга. Они просто не могут до такой степени сблизиться, чтобы отверстие уменьшилось до нуля.
А с выводами Вы зря торопитесь. Модель ещё не доделана. Есть только пикотехнологическая модель третичной структуры, которая построена автоматически.
Электроны вообще не бывают в виде колец! Они ни в виде ничего, они "размазаны" по некоторой области, причем трехмерной. В инертных газах это будут не кольца, а примерно сферы с некоторой, не определяемой точно толщиной, если же атом связан с какими-то другими атомом, то там уже это какие-то фигуры сложной формы.
Приведите статью, в которой говорится, что есть ЭКСПЕРИМЕНТАЛЬНЫЕ данные, противоречащие данной конфигурации.
Кушелев: Почему, собственно? Десятки исследователей пришли к выводу, что лептоны, в частности электроны имеют форму тора. Не знали?
Вот здесь подробнее можно узнать: http://nanoworld88.narod.ru/data/104.htm
Кольцегранные модели атомов: https://docs.google.com/document/d/1C_JL6W24jEktuwJgZocTB4R4sPyr-csg/edit
