Почему Пикотехнология точна на 100%
Высоко периодичные структуры белков
2017.12.27 09:58:21
Kushelev пишет:
Только что побеседовали по скайпу с Викторией Соколик (1 час 57 минут), в результате чего разобрались с осями, вокруг которых вращаются аминокислоты. Короче, по коду пи-спирали у Виктории получается пи-спираль без фолдинга. Разногласия уменьшились до вопроса кодирования 310-спирали. Виктория считает, что замена кодов альфа-спирали на коды 310-спирали (из моей таблицы) друг на друга не влияют на сборку правой спирали, которая будучи собранной из многогранников имеет угол 310-спирали. Превращением 310-спирали в альфа-спираль "заведуют радикалы аминокислотных остатков". Я считаю, что альфа-спираль и 310-спираль кодируются разными композиционными кодами и радикалы тут ни при чём. В качестве примера я предлагаю рассмотреть разные структуры поливалинов:

2017.12.27 12:18:29
Skype, 2017-12-25:
[24.12.2017 21:12:51] Кушелев Александр Юрьевич: Здравствуйте, уважаемая Виктория!
[24.12.2017 21:13:35] Кушелев Александр Юрьевич: Как Вы относитесь к идее сделать доклад, написать статью, опубликовать каталог на тему: "Определены структуры всех белков человека" ?
[24.12.2017 21:15:05] Кушелев Александр Юрьевич: В качестве приложения можно дать ссылку на все структуры, определенные по разным таблицам композиционного генетического кода.
[24.12.2017 21:16:14] Кушелев Александр Юрьевич: При этом часть белков по таблице Виктории Соколик и Александра Кушелева полностью совпадут. Их можно не дублировать.
[24.12.2017 21:17:28] Кушелев Александр Юрьевич: Можно предложить всем желающим составить собственную таблицу композиционного кода и сравнить результаты определения структур по разным таблицам.
[24.12.2017 21:17:49] Кушелев Александр Юрьевич: Мне, например, без разницы, какая таблица окажется более правильной.
[0:43:17] Виктория Соколик: Добрый вечер, Александр Юрьевич. Из Вашего обращения я не поняла, что Вам надо: доклад, статью или каталог структур? Это разные и по назначению и по объёму вещи. И ещё вопрос: где Вы намерены размещать эту информацию? Под каждый ресурс делается специальный проект, Вы ведь должны это понимать.
[0:44:28] Виктория Соколик: И ещё: прежде чем браться писать о "структуре"! уточняйте вторичной "всех белков" надо ещё внимательно на неё посмотреть.
[0:49:15] Виктория Соколик: Не в обиду будет сказано, но Ваши аппетиты и методы решения ГЛОБАЛЬНЫХ задач напоминают мне проповеди проповедников, которые учат любить весь мир, не умея полюбить даже одного человека. Это я к тому, что важнее определить 3D-структуру десятка, но очень нужных и перспективных белков, чем 2D-структуру всех подряд.
[0:51:20] Виктория Соколик: ***** и посмотрим на Ваши ВСЕ БЕЛКИ.
[3:07:09] Кушелев Александр Юрьевич: Благодарю! Что и где размещать - это Вам решать. Вы же разместили книгу "Пикотехнология белков". Я считаю, что это самое правильное Ваше решение. А это значит, что Вы сможете решить, что лучше сделать: Доклад на конференции, статью в научный журнал или небольшую книгу-каталог, например, в то же издательство, что и книга "Пикотехнология белков".
Безусловно надо написать "вторичной и выборочно третичной и даже четвертичной". Можно показать пространственные модели ключевых белков. Таких как фундаментальные альфа-310-бета-пи-спирали, программные спирали типа коллагена и наиболее интересные структуры из генома человека. Как выяснилось, каждая хромосома кодирует до 10 белков, представленных преимущественно программными спиралями. Музыка их сборки напоминает народные мелодии
Дублирую по скайпу одну из таких структур:

[3:10:09] Кушелев Александр Юрьевич: Очень зрелищными будут 6 циклов разных лизоцимов, которые замкнулись через дисульфидные мостики. Есть и ряд других белков, которые имеет смысл показать читателям/слушателям. Короче, давайте обсудим и сделаем, как проще и лучше. Лучше Вас вряд ли кто-то это сможет.
[25.12.2017 20:44:13] *** Пропущенные звонки от Виктория Соколик ***
[25.12.2017 21:57:55] Кушелев Александр Юрьевич: Добрый вечер, Виктория! Вы звонили?
[25.12.2017 21:58:56] Виктория Соколик: Да, Вы хотите побеседовать?
[25.12.2017 21:59:16] Кушелев Александр Юрьевич: Давайте через 30 секунд
[25.12.2017 22:00:04] Кушелев Александр Юрьевич: Вы звоните или я?
[25.12.2017 22:01:16] *** Звоним Виктория Соколик ***
[25.12.2017 23:58:54] *** Звонок завершен. Продолжительность: 1:57:39 ***
[26.12.2017 10:46:57] Кушелев Александр Юрьевич: Уважаемая Виктория! Разрешите разместить нашу вчерашнюю дискуссию на форуме с иллюстрациями и аудиозаписью. Я думаю, что участникам и читателям форума лаборатории Наномир будет интересно узнать, что углы пи-спирали и 310-спирали в наших с Вами моделях совпадают уже на уровне геометрического алгоритма. Что касается различия кодирования альфа- и 310-спирали, то это уже мелкие нюансы, т.к. угол альфа-спирали 97...100 градусов отличается от угла 310-спирали 119...121 градус всего градусов на 20. Примечательно и то, что бета-спираль в Вашей модели тоже является спиралью, а не плоским "тяжем", как считалось в старых учебниках молекулярной биологии. Теперь все желающие могут изучать молекулярную биология в т.ч. по новому учебнику "Пикотехнология белков", тем более, что уважаемый рецензент однозначно написал: "более чем полный массивный объем информации, увлекательная и яркая форма ее изложения и оформления делает монографию применимой также и в качестве учебника для студентов естественнонаучных специальностей, особенно таких, как биохимия, биофизика и биотехнология."
Иллюстрации, созданные в процессе дискуссии Соколик-Кушелев:




Оси вращения для текущего и предыдущего аминокислотных остатков показаны белыми линиями слева и справа соответственно.
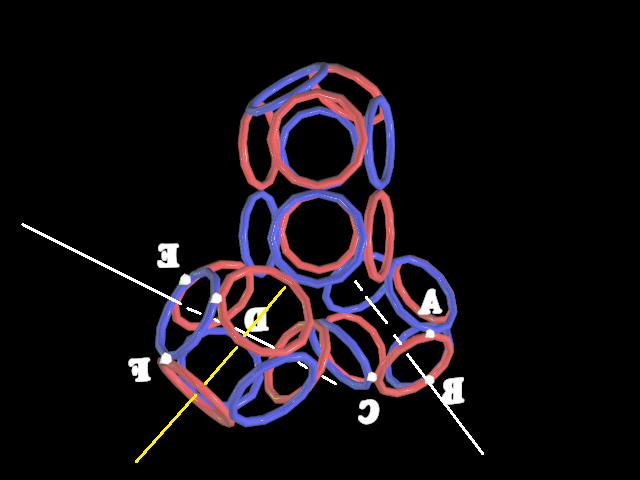
Кушелев: Ось вращения аминокислот в модели Кушелева показана белым цветом (слева).
Ось вращения аминокислот в модели Соколик показана желтым цветом.
Ось вращения следующего аминокислотного остатка показана белым цветом (справа) и одинакова в моделях Соколик и Кушелева.
Кушелев: Обратите внимание на цвет колец. Цветом обозначены магнитные полюса электронов, обращенные от ядра атома наружу. В модели Кушелева следующий аминокислотный остаток присоединен таким образом, что чередование магнитных полюсов одинаково для всех аминокислотных остатков, т.е. белок собирается из одинаковых магнитных изомеров.
В модели Соколик четные аминокислотные остатки являются противоположными магнитными изомерами по отношению к нечетным. В них электронная "шкура" молекулы как бы вывернута наизнанку.
Если это не учитывать, то будет нарушен принцип чередования магнитных полюсов электронов, что соответствует правилу Хюккеля для сопряженных химических соединений. Конечно, любой электрон в 8-электронной оболочке можно перевернуть. При этом энергия связи из-за магнитного отталкивания одноименных полюсов изменится на доли процента, но этого достаточно, чтобы магнитный изомер был нестабильным. Поэтому все аминокислоты являются не только L-изомерами, но и одинаковыми магнитными изомерами. Это особенно важно для работы рибосомы, которая держит аминокислоты пятью вандерваальсовыми "присосками", которые одновременно являются и водородными связями. Эти магнитно-водородные "присоски" не будут удерживать магнитный изомер аминокислоты с той же силой, если магнитное притяжение в них сменить на магнитное отталкивание. Однако, Виктория Соколик отвергает факт вращения тРНК, модель которого вы видите ниже:
Она считает, что аминокислота удерживается ACC-концом тРНК так, как об этом написано в современных учебниках. Кстати, давайте прочитаем ...

В современных учебниках написано и нарисовано, что аминокислота удерживается ССА-концом тРНК через единственную ковалентную или водородную связь. При этом ни о каких разных углах речи не идёт. В старых учебниках считается, что все аминокислоты присоединяются к предыдущим под одним углом, образуя линейную цепочку, которая после выхода из канала рибосомы сама чудесным образом решает, в какую вторичную структуру ей свернуться, в альфа-спираль, в 310-спираль, в пи-спираль или в бета-тяж. Ну или вообще не сворачиваться.
Модель механизма трансляции я опубликовал в рассылке "Новости лаборатории Наномир": http://nanoworld88.narod.ru/data/272.htm
Механизм декодирования угла поворота аминокислоты.
В дискуссии Соколик-Кушелев мы обсудили и модель метионина Met:
2017.12.16 18:20:4
Виктория Соколик пишет:
В монографии с картинками показаны оси вращения и точки отсчета углов, которые реализованы в моей программе. Я умышлено не шла по Вашему пути трех осей и углов для них, чтобы не повторять Ваших заблуждений. Читайте матчасть.
Аминокислота поворачивается вокруг вертикальной оси (оси симметрии tRNA)
Поворот на 120 градусов относительно оси симметрии tRNA формирует углы альфа-310-, бета-, пи-спиралей соответственно.
Технология трансляции генетического кода в структуру белка
Виктория докладывала о кодировании углов поворота аминокислот:
Казалось бы, что Виктория пишет всё правильно:

На уровне логики всё верно, если не делать тонких отличий 310-спирали от альфа-спирали.

А здесь и на геометрическом уровне всё правильно! Карты Рамачандрана... Это карты углов поворота следующего аминокислотного остатка относительно предыдущего. Вокруг этой самой оси:
Казалось бы, что Виктория всё понимает, правильно докладывает, показывает правильные картинки, называет правильные карты Рамачандрана...

И тут, вдруг, бац! И после правильных картинок в верхнем ряду идёт ...
нижний ряд, где показана другая ось, т.е. не ось вращения tRNA, не ось вращение аминокислоты относительно соседней, а ось белковой спирали. А углы ... антифолдировались! Как же я не заметил раньше? Просто не мог даже представить ошибку такого уровня...

Это всё равно, что рулить в другой плоскости
Смотрим ещё раз внимательно:

Здесь Виктория показывает вращение вокруг оси симметрии tRNA, т.е. правильно.

А о чём идёт речь на этом слайде? Что это за мистический фолдинг, который превращает угол 0 градусов в угол альфа-спирали, угол 120 градусов в угол бета-спирали, а угол 240 градусов в угол пи-спирали?
А здесь Виктория поменяла ось симметрии tRNA на ось симметрии белковой спирали. При этом смена системы координатных осей автоматически приводит к изменению углов, т.е. это преобразования координат при переходе в другую систему отсчета, а вовсе не фолдинг!
Книга Пикотехнология белков :

Кушелев: Осталось исправить досадную ошибку. Не фолдинг, а преобразование углов при переходе в другую систему отсчёта.
После исправления этой ошибки углы по алгоритму Виктории совпадут с углами альфа-310-спирали с точностью до +-10 градусов, т.к. угол 310-спирали отличается от угла альфа-спирали примерно на 20 градусов. С углом пи-спирали с точностью до нескольких градусов, т.к. угол пи-спирали тоже отличается от одиночного пи-угла. С углом бета-спирали.
-А как же фолдинг?
-"Скрипач не нужен"
2017.12.18 01:01:43
Skype, 2017-12-18:
[0:57:39] tiger2007: Кушелев , есть два вопроса .
[0:58:04] tiger2007: 1 откуда известно что программа работает верно .
[0:58:15] tiger2007: 2 для чего это нужно ?
[1:03:21] Кушелев Александр Юрьевич: Программа проверена тремя независимыми способами:
1. Обнаружена сильная корреляция с данными РСА (читайте книгу "Пикотехнология белков") https://picotechnology-of-proteines.nethouse.ru/page/1197850
2. Замкнулись модели фрагментов 6 разных белков через дисульфидные мостики. Вероятность случайного замыкания каждого примерно 1/30 миллиардов. А всех шести 10^-63
3. Обнаружены сверхдлинные фундаментальные и программные спирали.
Что касается "для чего это нужно?", то что Вы имеете в виду под словом "это"? Если Вы имеете в виду зачем определять структуры, то это нужно для разных целей. Ежедневно кто-то оплачивает примерно 60 структур по 10 000 евро (в среднем).






