*
Кольцегранные модели в физике и химии, молекулярной микробиологи и биохимии
3D генетический код Пикотехнология белков
*
*G
Пространственная структура белка закодирована в его нуклеотидной последовательности
Высоко периодичные структуры белков
96.6 кб, 22 августа в 19:30
Популярные теги Все теги
*
*
Уважаемые коллеги!
СОЗДАНИЕ РАСШИРЕННОЙ ВЕРСИИ ПИКОТЕХНОЛОГИИ
Только 3% белков поддаются кристаллизации, т.е. могут быть исследованы методом РСА (рентгено-структурного анализа). Метод РСА трудоёмок, затратен по времени и стоимости.
Метод Пикотех, основанный на открытии Композиционного генетического кода, бытро выдаёт точные, детальные и масштабируемые пространственные изображения белковых молекул.
Силами нескольких программистов создан Online service PROTEIN PICOTECHNOLOGY
Стуктуры 2D Пикотех выполняются в автоматическом режиме.
Структуры 3D Пикотех в зависимости от состава молекул выполняются либо в автоматическом (по геометрическому шаблону) либо в ручном режиме (с учётом физико-химических взаимодействий, а также свойств сустава Pro, являющихся ноу-хау Лаборатории Наномир).
Фактически запрограммирована табличная функция "код-структура". Пространственная структура белка на уровне геометрического алгоритма тоже получается в первом приближении.
Создвание программного обеспечения для построения структур 3D Пикотех - более сложная задача, требующая участия коллектива программистов.
Учитывая, что экономический эффект от создания полноценной системы "Пикотехнология 3D" трудно переоценить, хочу предложить Вам рассмотреть возможность решения этой грандиозной проблемы.
ЭКСПЕРИМЕНТАЛЬНОЕ ОПРЕДЕЛЕНИЕ СТРУКТУР ПРОГРАММЫНЫХ СПИРАЛЕЙ МЕТОДОМ РСА
В лаборатории Наномир создана новая технология определения структуры белковых молекул, которая работает примерной в миллиард раз быстрее РСА. Наша задача в кратчайшие сроки проверить новую технологию, т.к. от её внедрения человечество может получить фантастический эффект. Просим сообщить о возмоности экспериментально выяснить структуры белков, приведённых ниже, методом рентгено-структурного анализа.
Предлагаем проверить Пикотехнолгию белков на простых структурах типа поливалина:
ETC64880: https://www.ncbi.nlm.nih.gov/nuccore/564568708
OXS06857: https://www.ncbi.nlm.nih.gov/nuccore/1226034760
KOF67455: https://www.ncbi.nlm.nih.gov/nuccore/918287724
OUM69688: https://www.ncbi.nlm.nih.gov/nuccore/1198313040
PIS11988: https://www.ncbi.nlm.nih.gov/nuccore/1277217132
KOF75295: https://www.ncbi.nlm.nih.gov/nuccore/918302080
OLP77457: https://www.ncbi.nlm.nih.gov/nuccore/1129165616
KYM86846: https://www.ncbi.nlm.nih.gov/nuccore/1009361734?&fmt_mask=65536
OTF86159: https://www.ncbi.nlm.nih.gov/nuccore/CM007906.1?report=fasta&sat=37&satkey=307832265&itemID=1362
KKF17324: https://www.ncbi.nlm.nih.gov/nuccore/808866584?&fmt_mask=65536
XP_002382247: https://www.ncbi.nlm.nih.gov/nuccore/238502026
OLP98873: https://www.ncbi.nlm.nih.gov/nuccore/1129194414
CDW76368: https://www.ncbi.nlm.nih.gov/nuccore/678335661
AHB99005: https://www.ncbi.nlm.nih.gov/nuccore/564747871?&fmt_mask=65536
OAQ27448: https://www.ncbi.nlm.nih.gov/nuccore/1032646064
OUM69730: https://www.ncbi.nlm.nih.gov/nuccore/1198313040
OGL53990: https://www.ncbi.nlm.nih.gov/nuccore/1084158629
EIE92391: https://www.ncbi.nlm.nih.gov/nuccore/76151980
XP_001590524: https://www.ncbi.nlm.nih.gov/nuccore/156049114
OTG33229: https://www.ncbi.nlm.nih.gov/nuccore/CM007891.1?from=7600734&to=7601591&sat=37&sat_key=307832447
OLQ06943: https://www.ncbi.nlm.nih.gov/nuccore/1129204435
XP_001895277: https://www.ncbi.nlm.nih.gov/nuccore/170580473
XP_001584576: https://www.ncbi.nlm.nih.gov/nuccore/156030497
KOF84884: https://www.ncbi.nlm.nih.gov/nuccore/918315370
С уважением,
Александр Кушелев,
Руководитель Лаборатории Наномир.
+7 (903) 2003424
kushelev20120@yandex.ru
Кушелев на CASP - неудобный конкурс, неудобный конкурсант
http://subscribe.ru/archive/science.news.nanoworldnews/201402/16194748.html
Организаторы CASP 2014 рассекретили списки участников!
Их оказалось в 10 раз меньше заявленного количества...
 |
Пример белка из CASP8. Целевая структура показана цветом в ленточной модели. Показано наложение 354 предсказанных структур (серый цвет, визуализация остова). CASP (англ.Critical Assessment of protein Structure Prediction, критическая оценка предсказания белковых структур) — масштабный эксперимент попредсказанию белковых структур. Проходит с 1994 года с периодичностью каждые два года.[1] CASP объективно тестирует методы предсказания белковых структур и предоставляет независимую оценку структурного моделирования. Основная цель CASP — помощь в улучшении методов определения трехмерной структуры белков из их аминокислотных последовательностей. Более 100 исследовательских групп принимают участие в проекте на постоянной основе. CASP считается всемирным соревнованием в науке структурного моделирования. Википедия |
CASP 2012
http://nanoworld88.narod.ru/data/279.htm
diprospan: Почему один и тот же пост в среду 11 янв 2012 в 12:02 выглядит в оригинале eng и после перевода rus по разному ?
Kushelev: Это ещё мелочи. Почему утром в теме было 4 страницы, а вечером осталась одна? Похоже, что в конкурсе CASP нас будут ждать если не тройные стандарты, то двойные стандарты точно ...
Напомню, что в конкурсе CASP3 перед розыгрышем призов из списка участников неожиданно исчезло 5 лабораторий, в т.ч. и лаборатория Наномир: http://nanoworld88.narod.ru/forum/casp/cafasp.htm
Руководитель конкурса сообщил, что данные, накопленные на сервере CAFASP за полтора месяца, были удалены "по техническим причинам".
Вероятно, по тем же причинам в начале февраля исчезли все мои сообщения на форуме FORCASP в теме, которую я открыл 11 декабря 2011 года. Но, рукописи не горят, поэтому желающие могут прочесть копию уничтоженной темы: http://nanoworld88.narod.ru/forum/casp/index.htm
Подробнее см. в 297-ом выпуске рассылки "Новости лаборатории Наномир"
Виктория Соколик вынуждена портить модель, подстраивая реальность под устаревшие официальные взгляды...
О пригодности CASP для пикотехнологов можно прочесть в 284-ом выпуске рассылки "Новости лаборатории Наномир"
Пикотехнологические модели на конкурс CASP10 можно посмотреть в 310-ом выпуске рассылки "Новости лаборатории Наномир"
Попытки связи с организаторами конкурса CASP похожи на общение с радиоточкой...
Кстати, никаких данных, по которым можно было бы понять, что за группа Victoria принимает участие в конкурсе CASP, я не обнаружил. Это значит, что на конкурсе CASP скрыты данные, указанные при регистрации группы, например, сайт группы.
Научное открытие на конкурсе CASP прошло незамеченным...
Ни организаторы CASP, ни его участники, ни потенциальные заказчики белковых структур не заметили научное открытие, сделанное мною на конкурсе CASP в 2012 году. Ни у кого даже вопросов не возникло по структуре интерлейкина-34...
Пикотехнологическая модель интерлейкина-34
http://subscribe.ru/archive/science.news.nanoworldnews/201402/16194748.html
Кушелев: Глазам своим не верю! Неужто на сайте можно найти данные из лаборатории Наномир?!
Наша группа выступает под номером 469, но на самом деле в конкурсе принимают участие 44 участника ![]()
Виктория Соколик: Я Вам больше скажу, если Вы кликните на оранжевый график например R0001, то у Вас на экране появится окошко с его большим изображением и активными окнами в заголовке: http://predictioncenter.org/casprol/gdtplot.cgi?target=R0001&group=5&models=first Смело нажимайте Tables и получите данные по первой задаче для всех участников CASP, а заодно и искомый список участников. В других опциях заголовка можно ознакомиться с оценочными данными и критериями для представленных моделей всех участников. Удачи в анализе материала. Надеюсь, что данная информация положит конец Вашим эскападам в адрес организаторов этого конкурса. Все результаты на виду, нужно было только разобраться вместо того, чтобы сыпать беспочвенными обвинениями и подозрениями.
Кушелев: Я с самого начала считал организаторов порядочными людьми, пока они не исключили из списка участников 5 лабораторий, включая лабораторию Наномир
А в 2012-ом году списки участников были засекречены вообще. И всех, кто интересовался этими списками на форуме ForCASP забанили, а дискуссию, в которой принимало участие более 10 человек из разных стран, уничтожили. Копию этой дискуссии я скоро снова выложу на сервер. Начало дискуссии на форуме ForCASP опубликовано в 300-ом выпуске рассылки "Новости лаборатории Наномир"
Но времена меняются, и теперь списки участников CASP 2012 года стали доступны. Это радует. Давайте посмотрим данные группы Victoria.
В конкурсе CASP3 лаборатория Наномир числилась среди участников на последнем, 33-ем месте. В конкурсе CASP10 (2012г.) наша группа Victoria числится тоже на последнем, 44-ом месте. Как видим в конкурсе реально участвуют 44 группы.
Кушелев: В конкурсе CASP по прежнему в качестве входных данных предлагается аминокислотная последовательность, которая не содержит информации о вторичной структуре белка...
Пространственную модель и PDB-файл конкурсного белка на конкурс CASP не принимают, т.к. официально считается, что расстояния между атомами не могут быть меньше некоторой величины, которая не соответствует реальности.
2012.02.15 13:00:19
Уважаемая Виктория!
А что нам пишут организаторы CASP?
Dear member of CASP community,
Silvia Crivelli (LBNL, UC Davis, and a member of this community since 1998) is organizing a collaborative group to participate in CASP10 and possibly in CASP ROLL if time permits. This collaborative experiment will let different groups or individuals work on different components of the protein structure prediction pipeline (like alignment, loop modeling, scoring, etc) thus making it possible to leverage expertise at a large scale. The initial goal is to focus on ab initio targets. Certain groups may want to participate in the collaborative effort for all the ab initio targets while others may want to do just a few of them.
If you would like to learn more about this effort please visit wefold.wordpress.com or contact Silvia directly at SNCrivelli@lbl.gov.
Victoria пишет:
Они пишут, что
Silvia Crivelli (LBNL, UC Davis, член CASP с 1998) организует совместную группу, чтобы участвовать в CASP10 и возможно в CASP ROLL, если успеет. Этот совместный эксперимент позволит различным группам или индивидуумам работать над различными компонентами предсказания структуры белка (такими как выравнивание, моделирование петли, выигрыш, и т.д.) таким образом позволяя усилить экспертизу в большем масштабе. Начальная цель состоит в том, чтобы сосредоточиться на задачах. Определенные группы могут участвовать в совместном решении всех задач с самого начала, в то время как другие могут сделать только несколько из них.
Если Вы хотели бы узнать больше об этом намерении, пожалуйста, посетите wefold.wordpress.com или свяжитесь с Сильвией непосредственно в SNCrivelli@lbl.gov.
Дама стратегически мыслит, сколачивая большую группу с разделением труда для участия в конкурсе CASP ROLL .
Victoria пишет:
Дама стратегически мыслит, сколачивая большую группу с разделением труда для участия в конкурсе CASP ROLL .
Кушелев: Может быть напишем ей, расскажем о пикотехнологии? Нам любые зацепки могут помочь выйти на рынок белковых структур. Если предложит объединить усилия, то почему бы и нет? Главное условие, чтобы наше участие было задокументировано. Всё остальное - мелочи
2012.02.15 20:38:00
Пикотехнология белков, ДНК, РНК
Victoria пишет:
Я не представляю, что мы сможем сделать для Silvia Crivelli и её группы поскольку предполагается, что она разбивает каждую задачу на подзадачи и раздаёт участникам для выполнения громоздким трудо- и время-затратным методом моделирования по гомологии, в котором надо в PDB банке спецпрограммами отыскать подобные заданным куски аминокислотных последовательностей с известной структурой и рассчитывать/надеяться на то, что они будут иметь искомую структуру одной из задач CASP.
Мы же с Вами моделируем совсем по другому, буквально сразу получая готовый PDB файл с координатами атомов белка. Ну предложим мы группе Silvia Crivelli уже решенные нами модели, которые загружены на сайте конкурса CASP от имени нашей группы, и ЧТО?
Если бы они в рамках конформационного анализа хоть молекулярную динамику запускали, то можно было бы проверить на наших уже готовых файлах PDB, не сбивается ли пептид в кучу в гидрофобном или гидрофильном микроокружении, может он имеет возможность как-нибудь ещё удачнее сфолдировать свою конформацию из синтезированного структурного шаблона, который мы собственно и моделируем.
Но судя по тексту Silvia Crivelli о таких возможностях в её группе речь не идёт.
А.Ю., я думаю не нужно суетиться и во что бы то не стало стремиться нас засветить. Закончиться конкурс, посмотрим, как наши таланты оценят профессионалы и организуем собственный сервер предсказания/моделирования структур белка. А время и качество наших моделей помогут нас отобрать в естественном отборе лучших из уже существующих как минимум 25 серверов .
Кушелев: Какое совпадение! Я тоже так думал в 1998-ом году, особенно, когда организатор конкурса CASP3 прислал мне лично 43 dne-файла всех конкурсных белков. Но когда все данные, которые ставились на сервер в течение полутора месяцев неожиданно исчезли (и так у 5 разных групп из 33), я понял, что "не всё так просто". Поэтому практически не сомневаюсь, что нас с Вами ждёт та же участь и в этом году, если мы не сможем объединить усилия, например, с Silvia Crivelli.
Мы ей однозначно нужны, т.к. можем дать структуры всех белков даже точнее, чем это может РСА и др. экспериментальные методы. Когда она это увидит, то вряд ли откажется от сотрудничества. А её выкинуть с конкурса вряд ли получится. Поэтому у нас есть очень высокие шансы победить вместе с Silvia Crivelli и практически нулевые шансы дойти до финиша без сотрудничества. Нас выкинут, и никто об этом не узнает. Ведь списка участников нет и скорее всего не будет. Поэтому я бы связался с Сильвией и предложил ей выгодное сотрудничество. У нас будет беспроигрышный вариант в том случае, если Сильвия сможет задокументировать наше участие в CASP10. Речь не идёт о призовых местах, хотя и это исключать нельзя. Но пока мы там никто. Нас просто нет по документам. То, что Вам показывают, никто не видит. И если нас выкинут с конкурса, никто об этом не узнает.
В 1998-ом году результаты лаборатории Наномир видели все участники и гости конкурса на протяжении полутора месяцев. И то наше исчезновение из списка участников никто не заметил. А в этом году нас вообще нет в списке. Наши данные, конечно получат и проанализируют, только потенциальные клиенты об этом ничего не узнают. В таком случае у нас нет смысла участвовать в конкурсе вообще. А Сильвия нам может помочь стать полноправными участниками CASP10.
2012.02.15 21:20:22
Victoria пишет:
Ну, во-первых никто не видит не только группу Victoria, но и группу Silvia Crivelli, а также все остальные. Открыт только список автоматических серверов. А во-вторых, почему Вы решили, что вместе с ней надёжнее? Вы нашли её группу в списке участников предыдущих конкурсов CASP? Если Вы уверены, что так надежнее, то, как говориться, Вам и карты в руки: лично записывайтесь в группу Silvia Crivelli и отправляйте ей готовые файлы PDB для R0001, R0005, R0006 и т.д. Я ни за эту тётю, ни за другого дядю работать не хочу и не буду.
Уважаемая Виктория! Я Вас прекрасно понимаю. Я же сам так же думал. Но давайте рассуждать. Письмо от Сильвии мы с Вами получили. Похоже, что то же письмо получили и остальные участники CASP10. Верно? Значит о существовании Сильвии знаю все участники CASP10. Мы с Вами можем послать письма всем участникам CASP10? Не можем. Значит Сильвия сумела договориться с организаторами CASP10. А это значит, что с её помощью о нас тоже узнают все участники конкурса.
Конечно, можно пойти и другим путём. Попытаться через организаторов CASP10 разослать аналогичные письма с предложением о сотрудничестве со всеми участниками CASP10. Ну, а если не получится, то остаётся вариант сотрудничества с Сильвией. Иначе у нас шансов (нет, не выиграть конкурс!) просто остаться участниками "ноль целых, ноль десятых". Никто из участников CASP10 просто не узнает, что мы участвовали в этом конкурсе.
Теперь о надёжности. Я не утверждал, что вместе с Сильвией надёжнее. Я писал, что если она сможет задокументировать наше участие в CASP, а она похоже сможет, то наши шансы возрастут немерено. Я имею в виду шансы на то, что нас не выкинут из участников конкурса. А в случае победы группы Сильвии победим и мы. Вы же понимаете, что клиенты объединённой группы на самом деле наши клиенты Они придут к Сильвии, а без нас она не сможет им дать структуры белков...
Теперь по поводу "Я ни за эту тётю, ни за другого дядю работать не хочу и не буду".
Если будет задокументировано наше участие и то, что мы делаем, то Вам не придётся ни за кого работать. У Вас не смогут отнять то, что Вам принадлежит. Все делают свою работу, а в результате в группу придут клиенты. И без нас этим клиентам структуры белков дать не смогут. Мы же не предлагаем пользоваться нашей программой. Мы всего лишь даём попробовать на вкус результаты работы наших программ, алгоритмов, Ноу-Хау...
Вас же никто не уговаривает рассказывать секреты, которые позволяют Вам определять структуры конкурсных и вообще любых белков
Так что с Сильвией договариваться нужно именно Вам, тем более, что Вы знаете английский и сможете понять, что она напишет и написать, чтобы она поняла.
Я бы в первом же письме показал ей и модель интерлейкина-34 (димера), и модель гексамера инсулина, и спираль коллагена. Если она увидит пикотехнологические модели, то скорее всего заинтересуется. А в этом случае начнётся взаимовыгодное сотрудничество. Даже если с CASP она нам не сможет помочь, то через неё мы можем выйти на клиентов, которым нужны структуры белков.
Мы сейчас работаем не на кого-то, а на собственную перспективу. Достаточно найти клиентов на структуры белка, и нам больше не придётся считать копейки
Кстати, может быть Сильвии удастся уговорить организаторов раздобыть все нуклеотидные последовательности конкурсных белков...
2012.02.15 21:29:11
http://molbiol.ru/forums/index.php?show … amp;st=200

http://img-fotki.yandex.ru/get/5506/126 … b_orig.gif
2012.02.15 23:44:22
Victoria пишет:Kushelev пишет:
Письмо от Сильвии мы с Вами получили. Похоже, что то же письмо получили и остальные участники CASP10. Верно? Значит о существовании Сильвии знаю все участники CASP10. Мы с Вами можем послать письма всем участникам CASP10? Не можем. Значит Сильвия сумела договориться с организаторами CASP10. А это значит, что с её помощью о нас тоже узнают все участники конкурса..
Не верно. Письмо от Сильвии получили только Вы. Я полагаю потому, что Вы зарегистрировались на сайте CASP, но не выступили на CASP ROLL. Мне или кому-либо ещё, кто зарегистрировался и там, и там Сильвия писем не шлёт. Она просто не подозревает, что Вы в группе Victoria, вот и пытается собрать группу из волонтёров-одиночек, которые не вступили в игру.
Kushelev пишет:
Конечно, можно пойти и другим путём. Попытаться через организаторов CASP10 разослать аналогичные письма с предложением о сотрудничестве со всеми участниками CASP10. Ну, а если не получится, то остаётся вариант сотрудничества с Сильвией. Иначе у нас шансов (нет, не выиграть конкурс!) просто остаться участниками "ноль целых, ноль десятых". Никто из участников CASP10 просто не узнает, что мы участвовали в этом конкурсе.
Это против правил. Где взяла Сильвия Ваш электронный адрес ей виднее, может случайно набрела на форум Наномир по ключевому слову CASP ROLL. Мне трудно представить, что на правах завсегдатая конкурса она соревнуется по принципу "все средства хороши". Хотя везде только лишь люди...
Kushelev пишет:
Теперь о надёжности. Я не утверждал, что вместе с Сильвией надёжнее. Я писал, что если она сможет задокументировать наше участие в CASP, а она похоже сможет, то наши шансы возрастут немерено. Я имею в виду шансы на то, что нас не выкинут из участников конкурса. А в случае победы группы Сильвии победим и мы. Вы же понимаете, что клиенты объединённой группы на самом деле наши клиенты Они придут к Сильвии, а без нас она не сможет им дать структуры белков...
По Станиславскому "...НЕ ВЕРЮ"
Kushelev пишет:
Так что с Сильвией договариваться нужно именно Вам, тем более, что Вы знаете английский и сможете понять, что она напишет и написать, чтобы она поняла.
Я бы в первом же письме показал ей и модель интерлейкина-34 (димера), и модель гексамера инсулина, и спираль коллагена. Если она увидит пикотехнологические модели, то скорее всего заинтересуется. А в этом случае начнётся взаимовыгодное сотрудничество. Даже если с CASP она нам не сможет помочь, то через неё мы можем выйти на клиентов, которым нужны структуры белков.
Кстати, может быть Сильвии удастся уговорить организаторов раздобыть все нуклеотидные последовательности конкурсных белков...
А.Ю., постарайтесь на меня не обижаться, но я не буду вступать в переписку с Сильвией ни от своего, ни от Вашего имени. Вы ведь тоже не делаете того, что Вам не интересно.
2012.02.15 23:44:42
Victoria пишет:
Не верно. Письмо от Сильвии получили только Вы. Я полагаю потому, что Вы зарегистрировались на сайте CASP, но не выступили на CASP ROLL. Мне или кому-либо ещё, кто зарегистрировался и там, и там Сильвия писем не шлёт. Она просто не подозревает, что Вы в группе Victoria, вот и пытается собрать группу из волонтёров-одиночек, которые не вступили в игру.
А... Очень интересно. А как она смогла узнать о моём существовании? Почему я не могу узнать о её существовании, о Вашем (через CASP) и о существовании других участников CASP10 ?
2012.02.15 23:51:16
Victoria пишет:
Это против правил. Где взяла Сильвия Ваш электронный адрес ей виднее, может случайно набрела на форум Наномир по ключевому слову CASP ROLL. Мне трудно представить, что на правах завсегдатая конкурса она соревнуется по принципу "все средства хороши". Хотя везде только лишь люди...
Вообще-то я не вижу нарушений правил. Ведь в условиях CASP не сказано, сколько может быть участников в группе и т.д. Так что предложение Сильвии вроде как в рамках правил CASP. Что касается "всех средств", то пока я заметил только предложение по кооперации. Не вижу в этом ничего плохого. Наоборот, если у нас есть наиболее точные координаты атомов, а у Сильвии есть другие возможности, которых нет у нас, то объединение усилий - это шанс на победу по многим параметрам. Ведь CASP - это всего лишь тактика, а выход на клиентов - часть стратегии...
2012.02.15 23:53:04
Victoria пишет:
По Станиславскому "...НЕ ВЕРЮ"
А зачем тут верить, когда без Вас Сильвия просто не сможет дать клиенту pdb-файл ![]()
2012.02.15 23:58:30
Victoria пишет:
А.Ю., постарайтесь на меня не обижаться, но я не буду вступать в переписку с Сильвией ни от своего, ни от Вашего имени. Вы ведь тоже не делаете того, что Вам не интересно.
Кушелев: Уважаемая Виктория! Понятие "обида" для меня не существует. Что касается "неинтересно", то это может означать "непонятно". И мне нетрудно объяснить, почему Вам на самом деле интересно. Сильвия нам может помочь однозначно. Например, оповестив всех участников и гостей конкурса CASP о нашем существовании. При этом Вы ей ничего не должны
Кушелев: Администрация CASP удалила 4 сообщения из 5.
Вот, что осталось:http://predictioncenter.org/forcasp/vie … bf7044752b
При этом ответа так и не последовало. Список участников международного конкурса CASP10 организаторы держат в строгом секрете...
Зачем это нужно? Чтобы можно было незаметно избавиться от любого участника. Мой личный опыт показывает, что перед подведением итогов организаторы конкурса CASP избавляются даже от тех участников, которые есть в официальном списке.
В1998-ом году из списка участников исчезло 5 из 33
А которые не числятся в списке, о тех вообще речи нет. "Нет человека, и нет проблем"
2012.06.11 10:48:57
3212-спираль напоминает бета-спираль, только имеет пятиричную симметрию (как и семеричная спираль, квазисимметрию, естественно).

Оригинал: http://img-fotki.yandex.ru/get/6311/158 … 4_orig.jpg

Оригинал: http://img-fotki.yandex.ru/get/6113/158 … 2_orig.gif
Сбоку видны характерные ~прямые углы, как и в бета-спирали.
2012.06.11 12:27:46
Kushelev пишет:
13323133231

Оригинал: http://img-fotki.yandex.ru/get/6112/158 … 7_orig.jpg
Эта конструкция напоминает складчатый бета-слой...
Анимированную картинку 1.7 Мб на Яндекс-фотки через Скай-линк загрузить не удаётся. Через GPRS тем более...
Оригинал: http://img-fotki.yandex.ru/get/6213/158 … 1_orig.gif
Не, всё-таки с 5-ого раза загрузилась
2012.06.11 13:37:09
Kushelev пишет:
Любопытный повтор: 23323323 ... Интересно посмотреть на него в 3D...

Оригинал: http://img-fotki.yandex.ru/get/6113/158 … 5_orig.jpg

Оригинал: http://img-fotki.yandex.ru/get/6113/158 … 4_orig.gif
Поворот до совмещения с соседним аминокислотным остатком происходит на 45 градусов, значит спираль имеет 8-ричную квазисимметрию.
2012.06.11 21:19:42
Kushelev пишет:
Интересные повторы 131313131313 и 31131131131. Напомню код спирали с семеричной симметрией: 331331331

Оригинал: http://img-fotki.yandex.ru/get/6312/158 … d_orig.jpg
311311311

Оригинал: http://img-fotki.yandex.ru/get/6113/158 … e_orig.gif
А эту конструкцию вы уже видели в рассылке "Новости лаборатории Наномир":http://nanoworld88.narod.ru/data/212.htm

Оригинал: http://img-fotki.yandex.ru/get/6312/158 … 7_orig.gif
313131

Оригинал: http://img-fotki.yandex.ru/get/6113/158 … f_orig.jpg
2012.06.11 22:51:33
http://www.scientific.ru/dforum/common/1336239463
Biolmol () - 06.05.2012 18:09
Re: CASP10 - международный конкурс предсказания белковых структур ...
В ответ на http://www.scientific.ru/dforum/common/1336143121:
Регистрация на CASP10 начнется в последнюю неделю марта 2012 года. Испытания подключение к серверу ("сухого хода" для сервера предикторов) будет проводиться начиная с 16 апреля 2012 года. Первое предсказание цели будет выпущен не ранее чем через 1 мая; последнее предсказание цели будет выпущен не позднее июля 17; прогноз сезон завершится не позднее 31 июля. Уточнение эксперимент закончится не позднее 17 августа. Тезисы описания методов испытания в CASP10 будут собраны в сентябре. В то же время мы откроем регистрацию на встречу. В программе встречи будут доступны в ноябре. Встреча состоится 9-12 декабря, и примерно за месяц до этой группы с наиболее точные прогнозы и интересные методы получать приглашения дать переговорам.
Постарайтесь не попасть в неудобную ситуацию, как один из участников тут
http://sandywest.narod.ru/pikoteh2.html
***
pasha A - 06.05.2012 18:11
Re: CASP10 - международный конкурс предсказания белковых структур ...
› › › в ответ на: Re: CASP10 - международный конкурс предсказания белковых структур ... – Biolmol
Снес пост Кушелева в связи с нарушениями правил форума. Ваш коммент вынес в отдельную ветку в связи с прикольностью указанной Вами ссылки.
Кушелев: А вот и "снесённый" администратором Scientific.ru мой пост:
Molbiol () - 04.05.2012 18:52
CASP10 - международный конкурс предсказания белковых структур ...
: Стартовал CASP10.
: Участники соревнуются в точности определения структуры белка.
:
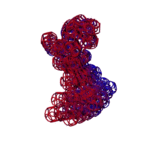
: Типовая структура (interleukin-34):
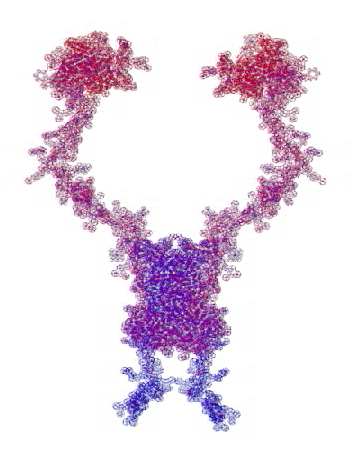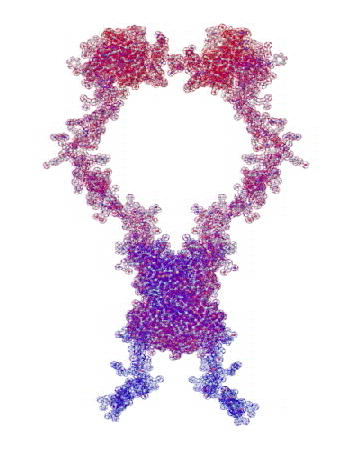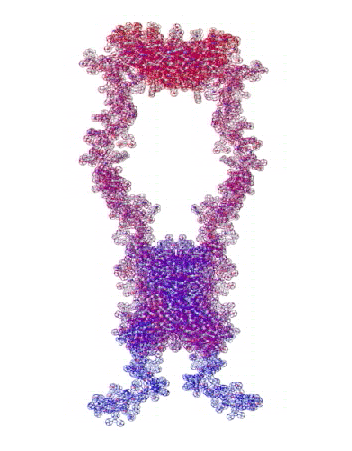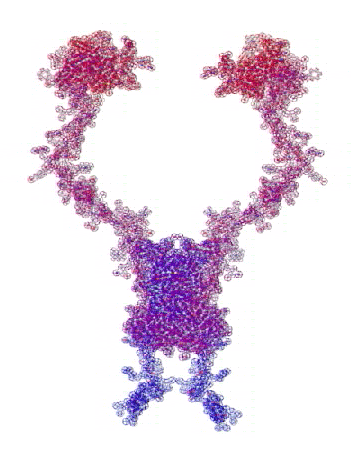
:
: Этот белок состоит из двух почти одинаковых субъединиц.
:
: Гексамер инсулина ПОДРОБНО
:
: Этот белок состоит из 6 одинаковых субъединиц. Анимация показывает динамику. Гексамер регулирует концентрацию глюкозы.
:
: А так выглядит таблица для каждого участника конкурса:
:
: В течение ближайших дней можно принять участие в конкурсе: http://predictioncenter.org/
:
: Обсудить текущие вопросы можно на форуме FORCASP:
:
: http://predictioncenter.org/forcasp/vie … 6834ef1697
:
: В частности, предложен новый стандарт для конкурса: http://nanoworld88.narod.ru/forum/casp/004.htm
"Как Кушелев ставил свои условия организаторам конкурса CASP и CAFASP"
2012.06.11 23:10:20
Вот текст, на который дал "прикольную" ссылку Trilobit, что так понравилось админу Scientific.ru: http://sandywest.narod.ru/pikoteh2.html
Кушелев: Битые ссылки я подправил...
Цитата:
Как Кушелев ставил свои условия организаторам конкурса CASP и CAFASP.
Кушелев:
Я принимал участие в разных конкурсах. Некоторые были честными, некоторые - надуваловка. Это относится не только к России.
Надуть Вас могут и на международном конкурсе. Вот конкретный пример. Международный конкурс CASP и CAFASP. На этом конкурсе нужно определить структуры приблизительно 40 белковых молекул по аминокислотной последовательности.
Я сообщил руководству этого конкурса, что информация о структуре белка содержится не в аминокислотной последовательности самого белка, а в нуклеотидной последовательности кодирующий белок информационной РНК.
Кому интересно узнать структуру белка по коду, а вручную считать утомительно, предлагаем воспользоваться нашей компьютерной программой. Программа написана на стандартном Паскале и позволяет преобразовать генетический код белка в композиционный код его структуры, согласно нашей таблице.
То есть Кушелев попросил, чтобы условия конкурса изменили на более для него удобные.
Кушелев:
После этого руководитель конкурса прислал мне 40 нуклеотидных последовательностей белков, структуру которых выставили на конкурс. Конкурс длился ~2 месяца. Каждые два-три дня приходили новые коды, а решения из лаборатории Наномир выставлялись на сервер CASP. Вот копия базы данных, в которой наша лаборатория числилась под номером 33
Трагическая ошибка Кушелева. Даже по окончанию ссылки видно, что это база данных CAFASP а вовсе не CASP!
Всё было хорошо до начала подведения итогов конкурса. В день подведения итогов из 33 участников неожиданно исчезло 5, в т.ч. лаборатория Наномир. Как будто мы и не участвовали в конкурсе.
А ведь так и есть! Кушелев хотел участвовать в конкурсе с другими условиями, но коварные организаторы не подчинились Кушелеву!
Если прогноз не будет принят CASP, а сервер разработчик не сможет исправить файл, затем этот прогноз будет удалён из CAFASP.
Кушелев:
Когда я поинтересовался, в чём дело, организатор сообщил, что по техническим причинам не может поставить данные, полученные от лаборатории Наномир на сервер.
Заметим, что по условиям конкурса организаторы и не должны были этого делать! Программа-участник должна была САМА отправить результат на сервер организаторов в требуемом формате.
Кушелев:- Но они же там уже были! - возразил я. На это мне уже ничего не ответили... Вот копия этой переписки:
Переписка на английском
Это прямая ложъ.
Кушелеву сообщили, что необходимо ознакомиться с условиями конкурса, где недвусмысленно напечатано:
Если прогноз не будет принят CASP, а сервер разработчик не сможет исправить файл, затем этот прогноз будет удалена из CAFASP.
Организатор конкурса Leszek просто из сил выбивался, чтобы помочь Кушелеву.
Leszek: У вас есть проблема в том, что есть некоторые технические требования к участию.
Мы не можем запустить все методы сами (я хочу найти время, чтобы сделать это).
Вот почему CAFASP будет сосредоточено только на серверах, которые дадут ответ на автоматизированный запрос посланный нами. Мы хотели бы отправить только аминокислотную последовательность, потому что мы считаем, что структура зависит от этого, а не только на основной последовательности ДНК, но мы могли бы попытаться сделать исключение (не обещаю). Но для этого нужно сервер, чтобы отправить нам ответ в автоматическом режиме.
Проверьте объявление CAFASP и оценка правил (http://www.cs.bgu.ac.il/ ~ dfischer/CAFASP2/evalr.html)
Пожалуйста, прочтите инструкции (!!!): http://www.cs.bgu.ac.il/ ~ dfischer/CAFASP2 / announce3.html (прилагается), формат CASP описано в: http://predictioncenter.llnl. gov/casp4/doc / casp4-format.html (прилагается).
CAFASP это особая дисциплина для серверов. Подводя итоги: вы можете настроить сервер для метода? С наилучшими пожеланиями,Leszek
Но Кушелев решил бороться до конца с правилами конкурса и с его организаторами:
Кушелев: -Мы готовы разместить сервер в вашем распоряжении, но ДНК-последовательность является необходимой в качестве вклада. Не будете ли Вы столь любезны дать нам образец входные данные с ДНК-последовательности, и мы подготовим программу в соответствии с ним на сервере. С наилучшими пожеланиями А. Кушелев.
-Я хотел бы использовать только формат PDB.
-Я считаю, что стандартные вторичные структуры это неправильно, так Я и послал вам только PDB.
-Мы не сделали сервер по техническим причинам, поэтому мы можем принять участие в CAFASP только через электронную почту.
Leszek: Но Вы, наверное, хотите, чтобы ваши предсказания были оценены как-то?
Но ваши результаты не будут оценены, если вы не представите CASP формат прогнозов.
Кушелев : Уважаемый сэр, я не нашел CASP Идентификационный номер нашего сервера. Так что я не знаю, если наши результаты были приняты во внимание. Не будете ли вы так добры, сообщите мне, что этим вопросом. С наилучшими пожеланиями А. Кушелев.
Leszek:Я не смог найти любую CASP отформатированные файлы с вашего сервера. С наилучшими пожеланиями.
Кушелев : Уважаемый сэр, мы направили наши результаты в формате CASP но они были отклонены. Таким образом, мы направили наши результаты только вам. Мы поняли вас, что вы включите наши результаты в базу данных под номером 33 (они находятся в базе данных CAFASP). Буду весьма признателен, если вы проясните ситуацию.
Ничего себе, несчастный Leszek оказывается должен переформатировать результаты Кушелева в нужный формат и разместить врукопашную на сервере!!!
Вообще-то это обязанность конкурсантов!
Кушелев: Уважаемый сэр, мы предсказывали 3D структуру объектов в формат PDB. Как я понял, вы лично вложили результаты в базе CAFASP данные под номером 33.
Я высоко ценю вашу волю, чтобы помочь нам.
Это Кушелев так издевается над одним из организаторов конкурса!
Это для нас очень важно знать, как работает наша программа. С наилучшими пожеланиями А. Кушелев
Напомним, оценке подвергалась вторичная структура!!! И наконец контрольный выстрел в голову Leszek.
Leszek: Вы думаете, 3D структуры или вторичная структура?
Кушелев: 3D-структуры только!
Было бы занятно посмотреть, каково было выражение лица Leszek, когда он прочитал это сообщение. И какие слова он произнёс.
Кушелев: На следующий год в лабораторию Наномир снова пришло приглашение участвовать в конкурсе CASP. Я ответил, что мы будем участвовать при условии, что будут опубликованы наши результаты предыдущего конкурса, где эксперты объяснят, куда делись наши данные. После этого организаторы CASP больше не беспокоят лабораторию Наномир.
И правильно.
А то придётся ещё программу за Кушелева доделывать и на первое место продвигать! А уж объяснить Кушелеву в чём его ошибки может и жизни не хватить.
© 2009 "Офигеология без границ"
Конец цитаты.
Кушелев: Переписка с организатором CASP
Подведём итог. Организаторы CASP за 14 лет не обратили внимание на открытие композиционного кода. Все эти годы десятки конкурсантов "бились саблями", когда рядом "лежал пулемёт"
Они и сегодня уничтожают любую информацию о пикотехнологии на форуме ForCASP
Какова же их цель? Зачем десятилетиями биться (и бить десятки исследовательских групп!) головой об стену, когда проблема давно решена?
2012.06.12 01:21:04
А.Ю., зачем сотрясать зря воздух? Принимая участие в конкурсе нужно делать всё самостоятельно: находить нуклеотидные последовательности для задач конкурса (что совсем не сложно), убирать клеш из декодированных структурных шаблонов белков (минимизировать их по энергии системы), загружать pdb. файлы белков на сайте конкурса и др.
Например, Вы с клешем справились? Если да, то Ваши модели я загружу на конкурс. Если нет, то изучайте матчасть .
Кушелев: При чём тут вообще конкурс? Десятки исследовательских групп десятками лет толкут воду в ступе, когда у них под носом развивается пикотехнология. Это значит, что к науке деятельность всех этих "учёных" не имеет отношения.
Что касается подготовки данных на конкурс, то я этим занимаюсь по мере возможности. Сегодня начинаю делать PDB-файлы белков от 691. Что касается клиринга, то попробуем. Хотя по большому счёту это - пустая трата времени.
Оценивать точность пикотехнологических моделей будут (если вообще будут) по ошибочным гипотезам, поэтому призовое место займёт не тот, кто даст более точные к реальности данные, а тот, кто даст данные, максимально близкие к ошибочным, но официальным представлениям. Победа в таком конкурсе - позор для исследователя
2012.06.12 12:31:55
Пикотехнологические модели конкурсных белков с 691 по 708 (по два вида)
(смотреть через зеркало)
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
